+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | X-31 Hemagglutinin Precursor HA0 at pH 4.8 | |||||||||
 マップデータ マップデータ | Sharpened map of the X-31 influenza hemagglutinin precursor HA0 at low pH. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | virus envelope / receptor binding / membrane fusion / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral budding from plasma membrane / virion component / clathrin-dependent endocytosis of virus by host cell / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Influenza A virus (A/Aichi/2/1968(H3N2)) (A型インフルエンザウイルス) / Influenza A virus (A/Aichi/2/1968(H3N2)) (A型インフルエンザウイルス) /  Tequatrovirus T4 (ウイルス) Tequatrovirus T4 (ウイルス) | |||||||||
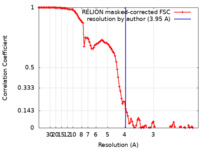
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.95 Å | |||||||||
 データ登録者 データ登録者 | Garcia-Moro E / Rosenthal PB | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2022 ジャーナル: Proc Natl Acad Sci U S A / 年: 2022タイトル: Reversible structural changes in the influenza hemagglutinin precursor at membrane fusion pH. 著者: Eva Garcia-Moro / Jie Zhang / Lesley J Calder / Nick R Brown / Steven J Gamblin / John J Skehel / Peter B Rosenthal /  要旨: The subunits of the influenza hemagglutinin (HA) trimer are synthesized as single-chain precursors (HA0s) that are proteolytically cleaved into the disulfide-linked polypeptides HA1 and HA2. Cleavage ...The subunits of the influenza hemagglutinin (HA) trimer are synthesized as single-chain precursors (HA0s) that are proteolytically cleaved into the disulfide-linked polypeptides HA1 and HA2. Cleavage is required for activation of membrane fusion at low pH, which occurs at the beginning of infection following transfer of cell-surface-bound viruses into endosomes. Activation results in extensive changes in the conformation of cleaved HA. To establish the overall contribution of cleavage to the mechanism of HA-mediated membrane fusion, we used cryogenic electron microscopy (cryo-EM) to directly image HA0 at neutral and low pH. We found extensive pH-induced structural changes, some of which were similar to those described for intermediates in the refolding of cleaved HA at low pH. They involve a partial extension of the long central coiled coil formed by melting of the preexisting secondary structure, threading it between the membrane-distal domains, and subsequent refolding as extended helices. The fusion peptide, covalently linked at its N terminus, adopts an amphipathic helical conformation over part of its length and is repositioned and packed against a complementary surface groove of conserved residues. Furthermore, and in contrast to cleaved HA, the changes in HA0 structure at low pH are reversible on reincubation at neutral pH. We discuss the implications of covalently restricted HA0 refolding for the cleaved HA conformational changes that mediate membrane fusion and for the action of antiviral drug candidates and cross-reactive anti-HA antibodies that can block influenza infectivity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14743.map.gz emd_14743.map.gz | 97.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14743-v30.xml emd-14743-v30.xml emd-14743.xml emd-14743.xml | 22.1 KB 22.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14743_fsc.xml emd_14743_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14743.png emd_14743.png | 74 KB | ||
| マスクデータ |  emd_14743_msk_1.map emd_14743_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-14743.cif.gz emd-14743.cif.gz | 7.1 KB | ||
| その他 |  emd_14743_additional_1.map.gz emd_14743_additional_1.map.gz emd_14743_half_map_1.map.gz emd_14743_half_map_1.map.gz emd_14743_half_map_2.map.gz emd_14743_half_map_2.map.gz | 91.3 MB 95.7 MB 95.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14743 http://ftp.pdbj.org/pub/emdb/structures/EMD-14743 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14743 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14743 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14743_validation.pdf.gz emd_14743_validation.pdf.gz | 779.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14743_full_validation.pdf.gz emd_14743_full_validation.pdf.gz | 778.8 KB | 表示 | |
| XML形式データ |  emd_14743_validation.xml.gz emd_14743_validation.xml.gz | 17.9 KB | 表示 | |
| CIF形式データ |  emd_14743_validation.cif.gz emd_14743_validation.cif.gz | 23.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14743 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14743 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14743 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14743 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14743.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14743.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map of the X-31 influenza hemagglutinin precursor HA0 at low pH. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
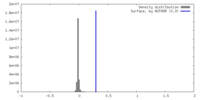
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14743_msk_1.map emd_14743_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
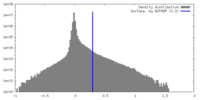
| 密度ヒストグラム |
-追加マップ: Map processed with the deepEMhancer software, used to...
| ファイル | emd_14743_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map processed with the deepEMhancer software, used to aid model building. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
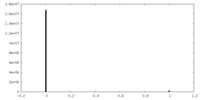
| 密度ヒストグラム |
-ハーフマップ: Half map 2.
| ファイル | emd_14743_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
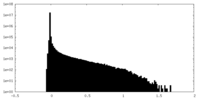
| 密度ヒストグラム |
-ハーフマップ: Half map 1.
| ファイル | emd_14743_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : X-31 Hemagglutinin Precursor (HA0)
| 全体 | 名称: X-31 Hemagglutinin Precursor (HA0) |
|---|---|
| 要素 |
|
-超分子 #1: X-31 Hemagglutinin Precursor (HA0)
| 超分子 | 名称: X-31 Hemagglutinin Precursor (HA0) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: Uncleaved precursor form of the X-31 influenza hemagglutinin with the T4 fibritin foldon attached to the C-terminus. |
|---|---|
| 由来(天然) | 生物種:  Influenza A virus (A/Aichi/2/1968(H3N2)) (A型インフルエンザウイルス) Influenza A virus (A/Aichi/2/1968(H3N2)) (A型インフルエンザウイルス) |
-分子 #1: Hemagglutinin,Fibritin
| 分子 | 名称: Hemagglutinin,Fibritin / タイプ: protein_or_peptide / ID: 1 詳細: T4 fibritin foldon attached to the C-terminal of HA0 as a trimerization domain. C-terminal His8-tag for protein purification.,T4 fibritin foldon attached to the C-terminal of HA0 as a ...詳細: T4 fibritin foldon attached to the C-terminal of HA0 as a trimerization domain. C-terminal His8-tag for protein purification.,T4 fibritin foldon attached to the C-terminal of HA0 as a trimerization domain. C-terminal His8-tag for protein purification.,T4 fibritin foldon attached to the C-terminal of HA0 as a trimerization domain. C-terminal His8-tag for protein purification.,T4 fibritin foldon attached to the C-terminal of HA0 as a trimerization domain. C-terminal His8-tag for protein purification. コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Tequatrovirus T4 (ウイルス) Tequatrovirus T4 (ウイルス) |
| 分子量 | 理論値: 62.027055 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QDLPGNDNST ATLCLGHHAV PNGTLVKTIT DDQIEVTNAT ELVQSSSTGK ICNNPHRILD GIDCTLIDAL LGDPHCDVFQ NETWDLFVE RSKAFSNCYP YDVPDYASLR SLVASSGTLE FITEGFTWTG VTQNGGSNAC KRGPGSGFFS RLNWLTKSGS T YPVLNVTM ...文字列: QDLPGNDNST ATLCLGHHAV PNGTLVKTIT DDQIEVTNAT ELVQSSSTGK ICNNPHRILD GIDCTLIDAL LGDPHCDVFQ NETWDLFVE RSKAFSNCYP YDVPDYASLR SLVASSGTLE FITEGFTWTG VTQNGGSNAC KRGPGSGFFS RLNWLTKSGS T YPVLNVTM PNNDNFDKLY IWGIHHPSTN QEQTSLYVQA SGRVTVSTRR SQQTIIPNIG SRPWVRGLSS RISIYWTIVK PG DVLVINS NGNLIAPRGY FKMRTGKSSI MRSDAPIDTC ISECITPNGS IPNDKPFQNV NKITYGACPK YVKQNTLKLA TGM RNVPEK QTRGLFGAIA GFIENGWEGM IDGWYGFRHQ NSEGTGQAAD LKSTQAAIDQ INGKLNRVIE KTNEKFHQIE KEFS EVEGR IQDLEKYVED TKIDLWSYNA ELLVALENQH TIDLTDSEMN KLFEKTRRQL RENAEEMGNG CFKIYHKCDN ACIES IRNG TYDHDVYRDE ALNNRFQIKG GGRENLYFQG GGGSGYIPEA PRDGQAYVRK DGEWVLLSTF LGHHHHHHHH UniProtKB: Hemagglutinin, Fibritin |
-分子 #5: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 5 / コピー数: 3 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.25 mg/mL |
|---|---|
| 緩衝液 | pH: 4.8 |
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: 4s blot. |
| 詳細 | The sample protein tended to aggregate and bind to the carbon film. 0.1% b-octyl glucoside was added. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-32 / 撮影したグリッド数: 2 / 実像数: 33977 / 平均露光時間: 8.0 sec. / 平均電子線量: 41.15 e/Å2 詳細: 15696 images on session 1, 18281 images on session 2. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 3.3000000000000003 µm 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)