+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Unbound state of a brocolli-pepper aptamer FRET tile | |||||||||
 マップデータ マップデータ | Map sharpened with a b-factor of 160. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RNA / origami / aptamer / fret | |||||||||
| 生物種 | synthetic construct (人工物) | |||||||||
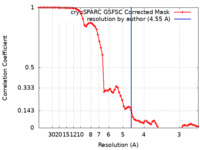
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.55 Å | |||||||||
 データ登録者 データ登録者 | McRae EKS / Vallina NS / Hansen BK / Boussebayle A / Andersen ES | |||||||||
| 資金援助 |  デンマーク, 1件 デンマーク, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Structure determination of Pepper-Broccoli FRET pair by RNA origami scaffolding 著者: McRae EKS / Vallina NS / Hansen BK / Boussebayle A / Andersen ES #1:  ジャーナル: Acta Crystallogr D Struct Biol / 年: 2018 ジャーナル: Acta Crystallogr D Struct Biol / 年: 2018タイトル: Real-space refinement in PHENIX for cryo-EM and crystallography. 著者: Afonine PV / Poon BK / Read RJ / Sobolev OV / Terwilliger TC / Urzhumtsev A / Adams PD #2:  ジャーナル: Acta Crystallogr D Struct Biol / 年: 2018 ジャーナル: Acta Crystallogr D Struct Biol / 年: 2018タイトル: ISOLDE: a physically realistic environment for model building into low-resolution electron-density maps. 著者: Croll TI #3:  ジャーナル: Nat Methods / 年: 2017 ジャーナル: Nat Methods / 年: 2017タイトル: cryoSPARC: algorithms for rapid unsupervised cryo-EM structure determination. 著者: Punjani A / Rubinstein JL / Fleet DJ / Brubaker MA | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14741.map.gz emd_14741.map.gz | 36.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14741-v30.xml emd-14741-v30.xml emd-14741.xml emd-14741.xml | 21.9 KB 21.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14741_fsc.xml emd_14741_fsc.xml | 7.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14741.png emd_14741.png | 28 KB | ||
| マスクデータ |  emd_14741_msk_1.map emd_14741_msk_1.map | 38.4 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-14741.cif.gz emd-14741.cif.gz | 6 KB | ||
| その他 |  emd_14741_additional_1.map.gz emd_14741_additional_1.map.gz emd_14741_half_map_1.map.gz emd_14741_half_map_1.map.gz emd_14741_half_map_2.map.gz emd_14741_half_map_2.map.gz | 19.2 MB 35.7 MB 35.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14741 http://ftp.pdbj.org/pub/emdb/structures/EMD-14741 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14741 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14741 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14741_validation.pdf.gz emd_14741_validation.pdf.gz | 902.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14741_full_validation.pdf.gz emd_14741_full_validation.pdf.gz | 902.2 KB | 表示 | |
| XML形式データ |  emd_14741_validation.xml.gz emd_14741_validation.xml.gz | 14.8 KB | 表示 | |
| CIF形式データ |  emd_14741_validation.cif.gz emd_14741_validation.cif.gz | 19 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14741 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14741 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14741 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14741 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14741.map.gz / 形式: CCP4 / 大きさ: 38.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14741.map.gz / 形式: CCP4 / 大きさ: 38.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
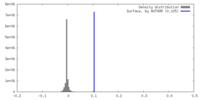
| 注釈 | Map sharpened with a b-factor of 160. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.29 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14741_msk_1.map emd_14741_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
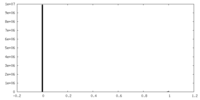
| 密度ヒストグラム |
-追加マップ: Unsharpened map
| ファイル | emd_14741_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
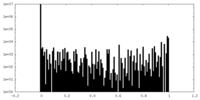
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_14741_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
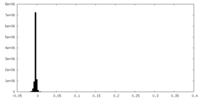
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_14741_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Unbound state of a brocolli-pepper aptamer FRET tile
| 全体 | 名称: Unbound state of a brocolli-pepper aptamer FRET tile |
|---|---|
| 要素 |
|
-超分子 #1: Unbound state of a brocolli-pepper aptamer FRET tile
| 超分子 | 名称: Unbound state of a brocolli-pepper aptamer FRET tile タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 120 KDa |
-分子 #1: brocolli-pepper aptamer
| 分子 | 名称: brocolli-pepper aptamer / タイプ: rna / ID: 1 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 120.700953 KDa |
| 配列 | 文字列: GGAUACGUCU ACGCUCAGUG ACGGACUCUC UUCGGAGAGU CUGACAUCCG AACCAUACAC GGAUGUGCCU CGCCGAACAG UCUACGGCG AGCUUAAGCG CUGGGGACGC CCAACGCAUC ACAAAGACUG AGUGAUGAAC CAGAAGUAUG GACUGGUUGC G UUGGUGGA ...文字列: GGAUACGUCU ACGCUCAGUG ACGGACUCUC UUCGGAGAGU CUGACAUCCG AACCAUACAC GGAUGUGCCU CGCCGAACAG UCUACGGCG AGCUUAAGCG CUGGGGACGC CCAACGCAUC ACAAAGACUG AGUGAUGAAC CAGAAGUAUG GACUGGUUGC G UUGGUGGA GACGGUCGGG UCCAGUUCGC UGUCGAGUAG AGUGUGGGCU CCAUCGACGC CGCUUUAAGG UCCCCAAUCG UG GCGUGUC GGCCUGCUUC GGCAGGCACU GGCGCCGGGA CCUUGAAGAG AUGAGAUUUC GAUCUCAUCU UUGGGUGUCU CUG GUGCUU GAGGGCCCUG UGUUCGCACA GGGCCGCUCA CUGGGUGUGG ACGUAUCC |
-分子 #2: POTASSIUM ION
| 分子 | 名称: POTASSIUM ION / タイプ: ligand / ID: 2 / コピー数: 1 / 式: K |
|---|---|
| 分子量 | 理論値: 39.098 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 40mM HEPES pH 7.5, 5mM MgCl2, 50mM KCl. Filtered through 0.22 um filter. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 288 K / 装置: LEICA EM GP 詳細: 3 uL sample, blotted onto double layer of whatman filter paper for 6 seconds.. |
| 詳細 | Sample was purified by size exclusion chromatography. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 1605 / 平均電子線量: 60.0 e/Å2 詳細: Collected with a calibrated pixel size of 0.647 Angstrom |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)