+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Membrane-bound CHMP2A-CHMP3 filament (410 Angstrom diameter) | ||||||||||||
 マップデータ マップデータ | Membrane-bound CHMP2A-CHMP3 filament (410 Angstrom diameter) | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Cryo Electron Microscopy / Helical Reconstruction / Membrane-bound CHMP2A-CHMP3 filament / Negative-curvature membrane / CYTOSOLIC PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of centriole elongation / regulation of endosome size / membrane invagination / multivesicular body-lysosome fusion / amphisome membrane / suppression of viral release by host / vesicle fusion with vacuole / ESCRT III complex disassembly / late endosome to lysosome transport / ESCRT III complex ...negative regulation of centriole elongation / regulation of endosome size / membrane invagination / multivesicular body-lysosome fusion / amphisome membrane / suppression of viral release by host / vesicle fusion with vacuole / ESCRT III complex disassembly / late endosome to lysosome transport / ESCRT III complex / kinetochore microtubule / endosome transport via multivesicular body sorting pathway / membrane coat / regulation of centrosome duplication / nuclear membrane reassembly / Sealing of the nuclear envelope (NE) by ESCRT-III / multivesicular body sorting pathway / positive regulation of exosomal secretion / midbody abscission / membrane fission / plasma membrane repair / late endosome to vacuole transport / ubiquitin-dependent protein catabolic process via the multivesicular body sorting pathway / multivesicular body assembly / phosphatidylcholine binding / multivesicular body membrane / exit from mitosis / regulation of mitotic spindle assembly / Translation of Replicase and Assembly of the Replication Transcription Complex / regulation of early endosome to late endosome transport / molecular function inhibitor activity / Lysosome Vesicle Biogenesis / mitotic metaphase chromosome alignment / Macroautophagy / nucleus organization / positive regulation of cytokinesis / ubiquitin-specific protease binding / viral budding via host ESCRT complex / autophagosome membrane / viral release from host cell / protein polymerization / autophagosome maturation / Pyroptosis / nuclear pore / phosphatidylinositol-4,5-bisphosphate binding / multivesicular body / Endosomal Sorting Complex Required For Transport (ESCRT) / viral budding from plasma membrane / HCMV Late Events / macroautophagy / establishment of protein localization / Late endosomal microautophagy / Budding and maturation of HIV virion / protein homooligomerization / kinetochore / autophagy / nuclear envelope / late endosome / protein transport / Translation of Replicase and Assembly of the Replication Transcription Complex / cytoplasmic vesicle / midbody / early endosome / protein domain specific binding / lysosomal membrane / apoptotic process / chromatin / extracellular exosome / identical protein binding / membrane / plasma membrane / cytosol 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
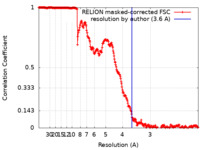
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | ||||||||||||
 データ登録者 データ登録者 | Azad K / Desfosses A / Effantin G / Schoehn G / Weissenhorn W | ||||||||||||
| 資金援助 |  フランス, 3件 フランス, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: Structural basis of CHMP2A-CHMP3 ESCRT-III polymer assembly and membrane cleavage. 著者: Kimi Azad / Delphine Guilligay / Cecile Boscheron / Sourav Maity / Nicola De Franceschi / Guidenn Sulbaran / Gregory Effantin / Haiyan Wang / Jean-Philippe Kleman / Patricia Bassereau / Guy ...著者: Kimi Azad / Delphine Guilligay / Cecile Boscheron / Sourav Maity / Nicola De Franceschi / Guidenn Sulbaran / Gregory Effantin / Haiyan Wang / Jean-Philippe Kleman / Patricia Bassereau / Guy Schoehn / Wouter H Roos / Ambroise Desfosses / Winfried Weissenhorn /   要旨: The endosomal sorting complex required for transport (ESCRT) is a highly conserved protein machinery that drives a divers set of physiological and pathological membrane remodeling processes. However, ...The endosomal sorting complex required for transport (ESCRT) is a highly conserved protein machinery that drives a divers set of physiological and pathological membrane remodeling processes. However, the structural basis of ESCRT-III polymers stabilizing, constricting and cleaving negatively curved membranes is yet unknown. Here we present cryo-EM structures of membrane-coated CHMP2A-CHMP3 filaments from Homo sapiens of two different diameters at 3.3 and 3.6 Å resolution. The structures reveal helical filaments assembled by CHMP2A-CHMP3 heterodimers in the open ESCRT-III conformation, which generates a partially positive charged membrane interaction surface, positions short N-terminal motifs for membrane interaction and the C-terminal VPS4 target sequence toward the tube interior. Inter-filament interactions are electrostatic, which may facilitate filament sliding upon VPS4-mediated polymer remodeling. Fluorescence microscopy as well as high-speed atomic force microscopy imaging corroborate that VPS4 can constrict and cleave CHMP2A-CHMP3 membrane tubes. We therefore conclude that CHMP2A-CHMP3-VPS4 act as a minimal membrane fission machinery. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14631.map.gz emd_14631.map.gz | 91 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14631-v30.xml emd-14631-v30.xml emd-14631.xml emd-14631.xml | 18.7 KB 18.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14631_fsc.xml emd_14631_fsc.xml | 27 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14631.png emd_14631.png | 163.5 KB | ||
| Filedesc metadata |  emd-14631.cif.gz emd-14631.cif.gz | 6.2 KB | ||
| その他 |  emd_14631_half_map_1.map.gz emd_14631_half_map_1.map.gz emd_14631_half_map_2.map.gz emd_14631_half_map_2.map.gz | 1.4 GB 1.4 GB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14631 http://ftp.pdbj.org/pub/emdb/structures/EMD-14631 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14631 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14631 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7zchMC  7zcgC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14631.map.gz / 形式: CCP4 / 大きさ: 1.7 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14631.map.gz / 形式: CCP4 / 大きさ: 1.7 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Membrane-bound CHMP2A-CHMP3 filament (410 Angstrom diameter) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.052 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Membrane-bound CHMP2A-CHMP3 filament (410 Angstrom diameter) half map...
| ファイル | emd_14631_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Membrane-bound CHMP2A-CHMP3 filament (410 Angstrom diameter) half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
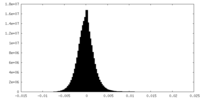
| 密度ヒストグラム |
-ハーフマップ: Membrane-bound CHMP2A-CHMP3 filament (410 Angstrom diameter) half map...
| ファイル | emd_14631_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Membrane-bound CHMP2A-CHMP3 filament (410 Angstrom diameter) half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
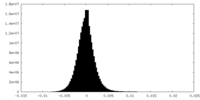
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Membrane-bound CHMP2A-CHMP3 filament (410 Angstrom Diameter) cryo...
| 全体 | 名称: Membrane-bound CHMP2A-CHMP3 filament (410 Angstrom Diameter) cryo-EM map |
|---|---|
| 要素 |
|
-超分子 #1: Membrane-bound CHMP2A-CHMP3 filament (410 Angstrom Diameter) cryo...
| 超分子 | 名称: Membrane-bound CHMP2A-CHMP3 filament (410 Angstrom Diameter) cryo-EM map タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Charged multivesicular body protein 2a
| 分子 | 名称: Charged multivesicular body protein 2a / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 17.305488 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: RKTPEELLRQ NQRALNRAMR ELDRERQKLE TQEKKIIADI KKMAKQGQMD AVRIMAKDLV RTRRYVRKFV LMRANIQAVS LKIQTLKSN NSMAQAMKGV TKAMGTMNRQ LKLPQIQKIM MEFERQAEIM DMKEEMMNDA IDDAMGDE UniProtKB: Charged multivesicular body protein 2a |
-分子 #2: Charged multivesicular body protein 3
| 分子 | 名称: Charged multivesicular body protein 3 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 18.429684 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: PPKELVNEWS LKIRKEMRVV DRQIRDIQRE EEKVKRSVKD AAKKGQKDVC IVLAKEMIRS RKAVSKLYAS KAHMNSVLMG MKNQLAVLR VAGSLQKSTE VMKAMQSLVK IPEIQATMRE LSKEMMKAGI IEEMLEDTFE SMDDQEEMEE EAEMEIDRIL UniProtKB: Charged multivesicular body protein 3 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 実像数: 5028 / 平均露光時間: 5.0 sec. / 平均電子線量: 24.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)