+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The molybdenum storage protein loaded with tungstate | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Metal storage protein / polytungstate / Keggin ion / EM / METAL BINDING PROTEIN | |||||||||
| 機能・相同性 | Molybdenum storage protein subunit alpha/beta / nutrient reservoir activity / molybdenum ion binding / Aspartate/glutamate/uridylate kinase / Acetylglutamate kinase-like superfamily / Amino acid kinase family / cytoplasm / Molybdenum storage protein subunit beta / Molybdenum storage protein subunit alpha 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Azotobacter vinelandii DJ (窒素固定) Azotobacter vinelandii DJ (窒素固定) | |||||||||
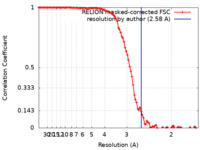
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.58 Å | |||||||||
 データ登録者 データ登録者 | Ermler U / Aziz I / Kaltwasser S / Kayastha K / Vonck J | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Inorg Biochem / 年: 2022 ジャーナル: J Inorg Biochem / 年: 2022タイトル: The molybdenum storage protein forms and deposits distinct polynuclear tungsten oxygen aggregates. 著者: Iram Aziz / Susann Kaltwasser / Kanwal Kayastha / Radhika Khera / Janet Vonck / Ulrich Ermler /  要旨: Some N-fixing bacteria store Mo to maintain the formation of the vital FeMo-cofactor dependent nitrogenase under Mo depleting conditions. The Mo storage protein (MoSto), developed for this purpose, ...Some N-fixing bacteria store Mo to maintain the formation of the vital FeMo-cofactor dependent nitrogenase under Mo depleting conditions. The Mo storage protein (MoSto), developed for this purpose, has the unique capability to compactly deposit molybdate as polyoxometalate (POM) clusters in a (αβ) hexameric cage; the same occurs with the physicochemically related tungstate. To explore the structural diversity of W-based POM clusters, MoSto loaded under different conditions with tungstate and two site-specifically modified MoSto variants were structurally characterized by X-ray crystallography or single-particle cryo-EM. The MoSto cage contains five major locations for POM clusters occupied among others by heptanuclear, Keggin ion and even Dawson-like species also found in bulk solvent under defined conditions. We found both lacunary derivatives of these archetypical POM clusters with missing WO units at positions exposed to bulk solvent and expanded derivatives with additional WO units next to protecting polypeptide segments or other POM clusters. The cryo-EM map, unexpectedly, reveals a POM cluster in the cage center anchored to the wall by a WO linker. Interestingly, distinct POM cluster structures can originate from identical, highly occupied core fragments of three to seven WO units that partly correspond to those found in MoSto loaded with molybdate. These core fragments are firmly bound to the complementary protein template in contrast to the more variable, less occupied residual parts of the visible POM clusters. Due to their higher stability, W-based POM clusters are, on average, larger and more diverse than their Mo-based counterparts. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14522.map.gz emd_14522.map.gz | 64.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14522-v30.xml emd-14522-v30.xml emd-14522.xml emd-14522.xml | 19.7 KB 19.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14522_fsc.xml emd_14522_fsc.xml | 9.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14522.png emd_14522.png | 56.8 KB | ||
| マスクデータ |  emd_14522_msk_1.map emd_14522_msk_1.map | 83.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-14522.cif.gz emd-14522.cif.gz | 6.4 KB | ||
| その他 |  emd_14522_half_map_1.map.gz emd_14522_half_map_1.map.gz emd_14522_half_map_2.map.gz emd_14522_half_map_2.map.gz | 65.3 MB 65.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14522 http://ftp.pdbj.org/pub/emdb/structures/EMD-14522 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14522 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14522 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14522_validation.pdf.gz emd_14522_validation.pdf.gz | 822.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14522_full_validation.pdf.gz emd_14522_full_validation.pdf.gz | 821.9 KB | 表示 | |
| XML形式データ |  emd_14522_validation.xml.gz emd_14522_validation.xml.gz | 16.9 KB | 表示 | |
| CIF形式データ |  emd_14522_validation.cif.gz emd_14522_validation.cif.gz | 22.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14522 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14522 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14522 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14522 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14522.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14522.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.831 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14522_msk_1.map emd_14522_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_14522_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_14522_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : MoSto dodecamer 2 x (AB)3
+超分子 #1: MoSto dodecamer 2 x (AB)3
+分子 #1: Molybdenum storage protein subunit beta
+分子 #2: Molybdenum storage protein subunit alpha
+分子 #3: MOLYBDATE ION
+分子 #4: ADENOSINE-5'-TRIPHOSPHATE
+分子 #5: MAGNESIUM ION
+分子 #6: W11-O35 cluster
+分子 #7: 1,1,3,3,5,7,7,9,11,15,15-undecakis($l^{1}-oxidanyl)-2$l^{4},4$l^{...
+分子 #8: W8-O26 cluster
+分子 #9: W10-O37 cluster
+分子 #10: tungstate cluster
+分子 #11: W3-O10 cluster
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 42.6 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.1 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)