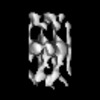+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1449 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The molecular architecture of cadherins in native epidermal desmosomes. | |||||||||
 マップデータ マップデータ | Image used for the isosurface representation, after segmentation and mild filtering | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 34.0 Å | |||||||||
 データ登録者 データ登録者 | Al-Amoudi A / Diez DC / Betts MJ / Frangakis AS | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2007 ジャーナル: Nature / 年: 2007タイトル: The molecular architecture of cadherins in native epidermal desmosomes. 著者: Ashraf Al-Amoudi / Daniel Castaño Díez / Matthew J Betts / Achilleas S Frangakis /  要旨: Desmosomes are cadherin-based adhesive intercellular junctions, which are present in tissues such as heart and skin. Despite considerable efforts, the molecular interfaces that mediate adhesion ...Desmosomes are cadherin-based adhesive intercellular junctions, which are present in tissues such as heart and skin. Despite considerable efforts, the molecular interfaces that mediate adhesion remain obscure. Here we apply cryo-electron tomography of vitreous sections from human epidermis to visualize the three-dimensional molecular architecture of desmosomal cadherins at close-to-native conditions. The three-dimensional reconstructions show a regular array of densities at approximately 70 A intervals along the midline, with a curved shape resembling the X-ray structure of C-cadherin, a representative 'classical' cadherin. Model-independent three-dimensional image processing of extracted sub-tomograms reveals the cadherin organization. After fitting the C-cadherin atomic structure into the averaged sub-tomograms, we see a periodic arrangement of a trans W-like and a cis V-like interaction corresponding to molecules from opposing membranes and the same cell membrane, respectively. The resulting model of cadherin organization explains existing two-dimensional data and yields insights into a possible mechanism of cadherin-based cell adhesion. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1449.map.gz emd_1449.map.gz | 728.2 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1449-v30.xml emd-1449-v30.xml emd-1449.xml emd-1449.xml | 8.4 KB 8.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1449.gif 1449.gif | 11 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1449 http://ftp.pdbj.org/pub/emdb/structures/EMD-1449 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1449 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1449 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1449_validation.pdf.gz emd_1449_validation.pdf.gz | 202.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1449_full_validation.pdf.gz emd_1449_full_validation.pdf.gz | 201.7 KB | 表示 | |
| XML形式データ |  emd_1449_validation.xml.gz emd_1449_validation.xml.gz | 4.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1449 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1449 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1449 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1449 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1449.map.gz / 形式: CCP4 / 大きさ: 1.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1449.map.gz / 形式: CCP4 / 大きさ: 1.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Image used for the isosurface representation, after segmentation and mild filtering | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 6 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
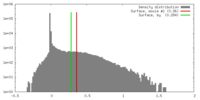
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Cadherin Organisation in Native Desmosomes
| 全体 | 名称: Cadherin Organisation in Native Desmosomes |
|---|---|
| 要素 |
|
-超分子 #1000: Cadherin Organisation in Native Desmosomes
| 超分子 | 名称: Cadherin Organisation in Native Desmosomes / タイプ: sample / ID: 1000 / Number unique components: 1 |
|---|
-超分子 #1: Desmosome
| 超分子 | 名称: Desmosome / タイプ: organelle_or_cellular_component / ID: 1 / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human / 組織: Epidermis Homo sapiens (ヒト) / 別称: Human / 組織: Epidermis |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| グリッド | 詳細: 200 mesh quantifoil grid |
|---|---|
| 凍結 | 凍結剤: NITROGEN / 装置: OTHER / 詳細: Vitrification instrument: Leica EMPact 2 / 手法: High pressure freezing |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F30 |
|---|---|
| 温度 | 平均: 100 K |
| 特殊光学系 | エネルギーフィルター - 名称: Gatan 2002 エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 30.0 eV |
| 撮影 | 平均電子線量: 40 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 49669 / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最小 デフォーカス(公称値): 3.8 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN / Tilt series - Axis1 - Min angle: 64.0 ° / Tilt series - Axis1 - Max angle: 64 ° |
| 実験機器 |  モデル: Tecnai F30 / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Average number of projections used in the 3D reconstructions: 417. |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 34.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 詳細 | Protocol: Rigid body. Semi-automated fitting |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)