登録情報 データベース : EMDB / ID : EMD-14428タイトル Structure of transcription factor UAF in complex with TBP and 35S rRNA promoter DNA Post-processed with deepEMhancer using highres target 複合体 : Upstream Activating Factor (UAF) bound to TBP and promoter DNAタンパク質・ペプチド : Histone H3タンパク質・ペプチド : Histone H4タンパク質・ペプチド : RNA polymerase I-specific transcription initiation factor RRN5タンパク質・ペプチド : RNA polymerase I-specific transcription initiation factor RRN9タンパク質・ペプチド : RNA polymerase I-specific transcription initiation factor RRN10タンパク質・ペプチド : Upstream activation factor subunit UAF30タンパク質・ペプチド : TATA-box-binding proteinDNA : Non-template DNADNA : Template DNA / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
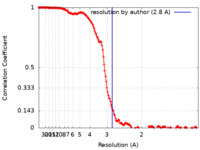
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Saccharomyces cerevisiae (パン酵母)手法 / / 解像度 : 2.8 Å Baudin F / Murciano B 資金援助 Organization Grant number 国 EIPOD fellowship under Marie Sklodowska-Curie Actions COFUND 664726
ジャーナル : Sci Adv / 年 : 2022タイトル : Mechanism of RNA polymerase I selection by transcription factor UAF.著者 : Florence Baudin / Brice Murciano / Herman K H Fung / Simon A Fromm / Simone Mattei / Julia Mahamid / Christoph W Müller / 要旨 : Preribosomal RNA is selectively transcribed by RNA polymerase (Pol) I in eukaryotes. The yeast transcription factor upstream activating factor (UAF) represses Pol II transcription and mediates Pol I ... Preribosomal RNA is selectively transcribed by RNA polymerase (Pol) I in eukaryotes. The yeast transcription factor upstream activating factor (UAF) represses Pol II transcription and mediates Pol I preinitiation complex (PIC) formation at the 35 ribosomal RNA gene. To visualize the molecular intermediates toward PIC formation, we determined the structure of UAF in complex with native promoter DNA and transcription factor TATA-box-binding protein (TBP). We found that UAF recognizes DNA using a hexameric histone-like scaffold with markedly different interactions compared with the nucleosome and the histone-fold-rich transcription factor IID (TFIID). In parallel, UAF positions TBP for Core Factor binding, which leads to Pol I recruitment, while sequestering it from DNA and Pol II/III-specific transcription factors. Our work thus reveals the structural basis of RNA Pol selection by a transcription factor. 履歴 登録 2022年2月23日 - ヘッダ(付随情報) 公開 2022年4月27日 - マップ公開 2022年4月27日 - 更新 2024年7月17日 - 現状 2024年7月17日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 ドイツ, 1件
ドイツ, 1件  引用
引用 ジャーナル: Sci Adv / 年: 2022
ジャーナル: Sci Adv / 年: 2022
 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_14428.map.gz
emd_14428.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-14428-v30.xml
emd-14428-v30.xml emd-14428.xml
emd-14428.xml EMDBヘッダ
EMDBヘッダ emd_14428_fsc.xml
emd_14428_fsc.xml FSCデータファイル
FSCデータファイル emd_14428.png
emd_14428.png emd_14428_msk_1.map
emd_14428_msk_1.map マスクマップ
マスクマップ emd-14428.cif.gz
emd-14428.cif.gz emd_14428_additional_1.map.gz
emd_14428_additional_1.map.gz emd_14428_half_map_1.map.gz
emd_14428_half_map_1.map.gz emd_14428_half_map_2.map.gz
emd_14428_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-14428
http://ftp.pdbj.org/pub/emdb/structures/EMD-14428 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14428
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14428 emd_14428_validation.pdf.gz
emd_14428_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_14428_full_validation.pdf.gz
emd_14428_full_validation.pdf.gz emd_14428_validation.xml.gz
emd_14428_validation.xml.gz emd_14428_validation.cif.gz
emd_14428_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14428
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14428 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14428
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14428
 F&H 検索
F&H 検索 EMPIAR-10964 (タイトル: Structure of transcription factor UAF in complex with TBP and 35S rRNA promoter DNA
EMPIAR-10964 (タイトル: Structure of transcription factor UAF in complex with TBP and 35S rRNA promoter DNA リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_14428.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_14428.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_14428_msk_1.map
emd_14428_msk_1.map 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)