+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single particle structure of keyhole limpet hemocyanin obtained via iDPC scanning transmission electron microscopy | |||||||||
 マップデータ マップデータ | sharpened map from cryosparc local refinement v3.3.1 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | iDPC / STEM / KLH / single particle analysis / OXYGEN TRANSPORT | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報oxygen carrier activity / oxidoreductase activity / extracellular region / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Megathura crenulata (無脊椎動物) Megathura crenulata (無脊椎動物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.51 Å | |||||||||
 データ登録者 データ登録者 | Mann D / Lazic I / Wirix M / de Haas F / Sachse C | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Methods / 年: 2022 ジャーナル: Nat Methods / 年: 2022タイトル: Single-particle cryo-EM structures from iDPC-STEM at near-atomic resolution. 著者: Ivan Lazić / Maarten Wirix / Max Leo Leidl / Felix de Haas / Daniel Mann / Maximilian Beckers / Evgeniya V Pechnikova / Knut Müller-Caspary / Ricardo Egoavil / Eric G T Bosch / Carsten Sachse /   要旨: In electron cryomicroscopy (cryo-EM), molecular images of vitrified biological samples are obtained by conventional transmission microscopy (CTEM) using large underfocuses and subsequently ...In electron cryomicroscopy (cryo-EM), molecular images of vitrified biological samples are obtained by conventional transmission microscopy (CTEM) using large underfocuses and subsequently computationally combined into a high-resolution three-dimensional structure. Here, we apply scanning transmission electron microscopy (STEM) using the integrated differential phase contrast mode also known as iDPC-STEM to two cryo-EM test specimens, keyhole limpet hemocyanin (KLH) and tobacco mosaic virus (TMV). The micrographs show complete contrast transfer to high resolution and enable the cryo-EM structure determination for KLH at 6.5 Å resolution, as well as for TMV at 3.5 Å resolution using single-particle reconstruction methods, which share identical features with maps obtained by CTEM of a previously acquired same-sized TMV data set. These data show that STEM imaging in general, and in particular the iDPC-STEM approach, can be applied to vitrified single-particle specimens to determine near-atomic resolution cryo-EM structures of biological macromolecules. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14407.map.gz emd_14407.map.gz | 59.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14407-v30.xml emd-14407-v30.xml emd-14407.xml emd-14407.xml | 18 KB 18 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
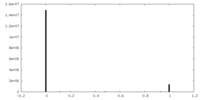
| FSC (解像度算出) |  emd_14407_fsc.xml emd_14407_fsc.xml | 8.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14407.png emd_14407.png | 259.7 KB | ||
| マスクデータ |  emd_14407_msk_1.map emd_14407_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-14407.cif.gz emd-14407.cif.gz | 7 KB | ||
| その他 |  emd_14407_half_map_1.map.gz emd_14407_half_map_1.map.gz emd_14407_half_map_2.map.gz emd_14407_half_map_2.map.gz | 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14407 http://ftp.pdbj.org/pub/emdb/structures/EMD-14407 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14407 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14407 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14407_validation.pdf.gz emd_14407_validation.pdf.gz | 1002.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14407_full_validation.pdf.gz emd_14407_full_validation.pdf.gz | 1002.4 KB | 表示 | |
| XML形式データ |  emd_14407_validation.xml.gz emd_14407_validation.xml.gz | 16.4 KB | 表示 | |
| CIF形式データ |  emd_14407_validation.cif.gz emd_14407_validation.cif.gz | 21.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14407 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14407 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14407 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14407 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14407.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14407.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map from cryosparc local refinement v3.3.1 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.44 Å | ||||||||||||||||||||||||||||||||||||
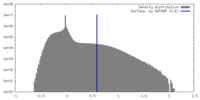
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14407_msk_1.map emd_14407_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_14407_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_14407_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : keyhole limpet hemocyanin
| 全体 | 名称: keyhole limpet hemocyanin |
|---|---|
| 要素 |
|
-超分子 #1: keyhole limpet hemocyanin
| 超分子 | 名称: keyhole limpet hemocyanin / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Megathura crenulata (無脊椎動物) Megathura crenulata (無脊椎動物) |
-分子 #1: keyhole limpet hemocyanin
| 分子 | 名称: keyhole limpet hemocyanin / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Megathura crenulata (無脊椎動物) Megathura crenulata (無脊椎動物) |
| 配列 | 文字列: MLSVRLLIVV LALANAENLV RKSVEHLTQE ETLDLQAALR ELQMDSSSIG FQKIAAAHGA PASCVHKDTS IACCIHGMPT FPHWHRAYVV HMERALQTKR RTSGLPYWDW TEPITQLPSL AADPVYIDSQ GGKAHTNYWY RGNIDFLDKK TNRAVDDRLF EKVKPGQHTH ...文字列: MLSVRLLIVV LALANAENLV RKSVEHLTQE ETLDLQAALR ELQMDSSSIG FQKIAAAHGA PASCVHKDTS IACCIHGMPT FPHWHRAYVV HMERALQTKR RTSGLPYWDW TEPITQLPSL AADPVYIDSQ GGKAHTNYWY RGNIDFLDKK TNRAVDDRLF EKVKPGQHTH LMESVLDALE QDEFCKFEIQ FELAHNAIHY LVGGKHDYSM ANLEYTAYDP IFFLHHSNVD RIFAIWQRLQ ELRNKDPKAM DCAQELLHQK MEPFSWEDND IPLTNEHSTP ADLFDYCELH YDYDTLNLNG MTPEELKTYL DERSSRARAF ASFRLKGFGG SANVFVYVCI PDDNDRNDDH CEKAGDFFVL GGPSEMKWQF YRPYLFDLSD TVHKMGMKLD GHYTVKAELF SVNGTALPDD LLPHPVVVHH PEKGFTDPPV KHHQSANLLV RKNINDLTRE EVLNLREAFH KFQEDRSVDG YQATAEYHGL PARCPRPDAK DRYACCVHGM PIFPHWHRLF VTQVEDALVG RGATIGIPYW DWTEPMTHIP GLAGNKTYVD SHGASHTNPF HSSVIAFEEN APHTKRQIDQ RLFKPATFGH HTDLFNQILY AFEQEDYCDF EVQFEITHNT IHAWTGGSEH FSMSSLHYTA FDPLFYFHHS NVDRLWAVWQ ALQMRRHKPY RAHCAISLEH MHLKPFAFSS PLNNNEKTHA NAMPNKIYDY ENVLHYTYED LTFGGISLEN IEKMIHENQQ EDRIYAGFLL AGIRTSANVD IFIKTTDSVQ HKAGTFAVLG GSKEMKWGFD RVFKFDITHV LKDLDLTADG DFEVTVDITE VDGTKLASSL IPHASVIREH ARVKFDKVPR SRLIRKNVDR LSPEEMNELR KALALLKEDK SAGGFQQLGA FHGEPKWCPS PEASKKFACC VHGMSVFPHW HRLLTVQSEN ALRRHGYDGA LPYWDWTSPL NHLPELADHE KYVDPEDGVE KHNPWFDGHI DTVDKTTTRS VQNKLFEQPE FGHYTSIAKQ VLLALEQDNF CDFEIQYEIA HNYIHALVGG AQPYGMASLR YTAFDPLFYL HHSNTDRIWA IWQALQKYRG KPYNVANCAV TSMREPLQPF GLSANINTDH VTKEHSVPFN VFDYKTNFNY EYDTLEFNGL SISQLNKKLE AIKSQDRFFA GFLLSGFKKS SLVKFNICTD SSNCHPAGEF YLLGDENEMP WAYDRVFKYD ITEKLHDLKL HAEDHFYIDY EVFDLKPASL GKDLFKQPSV IHEPRIGHHE GEVYQAEVTS ANRIRKNIEN LSLGELESLR AAFLEIENDG TYESIAKFHG SPGLCQLNGN PISCCVHGMP TFPHWHRLYV VVVENALLKK GSSVAVPYWD WTKRIEHLPH LISDATYYNS RQHHYETNPF HHGKITHENE ITTRDPKDSL FHSDYFYEQV LYALEQDNFC DFEIQLEILH NALHSLLGGK GKYSMSNLDY AAFDPVFFLH HATTDRIWAI WQDLQRFRKR PYREANCAIQ LMHTPLQPFD KSDNNDEATK THATPHDGFE YQNSFGYAYD NLELNHYSIP QLDHMLQERK RHDRVFAGFL LHNIGTSADG HVFVCLPTGE HTKDCSHEAG MFSILGGQTE MSFVFDRLYK LDITKALKKN GVHLQGDFDL EIEITAVNGS HLDSHVIHSP TILFEAGTDS AHTDDGHTEP VMIRKDITQL DKRQQLSLVK ALESMKADHS SDGFQAIASF HALPPLCPSP AASKRFACCV HGMATFPQWH RLYTVQFQDS LRKHGAVVGL PYWDWTLPRS ELPELLTVST IHDPETGRDI PNPFIGSKIE FEGENVHTKR DINRDRLFQG STKTHHNWFI EQALLALEQT NYCDFEVQFE IMHNGVHTWV GGKEPYGIGH LHYASYDPLF YIHHSQTDRI WAIWQSLQRF RGLSGSEANC AVNLMKTPLK PFSFGAPYNL NDHTHDFSKP EDTFDYQKFG YIYDTLEFAG WSIRGIDHIV RNRQEHSRVF AGFLLEGFGT SATVDFQVCR TAGDCEDAGY FTVLGGEKEM PWAFDRLYKY DITETLDKMN LRHDEIFQIE VTITSYDGTV LDSGLIPTPS IIYDPAHHDI SSHHLSLNKV RHDLSTLSER DIGSLKYALS SLQADTSADG FAAIASFHGL PAKCNDSHNN EVACCIHGMP TFPHWHRLYT LQFEQALRRH GSSVAVPYWD WTKPIHNIPH LFTDKEYYDV WRNKVMPNPF ARGYVPSHDT YTVRDVQEGL FHLTSTGEHS ALLNQALLAL EQHDYCDFAV QFEVMHNTIH YLVGGPQVYS LSSLHYASYD PIFFIHHSFV DKVWAVWQAL QEKRGLPSDR ADCAVSLMTQ NMRPFHYEIN HNQFTKKHAV PNDVFKYELL GYRYDNLEIG GMNLHEIEKE IKDKQHHVRV FAGFLLHGIR TSADVQFQIC KTSEDCHHGG QIFVLGGTKE MAWAYNRLFK YDITHALHDA HITPEDVFHP SEPFFIKVSV TAVNGTVLPA SILHAPTIIY EPGLDHHEDH HSSSMAGHGV RKEINTLTTA EVDNLKDAMR AVMADHGPNG YQAIAAFHGN PPMCPMPDGK NYSCCTHGMA TFPHWHRLYT KQMEDALTAH GARVGLPYWD GTTAFTALPT FVTDEEDNPF HHGHIDYLGV DTTRSPRDKL FNDPERGSES FFYRQVLLAL EQTDFCQFEV QFEITHNAIH SWTGGLTPYG MSTLEYTTYD PLFWLHHANT DRIWAIWQAL QEYRGLPYDH ANCEIQAMKR PLRPFSDPIN HNAFTHSNAK PTDVFEYSRF NFQYDNLRFH GMTIKKLEHE LEKQKEEDRT FAAFLLHGIK KSADVSFDVC NHDGECHFAG TFAILGGEHE MPWSFDRLFR YDITQVLKQM HLEYDSDFTF HMRIIDTSGK QLPSDLIKMP TVEHSPGGKH HEKHHEDHHE DILVRKNIHS LSHHEAEELR DALYKLQNDE SHGGYEHIAG FHGYPNLCPE KGDEKYPCCV HGMSIFPHWH RLHTIQFERA LKKHGSHLGI PYWDWTQTIS SLPTFFADSG NNNPFFKYHI RSINQDTVRD VNEAIFQQTK FGEFSSIFYL ALQALEEDNY CDFEVQYEIL HNEVHALIGG AEKYSMSTLE YSAFDPYFMI HHASLDKIWI IWQELQKRRV KPAHAGSCAG DIMHVPLHPF NYESVNNDDF TRENSLPNAV VDSHRFNYKY DNLNLHGHNI EELEEVLRSL RLKSRVFAGF VLSGIRTTAV VKVYIKSGTD SDDEYAGSFV ILGGAKEMPW AYERLYRFDI TETVHNLNLT DDHVKFRFDL KKYDHTELDA SVLPAPIIVR RPNNAVFDII EIPIGKDVNL PPKVVVKRGT KIMFMSVDEA VTTPMLNLGS YTAMFKCKVP PFSFHAFELG KMYSVESGDY FMTASTTELC NDNNLRIHVH VDDE UniProtKB: Hemocyanin 1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: OTHER / 平均電子線量: 35.0 e/Å2 詳細: STEM imaging was conducted using a Thermo Fisher Scientific Titan Krios G4, operated at 300 kV. The column was equipped with a standard high-brightness field emission gun (XFEG), three- ...詳細: STEM imaging was conducted using a Thermo Fisher Scientific Titan Krios G4, operated at 300 kV. The column was equipped with a standard high-brightness field emission gun (XFEG), three-condenser lens system, C-TWIN objective lens with wide-gap pole piece (11 mm and Cs = 2.7mm), Panther segmented STEM detector, a Ceta camera and Falcon 4 direct electron detection (DED) camera. A combination of different C2 apertures (20 mum and 50 mum) and C2/C3 lens current ratios were used to create different CSA of the beam. Alignment for STEM was done by aligning beam shift, beam tilt pivot points and beam tilt in STEM mode for the different convergence angles. For accurate determination of the COM, a de-scan alignment has additionally been performed. The CSA of the applied beams were measured with high precision using the Au cross-grating and Ceta camera by first recording the radii of the gold diffraction rings for calibration and subsequently measuring the radius of the bright-field (BF) disc. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: OTHER / 最大 デフォーカス(公称値): 0.0 µm / 最小 デフォーカス(公称値): 0.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)