+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1415 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM study of the Pseudomonas bacteriophage phiKZ. | |||||||||
 マップデータ マップデータ | This is a 3D model of the bacteriophage phiKZ based on the previously deposited reconstructions of the capsid (EMD-1392) and the tail (EMD-1393) | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Pseudomonas phage phiKZ (ファージ) Pseudomonas phage phiKZ (ファージ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 28.0 Å | |||||||||
 データ登録者 データ登録者 | Fokine A / Battisti A / Bowman V / Efimov A / Kurochkina L / Chipman P / Mesyanzhinov V / Rossmann M | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2007 ジャーナル: Structure / 年: 2007タイトル: Cryo-EM study of the Pseudomonas bacteriophage phiKZ. 著者: Andrei Fokine / Anthony J Battisti / Valorie D Bowman / Andrei V Efimov / Lidia P Kurochkina / Paul R Chipman / Vadim V Mesyanzhinov / Michael G Rossmann /  要旨: The phiKZ virus is one of the largest known bacteriophages. It infects Pseudomonas aeruginosa, which is frequently pathogenic in humans, and, therefore, has potential for phage therapy. The phiKZ ...The phiKZ virus is one of the largest known bacteriophages. It infects Pseudomonas aeruginosa, which is frequently pathogenic in humans, and, therefore, has potential for phage therapy. The phiKZ virion consists of an approximately 1450 A diameter icosahedral head and an approximately 2000 A long contractile tail. The structure of the phiKZ tail has been determined using cryo-electron microscopy. The phiKZ tail is much longer than that of bacteriophage T4. However, the helical parameters of their contractile sheaths, surrounding their tail tubes, are comparable. Although there is no recognizable sequence similarity between the phiKZ and T4 tail sheath proteins, they are similar in size and shape, suggesting that they evolved from a common ancestor. The phiKZ baseplate is significantly larger than that of T4 and has a flatter shape. Nevertheless, phiKZ, similar to T4, has a cell-puncturing device in the middle of its baseplate. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1415.map.gz emd_1415.map.gz | 12.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1415-v30.xml emd-1415-v30.xml emd-1415.xml emd-1415.xml | 8.8 KB 8.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1415.gif 1415.gif | 35.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1415 http://ftp.pdbj.org/pub/emdb/structures/EMD-1415 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1415 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1415 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1415_validation.pdf.gz emd_1415_validation.pdf.gz | 202.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1415_full_validation.pdf.gz emd_1415_full_validation.pdf.gz | 202 KB | 表示 | |
| XML形式データ |  emd_1415_validation.xml.gz emd_1415_validation.xml.gz | 4.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1415 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1415 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1415 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1415 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1415.map.gz / 形式: CCP4 / 大きさ: 48.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1415.map.gz / 形式: CCP4 / 大きさ: 48.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is a 3D model of the bacteriophage phiKZ based on the previously deposited reconstructions of the capsid (EMD-1392) and the tail (EMD-1393) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 8.4848 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
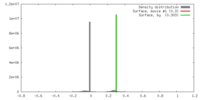
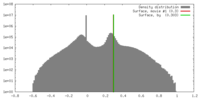
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Bacteriophage phiKZ
| 全体 | 名称: Bacteriophage phiKZ |
|---|---|
| 要素 |
|
-超分子 #1000: Bacteriophage phiKZ
| 超分子 | 名称: Bacteriophage phiKZ / タイプ: sample / ID: 1000 / 集合状態: one / Number unique components: 1 |
|---|
-超分子 #1: Pseudomonas phage phiKZ
| 超分子 | 名称: Pseudomonas phage phiKZ / タイプ: virus / ID: 1 / NCBI-ID: 169683 / 生物種: Pseudomonas phage phiKZ / データベース: NCBI / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 / 詳細: 50 mM tris-Acetate-EDTA buffer (pH 7.5) |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 40 % / 装置: HOMEMADE PLUNGER / 詳細: Vitrification instrument: in house manufactured / 手法: hand blot 3 seconds, plugging during blot |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM300FEG/T |
|---|---|
| アライメント法 | Legacy - 非点収差: live fft |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 112 / 平均電子線量: 20 e/Å2 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 33200 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 3.2 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 33000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 28.0 Å / 解像度の算出法: OTHER 詳細: This is a model of the phiKZ virion based on the previously deposited reconstructions of the phiKZ capsid and the tail part. |
|---|
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)