+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | S.c. Condensin peripheral Ycg1 subcomplex bound to DNA | |||||||||
 マップデータ マップデータ | cryoEM half map 1 of S.c. condensin peripheral Ycg1 complex bound to DNA | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | SMC-motor protein / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of meiotic DNA double-strand break formation / Condensation of Prometaphase Chromosomes / tRNA gene clustering / meiotic chromosome condensation / meiotic chromosome separation / condensin complex / rDNA chromatin condensation / synaptonemal complex assembly / mitotic chromosome condensation / chromosome condensation ...negative regulation of meiotic DNA double-strand break formation / Condensation of Prometaphase Chromosomes / tRNA gene clustering / meiotic chromosome condensation / meiotic chromosome separation / condensin complex / rDNA chromatin condensation / synaptonemal complex assembly / mitotic chromosome condensation / chromosome condensation / mitotic sister chromatid segregation / condensed chromosome / cell division / chromatin binding / nucleus / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
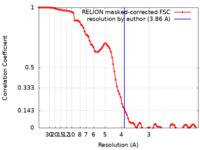
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.86 Å | |||||||||
 データ登録者 データ登録者 | Lecomte L / Hassler M / Haering C / Eustermann S | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2022 ジャーナル: Science / 年: 2022タイトル: A hold-and-feed mechanism drives directional DNA loop extrusion by condensin. 著者: Indra A Shaltiel / Sumanjit Datta / Léa Lecomte / Markus Hassler / Marc Kschonsak / Sol Bravo / Catherine Stober / Jenny Ormanns / Sebastian Eustermann / Christian H Haering /  要旨: Structural maintenance of chromosomes (SMC) protein complexes structure genomes by extruding DNA loops, but the molecular mechanism that underlies their activity has remained unknown. We show that ...Structural maintenance of chromosomes (SMC) protein complexes structure genomes by extruding DNA loops, but the molecular mechanism that underlies their activity has remained unknown. We show that the active condensin complex entraps the bases of a DNA loop transiently in two separate chambers. Single-molecule imaging and cryo-electron microscopy suggest a putative power-stroke movement at the first chamber that feeds DNA into the SMC-kleisin ring upon adenosine triphosphate binding, whereas the second chamber holds on upstream of the same DNA double helix. Unlocking the strict separation of "motor" and "anchor" chambers turns condensin from a one-sided into a bidirectional DNA loop extruder. We conclude that the orientation of two topologically bound DNA segments during the SMC reaction cycle determines the directionality of DNA loop extrusion. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13950.map.gz emd_13950.map.gz | 57 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13950-v30.xml emd-13950-v30.xml emd-13950.xml emd-13950.xml | 29.1 KB 29.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13950_fsc.xml emd_13950_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13950.png emd_13950.png | 63.7 KB | ||
| マスクデータ |  emd_13950_msk_1.map emd_13950_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-13950.cif.gz emd-13950.cif.gz | 8 KB | ||
| その他 |  emd_13950_additional_1.map.gz emd_13950_additional_1.map.gz emd_13950_additional_2.map.gz emd_13950_additional_2.map.gz emd_13950_half_map_1.map.gz emd_13950_half_map_1.map.gz emd_13950_half_map_2.map.gz emd_13950_half_map_2.map.gz | 40.4 MB 49.6 MB 49.6 MB 49.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13950 http://ftp.pdbj.org/pub/emdb/structures/EMD-13950 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13950 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13950 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13950_validation.pdf.gz emd_13950_validation.pdf.gz | 913.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13950_full_validation.pdf.gz emd_13950_full_validation.pdf.gz | 913.4 KB | 表示 | |
| XML形式データ |  emd_13950_validation.xml.gz emd_13950_validation.xml.gz | 15.9 KB | 表示 | |
| CIF形式データ |  emd_13950_validation.cif.gz emd_13950_validation.cif.gz | 20.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13950 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13950 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13950 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13950 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7qfwMC  7qenC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13950.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13950.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryoEM half map 1 of S.c. condensin peripheral Ycg1 complex bound to DNA | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_13950_msk_1.map emd_13950_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
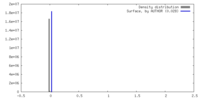
| 密度ヒストグラム |
-追加マップ: cryoEM map of S.c. condensin peripheral Ycg1 complex...
| ファイル | emd_13950_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryoEM map of S.c. condensin peripheral Ycg1 complex bound to DNA local-resolution filtered and postprocessed cryo map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
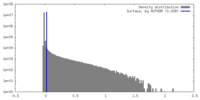
| 密度ヒストグラム |
-追加マップ: unsharpened cryoEM map of S.c. condensin peripheral Ycg1...
| ファイル | emd_13950_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened cryoEM map of S.c. condensin peripheral Ycg1 complex bound to DNA | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
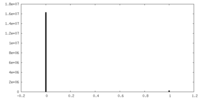
| 密度ヒストグラム |
-ハーフマップ: cryoEM half map 2 of S.c. condensin peripheral...
| ファイル | emd_13950_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryoEM half map 2 of S.c. condensin peripheral Ycg1 complex bound to DNA | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
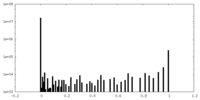
| 密度ヒストグラム |
-ハーフマップ: cryoEM half map 2 of S.c. condensin peripheral...
| ファイル | emd_13950_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryoEM half map 2 of S.c. condensin peripheral Ycg1 complex bound to DNA | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : S.c. Condensin peripheral Ycg1 complex bound to DNA
| 全体 | 名称: S.c. Condensin peripheral Ycg1 complex bound to DNA |
|---|---|
| 要素 |
|
-超分子 #1: S.c. Condensin peripheral Ycg1 complex bound to DNA
| 超分子 | 名称: S.c. Condensin peripheral Ycg1 complex bound to DNA / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 707 KDa |
-分子 #1: Condensin complex subunit 3
| 分子 | 名称: Condensin complex subunit 3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 117.981 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MQDPDGIDIN TKIFNSVAEV FQKAQGSYAG HRKHIAVLKK IQSKAVEQGY EDAFNFWFDK LVTKILPLKK NEIIGDRIVK LVAAFIASL ERELILAKKQ NYKLTNDEEG IFSRFVDQFI RHVLRGVESP DKNVRFRVLQ LLAVIMDNIG EIDESLFNLL I LSLNKRIY ...文字列: MQDPDGIDIN TKIFNSVAEV FQKAQGSYAG HRKHIAVLKK IQSKAVEQGY EDAFNFWFDK LVTKILPLKK NEIIGDRIVK LVAAFIASL ERELILAKKQ NYKLTNDEEG IFSRFVDQFI RHVLRGVESP DKNVRFRVLQ LLAVIMDNIG EIDESLFNLL I LSLNKRIY DREPTVRIQA VFCLTKFQDE EQTEHLTELS DNEENFEATR TLVASIQNDP SAEVRRAAML NLINDNNTRP YI LERARDV NIVNRRLVYS RILKSMGRKC FDDIEPHIFD QLIEWGLEDR ELSVRNACKR LIAHDWLNAL DGDLIELLEK LDV SRSSVC VKAIEALFQS RPDILSKIKF PESIWKDFTV EIAFLFRAIY LYCLDNNITE MLEENFPEAS KLSEHLNHYI LLRY HHNDI SNDSQSHFDY NTLEFIIEQL SIAAERYDYS DEVGRRSMLT VVRNMLALTT LSEPLIKIGI RVMKSLSINE KDFVT MAIE IINDIRDDDI EKQEQEEKIK SKKINRRNET SVDEEDENGT HNDEVNEDEE DDNISSFHSA VENLVQGNGN VSESDI INN LPPEKEASSA TIVLCLTRSS YMLELVNTPL TENILIASLM DTLITPAVRN TAPNIRELGV KNLGLCCLLD VKLAIDN MY ILGMCVSKGN ASLKYIALQV IVDIFSVHGN TVVDGEGKVD SISLHKIFYK VLKNNGLPEC QVIAAEGLCK LFLADVFT D DDLFETLVLS YFSPINSSNE ALVQAFAFCI PVYCFSHPAH QQRMSRTAAD ILLRLCVLWD DLQSSVIPEV DREAMLKPN IIFQQLLFWT DPRNLVNQTG STKKDTVQLT FLIDVLKIYA QIEKKEIKKM IITNINAIFL SSEQDYSTLK ELLEYSDDIA ENDNLDNVS KNALDKLRNN LNSLIEEINE RSETQTKDEN NTANDQYSSI LGNSFNKSSN DTIEHAADIT DGNNTELTKT T VNISAVDN TTEQSNSRKR TRSEAEQIDT SKNLENMSIQ DTSTVAKNVS FVLPDEKSDA MSIDEEDKDS ESFSEVC UniProtKB: Condensin complex subunit 3 |
-分子 #2: Condensin complex subunit 2
| 分子 | 名称: Condensin complex subunit 2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 92.721219 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTTQLRYENN DDDERVEYNL FTNRSTMMAN FEEWIKMATD NKINSRNSWN FALIDYFYDL DVLKDGENNI NFQKASATLD GCIKIYSSR VDSVTTETGK LLSGLAQRKT NGASNGDDSN GGNGEGLGGD SDEANIEIDP LTGMPISNDP DVNNTRRRVY N RVLETTLV ...文字列: MTTQLRYENN DDDERVEYNL FTNRSTMMAN FEEWIKMATD NKINSRNSWN FALIDYFYDL DVLKDGENNI NFQKASATLD GCIKIYSSR VDSVTTETGK LLSGLAQRKT NGASNGDDSN GGNGEGLGGD SDEANIEIDP LTGMPISNDP DVNNTRRRVY N RVLETTLV EFETIKMKEL DQELIIDPLF KKALVDFDEG GAKSLLLNTL NIDNTARVIF DASIKDTQNV GQGKLQRKEE EL IERDSLV DDENEPSQSL ISTRNDSTVN DSVISAPSME DEILSLGMDF IKFDQIAVCE ISGSIEQLRN VVEDINQAKD FIE NVNNRF DNFLTEEELQ AAVPDNAEDD SDGFDMGMQQ ELCYPDENHD NTSHDEQDDD NVNSTTGSIF EKDLMAYFDE NLNR NWRGR EHWKVRNFKK ANLVNKESDL LEETRTTIGD TTDKNTTDDK SMDTKKKHKQ KKVLEIDFFK TDDSFEDKVF ASKGR TKID MPIKNRKNDT HYLLPDDFHF STDRITRLFI KPAQKMSLFS HRKHTRGDVS SGLFEKSTVS ANHSNNDIPT IADEHF WAD NYERKEQEEK EKEQSKEVGD VVGGALDNPF EDDMDGVDFN QAFEGTDDNE EASVKLDLQD DEDHKFPIRE NKVTYSR VS KKVDVRRLKK NVWRSINNLI QEHDSRKNRE QSSNDSETHT EDESTKELKF SDIIQGISKM YSDDTLKDIS TSFCFICL L HLANEHGLQI THTENYNDLI VNYEDLATTQ AASLVGGGHH RPHHGGHHHH HHGGRIFYPY DVPDYAGYPY DVPDYAGSY PYDVPNYAAG H UniProtKB: Condensin complex subunit 2 |
-分子 #3: Synthetic DNA ligand
| 分子 | 名称: Synthetic DNA ligand / タイプ: dna / ID: 3 詳細: 50bp synthetic DNA ligand with the sequence: 5'-GTTGACAGTG TCGCAACCTG CACAGGCAAG CTGCTGAGTC TGGTGTAGAC-3' The DNA ligand was modelled as poly(dA) as the register could not be determined by cryoEM density. コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 15.615376 KDa |
| 配列 | 文字列: (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA) |
-分子 #4: synthetic DNA ligand
| 分子 | 名称: synthetic DNA ligand / タイプ: dna / ID: 4 詳細: 50bp synthetic DNA ligand with the sequence: 5'-GTCTACACCAGACTCAGCAGCTTGCCTGTGCAGGTTGCGACACTGTCAAC-3' The DNA ligand was modelled as poly(dT) as the register could not be determined by cryoEM density. コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 15.164683 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.707 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: GOLD / メッシュ: 200 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 45 sec. / 前処理 - 雰囲気: OTHER | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 実像数: 6544 / 平均露光時間: 8.0 sec. / 平均電子線量: 1.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 2.0 µm / 最小 デフォーカス(補正後): 1.0 µm / 倍率(補正後): 130000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)