+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of TDP43 core peptide amyloid fiber | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  TDP-43 (TAR DNA結合タンパク質43) / TDP-43 (TAR DNA結合タンパク質43) /  Amyloid (アミロイド) / Amyloid (アミロイド) /  Neurodegeneration (神経変性疾患) / Helical / Neurodegeneration (神経変性疾患) / Helical /  Cryo-EM (低温電子顕微鏡法) / Cryo-EM (低温電子顕微鏡法) /  RNA BINDING PROTEIN (RNA結合タンパク質) RNA BINDING PROTEIN (RNA結合タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報nuclear inner membrane organization /  interchromatin granule / perichromatin fibrils / 3'-UTR-mediated mRNA destabilization / 3'-UTR-mediated mRNA stabilization / intracellular non-membrane-bounded organelle / negative regulation by host of viral transcription / pre-mRNA intronic binding / response to endoplasmic reticulum stress / interchromatin granule / perichromatin fibrils / 3'-UTR-mediated mRNA destabilization / 3'-UTR-mediated mRNA stabilization / intracellular non-membrane-bounded organelle / negative regulation by host of viral transcription / pre-mRNA intronic binding / response to endoplasmic reticulum stress /  RNA splicing ...nuclear inner membrane organization / RNA splicing ...nuclear inner membrane organization /  interchromatin granule / perichromatin fibrils / 3'-UTR-mediated mRNA destabilization / 3'-UTR-mediated mRNA stabilization / intracellular non-membrane-bounded organelle / negative regulation by host of viral transcription / pre-mRNA intronic binding / response to endoplasmic reticulum stress / interchromatin granule / perichromatin fibrils / 3'-UTR-mediated mRNA destabilization / 3'-UTR-mediated mRNA stabilization / intracellular non-membrane-bounded organelle / negative regulation by host of viral transcription / pre-mRNA intronic binding / response to endoplasmic reticulum stress /  RNA splicing / negative regulation of protein phosphorylation / molecular condensate scaffold activity / mRNA 3'-UTR binding / RNA splicing / negative regulation of protein phosphorylation / molecular condensate scaffold activity / mRNA 3'-UTR binding /  regulation of protein stability / regulation of protein stability /  regulation of circadian rhythm / positive regulation of insulin secretion / regulation of circadian rhythm / positive regulation of insulin secretion /  転写後修飾 / cytoplasmic stress granule / positive regulation of protein import into nucleus / rhythmic process / 転写後修飾 / cytoplasmic stress granule / positive regulation of protein import into nucleus / rhythmic process /  遺伝子発現の調節 / 遺伝子発現の調節 /  double-stranded DNA binding / regulation of apoptotic process / amyloid fibril formation / double-stranded DNA binding / regulation of apoptotic process / amyloid fibril formation /  regulation of cell cycle / nuclear speck / RNA polymerase II cis-regulatory region sequence-specific DNA binding / negative regulation of gene expression / regulation of cell cycle / nuclear speck / RNA polymerase II cis-regulatory region sequence-specific DNA binding / negative regulation of gene expression /  lipid binding / lipid binding /  ミトコンドリア / ミトコンドリア /  DNA binding / DNA binding /  RNA binding / RNA binding /  核質 / identical protein binding / 核質 / identical protein binding /  細胞核 細胞核類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
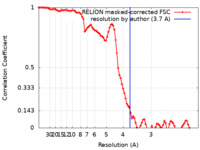
| 手法 | らせん対称体再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.7 Å クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Nazarov S / Lashuel H | |||||||||
| 資金援助 |  スイス, 1件 スイス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Neurosci / 年: 2023 ジャーナル: Nat Neurosci / 年: 2023タイトル: Seeding the aggregation of TDP-43 requires post-fibrillization proteolytic cleavage. 著者: Senthil T Kumar / Sergey Nazarov / Sílvia Porta / Niran Maharjan / Urszula Cendrowska / Malek Kabani / Francesco Finamore / Yan Xu / Virginia M-Y Lee / Hilal A Lashuel /   要旨: Despite the strong evidence linking the transactive response DNA-binding protein 43 (TDP-43) aggregation to the pathogenesis of frontotemporal lobar degeneration with TDP-43, amyotrophic lateral ...Despite the strong evidence linking the transactive response DNA-binding protein 43 (TDP-43) aggregation to the pathogenesis of frontotemporal lobar degeneration with TDP-43, amyotrophic lateral sclerosis and several neurodegenerative diseases, our knowledge of the sequence and structural determinants of its aggregation and neurotoxicity remains incomplete. Herein, we present a new method for producing recombinant full-length TDP-43 filaments that exhibit sequence and morphological features similar to those of brain-derived TDP-43 filaments. We show that TDP-43 filaments contain a β-sheet-rich helical amyloid core that is fully buried by the flanking structured domains of the protein. We demonstrate that the proteolytic cleavage of TDP-43 filaments and exposure of this amyloid core are necessary for propagating TDP-43 pathology and enhancing the seeding of brain-derived TDP-43 aggregates. Only TDP-43 filaments with exposed amyloid core efficiently seeded the aggregation of endogenous TDP-43 in cells. These findings suggest that inhibiting the enzymes mediating cleavage of TDP-43 aggregates represents a viable disease-modifying strategy to slow the progression of amyotrophic lateral sclerosis and other TDP-43 proteinopathies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13795.map.gz emd_13795.map.gz | 4.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13795-v30.xml emd-13795-v30.xml emd-13795.xml emd-13795.xml | 17.5 KB 17.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13795_fsc.xml emd_13795_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13795.png emd_13795.png | 1 MB | ||
| マスクデータ |  emd_13795_msk_1.map emd_13795_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-13795.cif.gz emd-13795.cif.gz | 5.5 KB | ||
| その他 |  emd_13795_additional_1.map.gz emd_13795_additional_1.map.gz emd_13795_half_map_1.map.gz emd_13795_half_map_1.map.gz emd_13795_half_map_2.map.gz emd_13795_half_map_2.map.gz | 6.1 MB 49.7 MB 49.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13795 http://ftp.pdbj.org/pub/emdb/structures/EMD-13795 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13795 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13795 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7q3uMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13795.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13795.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||
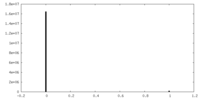
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_13795_msk_1.map emd_13795_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
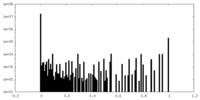
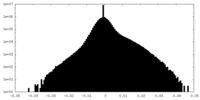
| 密度ヒストグラム |
-追加マップ: Post-processed and symmetrized map of the first (one...
| ファイル | emd_13795_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post-processed and symmetrized map of the first (one protofilament) conformation of TDP-43 CP fibrils | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
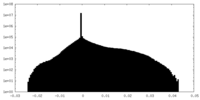
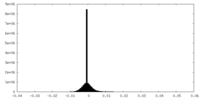
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_13795_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
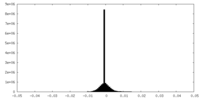
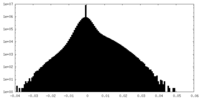
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_13795_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Helical filament from TDP43 core peptide
| 全体 | 名称: Helical filament from TDP43 core peptide |
|---|---|
| 要素 |
|
-超分子 #1: Helical filament from TDP43 core peptide
| 超分子 | 名称: Helical filament from TDP43 core peptide / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: TAR DNA-binding protein 43
| 分子 | 名称: TAR DNA-binding protein 43 / タイプ: protein_or_peptide / ID: 1 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 8.042687 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: NPGGFGNQGG FGNSRGGGAG LGNNQGSNMG GGMNFGAFSI NPAMMAAAQA ALQSSWGMMG MLASQQNQSG PSGNNQNQGN MQ UniProtKB:  TAR DNA結合タンパク質43 TAR DNA結合タンパク質43 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 81000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 3171 / 平均露光時間: 3.99 sec. / 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 103 |
|---|---|
| 得られたモデル |  PDB-7q3u: |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)