+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1341 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Three-dimensional structure of a human connexin26 gap junction channel reveals a plug in the vestibule. | |||||||||
 マップデータ マップデータ | Three Dimensional Structure of a Human Connexin26 Gap Junction Channel | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | Gap junction beta-2 protein (Cx26) / connexin complex 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 電子線結晶学 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 10.0 Å | |||||||||
 データ登録者 データ登録者 | Oshima A / Tani K / Hiroaki Y / Fujiyoshi Y / Sosinsky GE | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2007 ジャーナル: Proc Natl Acad Sci U S A / 年: 2007タイトル: Three-dimensional structure of a human connexin26 gap junction channel reveals a plug in the vestibule. 著者: Atsunori Oshima / Kazutoshi Tani / Yoko Hiroaki / Yoshinori Fujiyoshi / Gina E Sosinsky /  要旨: Connexin molecules form intercellular membrane channels facilitating electronic coupling and the passage of small molecules between adjoining cells. Connexin26 (Cx26) is the second smallest member of ...Connexin molecules form intercellular membrane channels facilitating electronic coupling and the passage of small molecules between adjoining cells. Connexin26 (Cx26) is the second smallest member of the gap junction protein family, and mutations in Cx26 cause certain hereditary human diseases such as skin disorders and hearing loss. Here, we report the electron crystallographic structure of a human Cx26 mutant (M34A). Although crystallization trials used hemichannel preparations, the density map revealed that two hemichannels redocked at their extracellular surfaces into full intercellular channels. These orthorhombic crystals contained two sets of symmetry-related intercellular channels within three lipid bilayers. The 3D map shows a prominent density in the pore of each hemichannel. This density contacts the innermost helices of the surrounding connexin subunits at the bottom of the vestibule. The density map suggests that physical blocking may play an important role that underlies gap junction channel regulation. Our structure allows us to suggest that the two docked hemichannels can be independent and may regulate their activity autonomously with a plug in the vestibule. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1341.map.gz emd_1341.map.gz | 1.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1341-v30.xml emd-1341-v30.xml emd-1341.xml emd-1341.xml | 11.2 KB 11.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1341.gif 1341.gif | 43.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1341 http://ftp.pdbj.org/pub/emdb/structures/EMD-1341 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1341 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1341 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1341_validation.pdf.gz emd_1341_validation.pdf.gz | 206.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1341_full_validation.pdf.gz emd_1341_full_validation.pdf.gz | 205.5 KB | 表示 | |
| XML形式データ |  emd_1341_validation.xml.gz emd_1341_validation.xml.gz | 4.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1341 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1341 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1341 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1341 | HTTPS FTP |
-関連構造データ
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1341.map.gz / 形式: CCP4 / 大きさ: 1.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1341.map.gz / 形式: CCP4 / 大きさ: 1.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Three Dimensional Structure of a Human Connexin26 Gap Junction Channel | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X: 1.98571 Å / Y: 2.00714 Å / Z: 2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
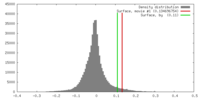
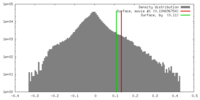
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 18 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Human Connexin26 Gap Junction Channel
| 全体 | 名称: Human Connexin26 Gap Junction Channel |
|---|---|
| 要素 |
|
-超分子 #1000: Human Connexin26 Gap Junction Channel
| 超分子 | 名称: Human Connexin26 Gap Junction Channel / タイプ: sample / ID: 1000 / 集合状態: 12 connexon form a gap junction / Number unique components: 1 |
|---|
-分子 #1: connexin26
| 分子 | 名称: connexin26 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: gap junction beta 2 / コピー数: 12 / 集合状態: dodecamer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: cell membrane Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: cell membrane |
| 組換発現 | 生物種: Sf9 cells / 組換プラスミド: pBlueBac4.5 |
| 配列 | GO: connexin complex / InterPro: Gap junction beta-2 protein (Cx26) |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線結晶学 |
| 試料の集合状態 | 2D array |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 5.8 詳細: 10 mM MES, 100 mM NaCl, 50 mM MgCl2, 5 mM CaCl2, 2 mM DTT, 100 uM carbenoxolone (SIGMA), 0.005% NaN3, 1% glycerol |
| 染色 | タイプ: NEGATIVE / 詳細: Vitrification |
| グリッド | 詳細: molybdenum grid covered with a thin carbon film |
| 凍結 | 凍結剤: NITROGEN / チャンバー内温度: 100 K / 装置: REICHERT-JUNG PLUNGER / 詳細: Vitrification instrument: Reichert plunger 手法: The grid was blotted with filter paper and plunged into liquid nitrogen. |
| 詳細 | crystals grown in dialysis buffer |
| 結晶化 | 詳細: crystals grown in dialysis buffer |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL KYOTO-3000SFF |
|---|---|
| 温度 | 最低: 4.0 K / 最高: 4.0 K / 平均: 4.0 K |
| アライメント法 | Legacy - 非点収差: objective astigmatism was corrected using a quadrupole stigmator at 100,000 times magnification |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 254 / 平均電子線量: 25 e/Å2 / ビット/ピクセル: 12 |
| Tilt angle min | 0 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 59100 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 1.6 mm / 最大 デフォーカス(公称値): 19.4 µm / 最小 デフォーカス(公称値): 4.7 µm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダー: Top entry liquid helium cooled cryo specimen holder. 試料ホルダーモデル: OTHER / Tilt angle max: 45 / Tilt series - Axis1 - Min angle: 0 ° / Tilt series - Axis1 - Max angle: 45 ° |
- 画像解析
画像解析
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 10.0 Å / ソフトウェア - 名称: MRC |
|---|---|
| 結晶パラメータ | 単位格子 - A: 112.4 Å / 単位格子 - B: 111.2 Å / 単位格子 - C: 300.0 Å / 単位格子 - γ: 90.0 ° / 単位格子 - α: 90.0 ° / 単位格子 - β: 90.0 ° / 面群: P 2 21 21 |
| CTF補正 | 詳細: Each crystal |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)