+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11965 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | A reconstruction of Puumala virus-like particle glycoprotein spike lattice in the presence of fab 4G2 which is absent from the reconstruction. | |||||||||
 マップデータ マップデータ | A reconstruction of Puumala virus-like particle glycoprotein spike lattice in the presence of fab 4G2 which is absent from the reconstruction. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Puumala virus - Sotkamo (ウイルス) Puumala virus - Sotkamo (ウイルス) | |||||||||
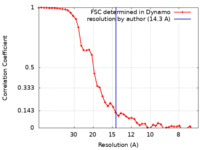
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 14.3 Å | |||||||||
 データ登録者 データ登録者 | Rissanen I / Stass R / Huiskonen JT / Bowden TA | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2020 ジャーナル: Elife / 年: 2020タイトル: Molecular rationale for antibody-mediated targeting of the hantavirus fusion glycoprotein. 著者: Ilona Rissanen / Robert Stass / Stefanie A Krumm / Jeffrey Seow / Ruben Jg Hulswit / Guido C Paesen / Jussi Hepojoki / Olli Vapalahti / Åke Lundkvist / Olivier Reynard / Viktor Volchkov / ...著者: Ilona Rissanen / Robert Stass / Stefanie A Krumm / Jeffrey Seow / Ruben Jg Hulswit / Guido C Paesen / Jussi Hepojoki / Olli Vapalahti / Åke Lundkvist / Olivier Reynard / Viktor Volchkov / Katie J Doores / Juha T Huiskonen / Thomas A Bowden /      要旨: The intricate lattice of Gn and Gc glycoprotein spike complexes on the hantavirus envelope facilitates host-cell entry and is the primary target of the neutralizing antibody-mediated immune response. ...The intricate lattice of Gn and Gc glycoprotein spike complexes on the hantavirus envelope facilitates host-cell entry and is the primary target of the neutralizing antibody-mediated immune response. Through study of a neutralizing monoclonal antibody termed mAb P-4G2, which neutralizes the zoonotic pathogen Puumala virus (PUUV), we provide a molecular-level basis for antibody-mediated targeting of the hantaviral glycoprotein lattice. Crystallographic analysis demonstrates that P-4G2 binds to a multi-domain site on PUUV Gc and may preclude fusogenic rearrangements of the glycoprotein that are required for host-cell entry. Furthermore, cryo-electron microscopy of PUUV-like particles in the presence of P-4G2 reveals a lattice-independent configuration of the Gc, demonstrating that P-4G2 perturbs the (Gn-Gc) lattice. This work provides a structure-based blueprint for rationalizing antibody-mediated targeting of hantaviruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11965.map.gz emd_11965.map.gz | 6.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11965-v30.xml emd-11965-v30.xml emd-11965.xml emd-11965.xml | 13.6 KB 13.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11965_fsc.xml emd_11965_fsc.xml | 4.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11965.png emd_11965.png | 111.9 KB | ||
| マスクデータ |  emd_11965_msk_1.map emd_11965_msk_1.map | 6.9 MB |  マスクマップ マスクマップ | |
| その他 |  emd_11965_half_map_1.map.gz emd_11965_half_map_1.map.gz emd_11965_half_map_2.map.gz emd_11965_half_map_2.map.gz | 5.7 MB 5.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11965 http://ftp.pdbj.org/pub/emdb/structures/EMD-11965 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11965 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11965 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11965_validation.pdf.gz emd_11965_validation.pdf.gz | 535.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11965_full_validation.pdf.gz emd_11965_full_validation.pdf.gz | 534.4 KB | 表示 | |
| XML形式データ |  emd_11965_validation.xml.gz emd_11965_validation.xml.gz | 10.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11965 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11965 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11965 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11965 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11965.map.gz / 形式: CCP4 / 大きさ: 6.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11965.map.gz / 形式: CCP4 / 大きさ: 6.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | A reconstruction of Puumala virus-like particle glycoprotein spike lattice in the presence of fab 4G2 which is absent from the reconstruction. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.52 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
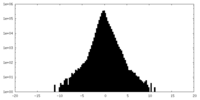
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_11965_msk_1.map emd_11965_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
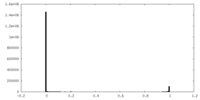
| 密度ヒストグラム |
-ハーフマップ: Half map (even) used to estimate resolution by FSC.
| ファイル | emd_11965_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map (even) used to estimate resolution by FSC. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
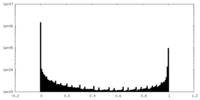
| 密度ヒストグラム |
-ハーフマップ: Half map (odd) used to estimate resolution by FSC.
| ファイル | emd_11965_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map (odd) used to estimate resolution by FSC. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
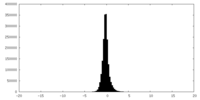
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Puumala virus - Sotkamo
| 全体 | 名称:  Puumala virus - Sotkamo (ウイルス) Puumala virus - Sotkamo (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Puumala virus - Sotkamo
| 超分子 | 名称: Puumala virus - Sotkamo / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 / NCBI-ID: 11606 / 生物種: Puumala virus - Sotkamo / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: Yes / ウイルス・中空状態: Yes |
|---|---|
| 宿主 | 生物種:  Myodes glareolus (ネズミ) Myodes glareolus (ネズミ) |
| Host system | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 / 詳細: PBS |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 4.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)