+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11964 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Puumala virus-like particle glycoprotein spike in complex with fab fragment P-4G2. | |||||||||
 マップデータ マップデータ | A reconstruction of an isolated Puumala virus-like particle glycoprotein spike decorated with four 4G2 fab fragments. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated suppression of host TRAF-mediated signal transduction / host cell Golgi membrane / host cell mitochondrion / host cell surface / host cell endoplasmic reticulum membrane / symbiont-mediated suppression of host innate immune response / virus-mediated perturbation of host defense response / virion membrane / cell surface / signal transduction ...symbiont-mediated suppression of host TRAF-mediated signal transduction / host cell Golgi membrane / host cell mitochondrion / host cell surface / host cell endoplasmic reticulum membrane / symbiont-mediated suppression of host innate immune response / virus-mediated perturbation of host defense response / virion membrane / cell surface / signal transduction / membrane / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Myodes glareolus (ネズミ) / Myodes glareolus (ネズミ) /  Puumala orthohantavirus (ウイルス) / Puumala orthohantavirus (ウイルス) /  Puumala virus - Sotkamo (ウイルス) Puumala virus - Sotkamo (ウイルス) | |||||||||
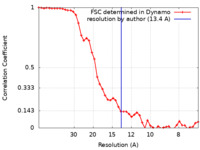
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 13.4 Å | |||||||||
 データ登録者 データ登録者 | Rissanen I / Stass R / Huiskonen JT / Bowden TA | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2020 ジャーナル: Elife / 年: 2020タイトル: Molecular rationale for antibody-mediated targeting of the hantavirus fusion glycoprotein. 著者: Ilona Rissanen / Robert Stass / Stefanie A Krumm / Jeffrey Seow / Ruben Jg Hulswit / Guido C Paesen / Jussi Hepojoki / Olli Vapalahti / Åke Lundkvist / Olivier Reynard / Viktor Volchkov / ...著者: Ilona Rissanen / Robert Stass / Stefanie A Krumm / Jeffrey Seow / Ruben Jg Hulswit / Guido C Paesen / Jussi Hepojoki / Olli Vapalahti / Åke Lundkvist / Olivier Reynard / Viktor Volchkov / Katie J Doores / Juha T Huiskonen / Thomas A Bowden /      要旨: The intricate lattice of Gn and Gc glycoprotein spike complexes on the hantavirus envelope facilitates host-cell entry and is the primary target of the neutralizing antibody-mediated immune response. ...The intricate lattice of Gn and Gc glycoprotein spike complexes on the hantavirus envelope facilitates host-cell entry and is the primary target of the neutralizing antibody-mediated immune response. Through study of a neutralizing monoclonal antibody termed mAb P-4G2, which neutralizes the zoonotic pathogen Puumala virus (PUUV), we provide a molecular-level basis for antibody-mediated targeting of the hantaviral glycoprotein lattice. Crystallographic analysis demonstrates that P-4G2 binds to a multi-domain site on PUUV Gc and may preclude fusogenic rearrangements of the glycoprotein that are required for host-cell entry. Furthermore, cryo-electron microscopy of PUUV-like particles in the presence of P-4G2 reveals a lattice-independent configuration of the Gc, demonstrating that P-4G2 perturbs the (Gn-Gc) lattice. This work provides a structure-based blueprint for rationalizing antibody-mediated targeting of hantaviruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11964.map.gz emd_11964.map.gz | 6.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11964-v30.xml emd-11964-v30.xml emd-11964.xml emd-11964.xml | 19 KB 19 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11964_fsc.xml emd_11964_fsc.xml | 4.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11964.png emd_11964.png | 47.8 KB | ||
| マスクデータ |  emd_11964_msk_1.map emd_11964_msk_1.map | 6.9 MB |  マスクマップ マスクマップ | |
| その他 |  emd_11964_half_map_1.map.gz emd_11964_half_map_1.map.gz emd_11964_half_map_2.map.gz emd_11964_half_map_2.map.gz | 5.5 MB 5.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11964 http://ftp.pdbj.org/pub/emdb/structures/EMD-11964 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11964 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11964 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11964_validation.pdf.gz emd_11964_validation.pdf.gz | 452.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11964_full_validation.pdf.gz emd_11964_full_validation.pdf.gz | 451.9 KB | 表示 | |
| XML形式データ |  emd_11964_validation.xml.gz emd_11964_validation.xml.gz | 10.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11964 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11964 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11964 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11964 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11964.map.gz / 形式: CCP4 / 大きさ: 6.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11964.map.gz / 形式: CCP4 / 大きさ: 6.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | A reconstruction of an isolated Puumala virus-like particle glycoprotein spike decorated with four 4G2 fab fragments. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.52 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
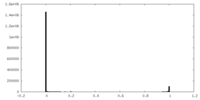
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_11964_msk_1.map emd_11964_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
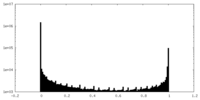
| 密度ヒストグラム |
-ハーフマップ: Half map (odd) used to estimate resolution by FSC.
| ファイル | emd_11964_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map (odd) used to estimate resolution by FSC. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
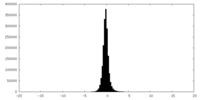
| 密度ヒストグラム |
-ハーフマップ: Half map (even) used to estimate resolution by FSC.
| ファイル | emd_11964_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map (even) used to estimate resolution by FSC. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
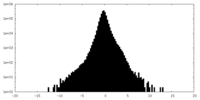
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Puumala virus - Sotkamo
| 全体 | 名称:  Puumala virus - Sotkamo (ウイルス) Puumala virus - Sotkamo (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Puumala virus - Sotkamo
| 超分子 | 名称: Puumala virus - Sotkamo / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 / NCBI-ID: 11606 / 生物種: Puumala virus - Sotkamo / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: Yes / ウイルス・中空状態: Yes |
|---|---|
| 宿主 | 生物種:  Myodes glareolus (ネズミ) Myodes glareolus (ネズミ) |
| Host system | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Heavy chain of fab fragment P-4G2
| 分子 | 名称: Heavy chain of fab fragment P-4G2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Myodes glareolus (ネズミ) Myodes glareolus (ネズミ) |
| 分子量 | 理論値: 25.018047 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: TGQVQMKESG GGLVQPGKSL KLSCAASGFT FSDFWMSWVR QPSGKGLEWV ARINTNGDTT HYTDDMKGRF TISRDNAKTT LYLEMSPLK SEDTAMYYCT RDRLGPFDYW GQGTTVTVSS ATTKGPSVYP LAPGSAAQTN SMVTLGCLVK GYFPEPVTVT W NSGSLSSG ...文字列: TGQVQMKESG GGLVQPGKSL KLSCAASGFT FSDFWMSWVR QPSGKGLEWV ARINTNGDTT HYTDDMKGRF TISRDNAKTT LYLEMSPLK SEDTAMYYCT RDRLGPFDYW GQGTTVTVSS ATTKGPSVYP LAPGSAAQTN SMVTLGCLVK GYFPEPVTVT W NSGSLSSG VHTFPAVLQS DLYTLSSSVT VPSSTWPSQT VTCNVAHPAS STKVDKKIVP RDCGGTKHHH HHH |
-分子 #2: Light chain of fab fragment P-4G2
| 分子 | 名称: Light chain of fab fragment P-4G2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Myodes glareolus (ネズミ) Myodes glareolus (ネズミ) |
| 分子量 | 理論値: 23.881582 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: TGDIVMTQSP AFPPVSLGDT VTITCQASQS VYKNLAWYQQ KPGKSPRLLI FGMSSLADGV PSRFSASGSD KQYSLKIRGL QPEDAAIYY CQQHYIAPYT FGAGTRLEIK RTDAAPTVSI FPPSSEQLTS GGASVVCFLN NFYPKDINVK WKIDGSERQN G VLNSWTDQ ...文字列: TGDIVMTQSP AFPPVSLGDT VTITCQASQS VYKNLAWYQQ KPGKSPRLLI FGMSSLADGV PSRFSASGSD KQYSLKIRGL QPEDAAIYY CQQHYIAPYT FGAGTRLEIK RTDAAPTVSI FPPSSEQLTS GGASVVCFLN NFYPKDINVK WKIDGSERQN G VLNSWTDQ DSKDSTYSMS STLTLTKDEY ERHNSYTCEA THKTSTSPIV KSFNRNEC |
-分子 #3: Envelope polyprotein
| 分子 | 名称: Envelope polyprotein / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Puumala orthohantavirus (ウイルス) Puumala orthohantavirus (ウイルス) |
| 分子量 | 理論値: 49.827898 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GPGETQNLNS GWTDTAHGSG IIPMRTDLEL DFSLPSSASY TYRRQLQNPA NEQEKIPFHL QISKQVIHAE IQHLGHWMDG TFNLKTAFH CYGSCEKYAY PWQTAGCFIE KDYEYESGWG CNPPDCPGVG TGCTACGVYL DKLKSVGKAF KIVSLRYTRK A CIQLGTEQ ...文字列: GPGETQNLNS GWTDTAHGSG IIPMRTDLEL DFSLPSSASY TYRRQLQNPA NEQEKIPFHL QISKQVIHAE IQHLGHWMDG TFNLKTAFH CYGSCEKYAY PWQTAGCFIE KDYEYESGWG CNPPDCPGVG TGCTACGVYL DKLKSVGKAF KIVSLRYTRK A CIQLGTEQ TCKSVDSNDC LVTTSVKVCL IGTVSKFQPS DTLLFLGPLE QGGLIFKQWC TTTCQFGDPG DIMSTPVGMK CP ELNGSFR KKCAFATTPV CQFDGNTLSG YKRMIATKDS FQSFNVTEPH ISASSLEWID PDSSLRDHIN VIVGRDLSFQ DLS ETPCQV DLATTSIDGA WGSGVGFNLV CSVSLTECST FLTSIKACDS AMCYGSTTAN LLRGQNTVHI VGKGGHSGSK FMCC HDTKC SSTGLVAAAP HLDRVTGYNQ ADSDKIFDDG APECGISCWF TKSGEGTETS QVAPA |
-分子 #4: Envelope polyprotein
| 分子 | 名称: Envelope polyprotein / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Puumala orthohantavirus (ウイルス) Puumala orthohantavirus (ウイルス) |
| 分子量 | 理論値: 40.061566 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: ETGELKIECP HTIGLGQGLV IGSVELPPVP LTQVESLKLE SSCNFDLHTS TSSQQPFTKW TWEMKSDLAE NTQASSTSFQ TKSSEINLR GLCLVPPLVI ETAARTRKTI ACFDLSCNQT ACQPTVFLIG PIQTCITTKS CLLGLGDQRI QVNYEKTYCV S GQLVEGVC ...文字列: ETGELKIECP HTIGLGQGLV IGSVELPPVP LTQVESLKLE SSCNFDLHTS TSSQQPFTKW TWEMKSDLAE NTQASSTSFQ TKSSEINLR GLCLVPPLVI ETAARTRKTI ACFDLSCNQT ACQPTVFLIG PIQTCITTKS CLLGLGDQRI QVNYEKTYCV S GQLVEGVC FNPVHTMALS QPSHTYDIVT VMVRCFLIAK KVSTGDSMKL EKSFETLVQK TSCTGNGFQG YYICLVGSSS EP LYIPTLD DYRSAEVLSR MAFAPHGEDH DVEKNAISAM RIIGKVTGKA PSTESSDTIQ GVAFSGNPLY TSTGVLTAKD DPV YIWAPG IIMEGNHSVC DKKTLPLTWT GFIPLPGEIE KTGTKHHHHH H |
-分子 #7: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 7 / コピー数: 1 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: C-flat-2/1 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 4.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)