+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1192 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
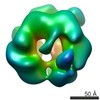
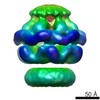
| タイトル | Conformational changes in the AAA ATPase p97-p47 adaptor complex. | |||||||||
 マップデータ マップデータ | AAA ATPase p97 in complex with p47 adaptor, incubated with ADP | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 27.0 Å | |||||||||
 データ登録者 データ登録者 | Beuron F / Dreveny I / Yuan X / Pye VE / McKeown C / Briggs LC / Cliff MJ / Kaneko Y / Wallis R / Isaacson RL ...Beuron F / Dreveny I / Yuan X / Pye VE / McKeown C / Briggs LC / Cliff MJ / Kaneko Y / Wallis R / Isaacson RL / Ladbury JE / Matthews SJ / Kondo H / Zhang X / Freemont PS | |||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2006 ジャーナル: EMBO J / 年: 2006タイトル: Conformational changes in the AAA ATPase p97-p47 adaptor complex. 著者: Fabienne Beuron / Ingrid Dreveny / Xuemei Yuan / Valerie E Pye / Ciaran McKeown / Louise C Briggs / Matthew J Cliff / Yayoi Kaneko / Russell Wallis / Rivka L Isaacson / John E Ladbury / Steve ...著者: Fabienne Beuron / Ingrid Dreveny / Xuemei Yuan / Valerie E Pye / Ciaran McKeown / Louise C Briggs / Matthew J Cliff / Yayoi Kaneko / Russell Wallis / Rivka L Isaacson / John E Ladbury / Steve J Matthews / Hisao Kondo / Xiaodong Zhang / Paul S Freemont /  要旨: The AAA+ATPase p97/VCP, helped by adaptor proteins, exerts its essential role in cellular events such as endoplasmic reticulum-associated protein degradation or the reassembly of Golgi, ER and the ...The AAA+ATPase p97/VCP, helped by adaptor proteins, exerts its essential role in cellular events such as endoplasmic reticulum-associated protein degradation or the reassembly of Golgi, ER and the nuclear envelope after mitosis. Here, we report the three-dimensional cryo-electron microscopy structures at approximately 20 Angstroms resolution in two nucleotide states of the endogenous hexameric p97 in complex with a recombinant p47 trimer, one of the major p97 adaptor proteins involved in membrane fusion. Depending on the nucleotide state, we observe the p47 trimer to be in two distinct arrangements on top of the p97 hexamer. By combining the EM data with NMR and other biophysical measurements, we propose a model of ATP-dependent p97(N) domain motions that lead to a rearrangement of p47 domains, which could result in the disassembly of target protein complexes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1192.map.gz emd_1192.map.gz | 12.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1192-v30.xml emd-1192-v30.xml emd-1192.xml emd-1192.xml | 10.5 KB 10.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1192.gif 1192.gif | 28.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1192 http://ftp.pdbj.org/pub/emdb/structures/EMD-1192 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1192 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1192 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1192_validation.pdf.gz emd_1192_validation.pdf.gz | 193.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1192_full_validation.pdf.gz emd_1192_full_validation.pdf.gz | 192.2 KB | 表示 | |
| XML形式データ |  emd_1192_validation.xml.gz emd_1192_validation.xml.gz | 5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1192 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1192 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1192 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1192 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1192.map.gz / 形式: CCP4 / 大きさ: 12.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1192.map.gz / 形式: CCP4 / 大きさ: 12.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | AAA ATPase p97 in complex with p47 adaptor, incubated with ADP | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.5 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Rat endogenous AAA ATPase p97 bound to p47, 'ADP' form
| 全体 | 名称: Rat endogenous AAA ATPase p97 bound to p47, 'ADP' form |
|---|---|
| 要素 |
|
-超分子 #1000: Rat endogenous AAA ATPase p97 bound to p47, 'ADP' form
| 超分子 | 名称: Rat endogenous AAA ATPase p97 bound to p47, 'ADP' form タイプ: sample / ID: 1000 集合状態: one homohexamer of p97 bound to one trimer of p47 Number unique components: 2 |
|---|---|
| 分子量 | 実験値: 650 KDa / 理論値: 720 KDa / 手法: STEM |
-分子 #1: p97
| 分子 | 名称: p97 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: VCP / コピー数: 6 / 集合状態: homohexamer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 97 MDa / 理論値: 89 MDa |
-分子 #2: p47
| 分子 | 名称: p47 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 集合状態: trimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 47 MDa / 理論値: 41 MDa |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.7 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 20 mM Tris-HCl, 150 mM KCl, 1 mM DTT, pH 7.4 |
| グリッド | 詳細: lacey carbon film |
| 凍結 | 凍結剤: ETHANE / 装置: LEICA KF80 / 詳細: Vitrification instrument: Reichert KF 80 plunger / 手法: blot for 2 seconds befre plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM200FEG |
|---|---|
| 日付 | 2002年6月6日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: OTHER / デジタル化 - サンプリング間隔: 6.35 µm / 実像数: 4 / 平均電子線量: 15 e/Å2 / ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 1.4 mm / 最大 デフォーカス(公称値): 1.9 µm / 最小 デフォーカス(公称値): 1.1 µm / 倍率(公称値): 38000 |
| 試料ステージ | 試料ホルダー: eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| CTF補正 | 詳細: each particle |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C3 (3回回転対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 27.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: Imagic / 使用した粒子像数: 2700 |
| 最終 2次元分類 | クラス数: 44 |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















