+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11906 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | In situ subtomogram averaging structure of the type III secretion system of yersinia enterocolitica - basal body | |||||||||
 マップデータ マップデータ | Subtomogram average structure of the type III secretion system of yersinia enterocolitica - focused on the basal body. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Yersinia enterocolitica (腸炎エルシニア) Yersinia enterocolitica (腸炎エルシニア) | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 33.0 Å | |||||||||
 データ登録者 データ登録者 | Berger C / Ravelli RBG / Lopez-Iglesias C / Kudryashev M / Diepold A / Peters PJ | |||||||||
| 資金援助 |  オランダ, 1件 オランダ, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Struct Biol / 年: 2021 ジャーナル: J Struct Biol / 年: 2021タイトル: Structure of the Yersinia injectisome in intracellular host cell phagosomes revealed by cryo FIB electron tomography. 著者: Casper Berger / Raimond B G Ravelli / Carmen López-Iglesias / Mikhail Kudryashev / Andreas Diepold / Peter J Peters /   要旨: Many pathogenic bacteria use the type III secretion system (T3SS), or injectisome, to secrete toxins into host cells. These protruding systems are primary targets for drug and vaccine development. ...Many pathogenic bacteria use the type III secretion system (T3SS), or injectisome, to secrete toxins into host cells. These protruding systems are primary targets for drug and vaccine development. Upon contact between injectisomes and host membranes, toxin secretion is triggered. How this works structurally and functionally is yet unknown. Using cryo-focused ion beam milling and cryo-electron tomography, we visualized injectisomes of Yersinia enterocolitica inside the phagosomes of infected human myeloid cells in a close-to-native state. We observed that a minimum needle length is required for injectisomes to contact the host membrane and bending of host membranes by some injectisomes that contact the host. Through subtomogram averaging, the structure of the entire injectisome was determined, which revealed structural differences in the cytosolic sorting platform compared to other bacteria. These findings contribute to understanding how injectisomes secrete toxins into host cells and provides the indispensable native context. The application of these cryo-electron microscopy techniques paves the way for the study of the 3D structure of infection-relevant protein complexes in host-pathogen interactions. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11906.map.gz emd_11906.map.gz | 59.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11906-v30.xml emd-11906-v30.xml emd-11906.xml emd-11906.xml | 17.9 KB 17.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11906_fsc.xml emd_11906_fsc.xml | 9.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11906.png emd_11906.png | 77.8 KB | ||
| マスクデータ |  emd_11906_msk_1.map emd_11906_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| その他 |  emd_11906_additional_1.map.gz emd_11906_additional_1.map.gz emd_11906_half_map_1.map.gz emd_11906_half_map_1.map.gz emd_11906_half_map_2.map.gz emd_11906_half_map_2.map.gz | 58.7 MB 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11906 http://ftp.pdbj.org/pub/emdb/structures/EMD-11906 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11906 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11906 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11906_validation.pdf.gz emd_11906_validation.pdf.gz | 842.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11906_full_validation.pdf.gz emd_11906_full_validation.pdf.gz | 841.3 KB | 表示 | |
| XML形式データ |  emd_11906_validation.xml.gz emd_11906_validation.xml.gz | 14.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11906 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11906 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11906 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11906 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11906.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11906.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Subtomogram average structure of the type III secretion system of yersinia enterocolitica - focused on the basal body. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 5.452 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_11906_msk_1.map emd_11906_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
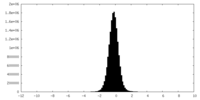
| 密度ヒストグラム |
-追加マップ: Subtomogram average structure of the type III secretion...
| ファイル | emd_11906_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Subtomogram average structure of the type III secretion system of yersinia enterocolitica - focused on the basal body and filtered to 3.3 nm resolution. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
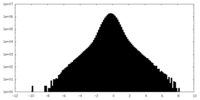
| 密度ヒストグラム |
-ハーフマップ: half map of all the even particles
| ファイル | emd_11906_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map of all the even particles | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
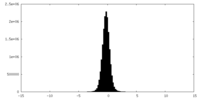
| 密度ヒストグラム |
-ハーフマップ: half map of all the odd particles
| ファイル | emd_11906_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map of all the odd particles | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
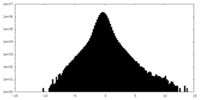
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Basal body of the Type III secretion system of Yersinia enterocolitica
| 全体 | 名称: Basal body of the Type III secretion system of Yersinia enterocolitica |
|---|---|
| 要素 |
|
-超分子 #1: Basal body of the Type III secretion system of Yersinia enterocolitica
| 超分子 | 名称: Basal body of the Type III secretion system of Yersinia enterocolitica タイプ: cell / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Yersinia enterocolitica (腸炎エルシニア) / 株: E40 Yersinia enterocolitica (腸炎エルシニア) / 株: E40 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 詳細: CElls were grown in RPMI 1640 medium on the grid. 2 ul of RPMI 1640 was applied to the grid just prior to vitrification |
|---|---|
| グリッド | モデル: UltrAuFoil R2/2 / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 85 % / チャンバー内温度: 310 K / 装置: HOMEMADE PLUNGER 詳細: FEI vitrobot modified with a jet-vitrification module and a blotforce feedback mechanism.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 平均露光時間: 24.0 sec. / 平均電子線量: 2.86 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最小 デフォーカス(公称値): -0.005 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)