+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1155 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The mechanism of HIV-1 core assembly: insights from three-dimensional reconstructions of authentic virions. | |||||||||
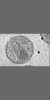
 マップデータ マップデータ | Tomographic reconstruction of HIV-1 virions | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Human immunodeficiency virus (ヒト免疫不全ウイルス) Human immunodeficiency virus (ヒト免疫不全ウイルス) | |||||||||
| 手法 | 電子線トモグラフィー法 / クライオ電子顕微鏡法 / ネガティブ染色法 | |||||||||
 データ登録者 データ登録者 | Briggs JAG / Grunewald K / Glass B / Forster F | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2006 ジャーナル: Structure / 年: 2006タイトル: The mechanism of HIV-1 core assembly: insights from three-dimensional reconstructions of authentic virions. 著者: John A G Briggs / Kay Grünewald / Bärbel Glass / Friedrich Förster / Hans-Georg Kräusslich / Stephen D Fuller /  要旨: Infectious HIV particles contain a characteristic cone-shaped core encasing the viral RNA and replication proteins. The core exhibits significant heterogeneity in size and shape, yet consistently ...Infectious HIV particles contain a characteristic cone-shaped core encasing the viral RNA and replication proteins. The core exhibits significant heterogeneity in size and shape, yet consistently forms a well-defined structure. The mechanism by which the core is assembled in the maturing virion remains poorly understood. Using cryo-electron tomography, we have produced three-dimensional reconstructions of authentic, unstained HIV-1. These reveal the viral morphology with unprecedented clarity and suggest the following mechanism for core formation inside the extracellular virion: core growth initiates at the narrow end of the cone and proceeds toward the distal side of the virion until limited by the viral membrane. Curvature and closure of the broad end of the core are then directed by the inner surface of the viral membrane. This mechanism accommodates significant flexibility in lattice growth while ensuring the closure of cores of variable size and shape. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1155.map.gz emd_1155.map.gz | 2.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1155-v30.xml emd-1155-v30.xml emd-1155.xml emd-1155.xml | 8.2 KB 8.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1155.gif 1155.gif | 33 KB | ||
| その他 |  emd_1155_additional_1.map.gz emd_1155_additional_1.map.gz | 3.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1155 http://ftp.pdbj.org/pub/emdb/structures/EMD-1155 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1155 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1155 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1155_validation.pdf.gz emd_1155_validation.pdf.gz | 208.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1155_full_validation.pdf.gz emd_1155_full_validation.pdf.gz | 207.7 KB | 表示 | |
| XML形式データ |  emd_1155_validation.xml.gz emd_1155_validation.xml.gz | 4.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1155 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1155 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1155 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1155 | HTTPS FTP |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1155.map.gz / 形式: CCP4 / 大きさ: 3.4 MB / タイプ: IMAGE STORED AS SIGNED BYTE ダウンロード / ファイル: emd_1155.map.gz / 形式: CCP4 / 大きさ: 3.4 MB / タイプ: IMAGE STORED AS SIGNED BYTE | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Tomographic reconstruction of HIV-1 virions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
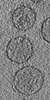
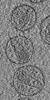
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 16.4 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-添付マップデータ: emd 1155 additional 1.map
| ファイル | emd_1155_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
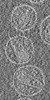
| 投影像・断面図 |
| ||||||||||||
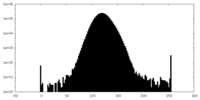
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : HIV-1 virions
| 全体 | 名称: HIV-1 virions |
|---|---|
| 要素 |
|
-超分子 #1000: HIV-1 virions
| 超分子 | 名称: HIV-1 virions / タイプ: sample / ID: 1000 / Number unique components: 1 |
|---|
-超分子 #1: Human immunodeficiency virus
| 超分子 | 名称: Human immunodeficiency virus / タイプ: virus / ID: 1 / Name.synonym: HIV-1 / NCBI-ID: 12721 / 生物種: Human immunodeficiency virus / データベース: NCBI / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No / Syn species name: HIV-1 |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) / 別称: VERTEBRATES Homo sapiens (ヒト) / 別称: VERTEBRATES |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線トモグラフィー法 |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 / 詳細: PBS |
|---|---|
| 染色 | タイプ: NEGATIVE / 詳細: unstained |
| グリッド | 詳細: holey carbon film |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM300FEG/T |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF2000 エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 日付 | 2004年10月22日 |
| 撮影 | 平均電子線量: 50 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 36500 / 照射モード: FLOOD BEAM / 撮影モード: OTHER / Cs: 2.0 mm / 最大 デフォーカス(公称値): 6.5 µm / 最小 デフォーカス(公称値): 5.5 µm |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN / Tilt series - Axis1 - Min angle: 68 ° / Tilt series - Axis1 - Max angle: 68 ° / Tilt series - Axis1 - Angle increment: 2 ° |
- 画像解析
画像解析
| 最終 再構成 | アルゴリズム: OTHER / ソフトウェア - 名称:  IMOD IMOD |
|---|
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)