+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1134 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Polymorphism and double hexamer structure in the archaeal minichromosome maintenance (MCM) helicase from Methanobacterium thermoautotrophicum. | |||||||||
 マップデータ マップデータ | test map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | MCM domain / DNA replication initiation 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Methanothermobacter thermautotrophicus (古細菌) Methanothermobacter thermautotrophicus (古細菌) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 25.0 Å | |||||||||
 データ登録者 データ登録者 | Gomez-Llorente Y / Fletcher RJ / Chen XS / Carazo JM / San Martin C | |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2005 ジャーナル: J Biol Chem / 年: 2005タイトル: Polymorphism and double hexamer structure in the archaeal minichromosome maintenance (MCM) helicase from Methanobacterium thermoautotrophicum. 著者: Yacob Gómez-Llorente / Ryan J Fletcher / Xiaojiang S Chen / José M Carazo / Carmen San Martín /  要旨: Methanobacterium thermoautotrophicum minichromosome maintenance complex (mtMCM), a cellular replicative helicase, is a useful model for the more complex eukaryotic MCMs. Biochemical and ...Methanobacterium thermoautotrophicum minichromosome maintenance complex (mtMCM), a cellular replicative helicase, is a useful model for the more complex eukaryotic MCMs. Biochemical and crystallographic evidence indicates that mtMCM assembles as a double hexamer (dHex), but previous electron microscopy studies reported only the presence of single heptamers or single hexamers (Pape, T., Meka, H., Chen, S., Vicentini, G., Van Heel, M., and Onesti, S. (2003) EMBO Rep. 4, 1079-1083; Yu, X., VanLoock, M. S., Poplawski, A., Kelman, Z., Xiang, T., Tye, B. K., and Egelman, E. H. (2002) EMBO Rep. 3, 792-797). Here we present the first three-dimensional electron microscopy reconstruction of the full-length mtMCM dHex in which two hexamers contact each other via the structurally well defined N-terminal domains. The dHex has obvious side openings that resemble the side channels of LTag (large T antigen). 6-fold and 7-fold rings were observed in the same mtMCM preparation, but we determined that assembly as a double ring favors 6-fold structures. Additionally, open rings were also detected, which suggests a direct mtMCM loading mechanism onto DNA. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1134.map.gz emd_1134.map.gz | 2.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1134-v30.xml emd-1134-v30.xml emd-1134.xml emd-1134.xml | 9.4 KB 9.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1134.gif 1134.gif | 10.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1134 http://ftp.pdbj.org/pub/emdb/structures/EMD-1134 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1134 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1134 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1134_validation.pdf.gz emd_1134_validation.pdf.gz | 217.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1134_full_validation.pdf.gz emd_1134_full_validation.pdf.gz | 216.8 KB | 表示 | |
| XML形式データ |  emd_1134_validation.xml.gz emd_1134_validation.xml.gz | 4.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1134 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1134 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1134 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1134 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1134.map.gz / 形式: CCP4 / 大きさ: 3.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1134.map.gz / 形式: CCP4 / 大きさ: 3.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | test map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.5 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Archaeal helicase MCM from Methanobacterium thermoautotrophicum
| 全体 | 名称: Archaeal helicase MCM from Methanobacterium thermoautotrophicum |
|---|---|
| 要素 |
|
-超分子 #1000: Archaeal helicase MCM from Methanobacterium thermoautotrophicum
| 超分子 | 名称: Archaeal helicase MCM from Methanobacterium thermoautotrophicum タイプ: sample / ID: 1000 / 集合状態: homododecamer / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 900 KDa / 理論値: 900 KDa / 手法: gel filtration |
-分子 #1: minichromosome maintenance protein
| 分子 | 名称: minichromosome maintenance protein / タイプ: protein_or_peptide / ID: 1 / Name.synonym: MCM / コピー数: 12 / 集合状態: dodecamer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Methanothermobacter thermautotrophicus (古細菌) Methanothermobacter thermautotrophicus (古細菌)株: Delta H / 別称: Methanobacterium thermoautotrophicum |
| 分子量 | 実験値: 75.6 MDa / 理論値: 75.6 MDa |
| 組換発現 | 生物種:  |
| 配列 | GO: DNA replication initiation / InterPro: MCM domain |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: 50 mM Tris.HCl pH 8.0, 1 mM DTT, 50 mM to 1M NaCl |
| 染色 | タイプ: NEGATIVE / 詳細: 2% uranyl acetate |
| グリッド | 詳細: glow discharged, collodion/carbon coated copper grids |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 1200EXII |
|---|---|
| アライメント法 | Legacy - 非点収差: visually corrected at 100,000x |
| 詳細 | he make and model of the microscope. Jeol 1200 EX-II. Standard Jeol 1200 holder |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 平均電子線量: 10 e/Å2 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 80 kV / 電子線源: TUNGSTEN HAIRPIN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 5.6 mm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダー: side entry / 試料ホルダーモデル: OTHER |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C6 (6回回転対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 25.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: SPIDER, XMIPP / 詳細: reconstructed with ART / 使用した粒子像数: 1200 |
|---|---|
| 最終 角度割当 | 詳細: theta between 75 and 90, phi between 0 and 360 |
-原子モデル構築 1
| ソフトウェア | 名称: Amira |
|---|
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera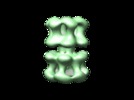





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















