[日本語] English
 万見
万見- EMDB-11082: Cryo-EM structure of the A. baumannii MlaBDEF complex bound to APPNHP -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11082 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the A. baumannii MlaBDEF complex bound to APPNHP | |||||||||
 マップデータ マップデータ | Map of the A. baumannii MlaBDEF complex, bound to APPNHP, at 3.9A resolution. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | lipid transport / antibiotic resistance / ABC transporter / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報phospholipid transporter activity / ATP-binding cassette (ABC) transporter complex / phospholipid binding / ATP hydrolysis activity / ATP binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Acinetobacter baumannii (バクテリア) Acinetobacter baumannii (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Mann D / Bergeron JRC | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2021 ジャーナル: Commun Biol / 年: 2021タイトル: Structure and lipid dynamics in the maintenance of lipid asymmetry inner membrane complex of A. baumannii. 著者: Daniel Mann / Junping Fan / Kamolrat Somboon / Daniel P Farrell / Andrew Muenks / Svetomir B Tzokov / Frank DiMaio / Syma Khalid / Samuel I Miller / Julien R C Bergeron /     要旨: Multi-resistant bacteria are a major threat in modern medicine. The gram-negative coccobacillus Acinetobacter baumannii currently leads the WHO list of pathogens in critical need for new therapeutic ...Multi-resistant bacteria are a major threat in modern medicine. The gram-negative coccobacillus Acinetobacter baumannii currently leads the WHO list of pathogens in critical need for new therapeutic development. The maintenance of lipid asymmetry (MLA) protein complex is one of the core machineries that transport lipids from/to the outer membrane in gram-negative bacteria. It also contributes to broad-range antibiotic resistance in several pathogens, most prominently in A. baumannii. Nonetheless, the molecular details of its role in lipid transport has remained largely elusive. Here, we report the cryo-EM maps of the core MLA complex, MlaBDEF, from the pathogen A. baumannii, in the apo-, ATP- and ADP-bound states, revealing multiple lipid binding sites in the cytosolic and periplasmic side of the complex. Molecular dynamics simulations suggest their potential trajectory across the membrane. Collectively with the recently-reported structures of the E. coli orthologue, this data also allows us to propose a molecular mechanism of lipid transport by the MLA system. #1:  ジャーナル: Biorxiv / 年: 2020 ジャーナル: Biorxiv / 年: 2020タイトル: Structure and lipid dynamics in the A. baumannii maintenance of lipid asymmetry (MLA) inner membrane complex 著者: Mann D / Fan J / Farrell DP / Somboon K / Muenks A / Tzokov SB / Khalid S / Dimaio F / Miller SI / Bergeron JRC | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11082.map.gz emd_11082.map.gz | 59.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11082-v30.xml emd-11082-v30.xml emd-11082.xml emd-11082.xml | 15 KB 15 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_11082.png emd_11082.png | 168.6 KB | ||
| マスクデータ |  emd_11082_msk_1.map emd_11082_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-11082.cif.gz emd-11082.cif.gz | 6.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11082 http://ftp.pdbj.org/pub/emdb/structures/EMD-11082 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11082 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11082 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11082_validation.pdf.gz emd_11082_validation.pdf.gz | 291.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11082_full_validation.pdf.gz emd_11082_full_validation.pdf.gz | 291 KB | 表示 | |
| XML形式データ |  emd_11082_validation.xml.gz emd_11082_validation.xml.gz | 6.3 KB | 表示 | |
| CIF形式データ |  emd_11082_validation.cif.gz emd_11082_validation.cif.gz | 7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11082 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11082 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11082 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11082 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6z5uMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10425 (タイトル: Cryo-EM structure of the A.baumannii MlaBDEF complex bound to AppNHp EMPIAR-10425 (タイトル: Cryo-EM structure of the A.baumannii MlaBDEF complex bound to AppNHpData size: 135.6 Data #1: MotionCor2 aligned micrographs [micrographs - single frame]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11082.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11082.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map of the A. baumannii MlaBDEF complex, bound to APPNHP, at 3.9A resolution. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
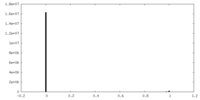
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_11082_msk_1.map emd_11082_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
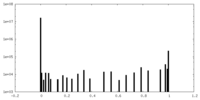
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : MlaBDEF complex
| 全体 | 名称: MlaBDEF complex |
|---|---|
| 要素 |
|
-超分子 #1: MlaBDEF complex
| 超分子 | 名称: MlaBDEF complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  Acinetobacter baumannii (バクテリア) Acinetobacter baumannii (バクテリア) |
-分子 #1: ABC transporter permease
| 分子 | 名称: ABC transporter permease / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Acinetobacter baumannii (バクテリア) Acinetobacter baumannii (バクテリア) |
| 分子量 | 理論値: 27.322443 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNTIAWLGRL VIERIRGIGV AALMLLQIIF SLPSAGGFGR FVYQMHRVGV MSLLIITVSG LFIGLVLGLQ GYSILVNVGS ESMLGTMVS LTLLRELAPV VAALLFAGRA GSALTAEIGS MKQSEQLASM EMIGVDPLKQ IVSPRLWAGI VSLPMLTVIF A AIGIVGGK ...文字列: MNTIAWLGRL VIERIRGIGV AALMLLQIIF SLPSAGGFGR FVYQMHRVGV MSLLIITVSG LFIGLVLGLQ GYSILVNVGS ESMLGTMVS LTLLRELAPV VAALLFAGRA GSALTAEIGS MKQSEQLASM EMIGVDPLKQ IVSPRLWAGI VSLPMLTVIF A AIGIVGGK LVGVDFLGVD EGSFWSGMQN NVQFGHDVVN GIIKSIVFAL LCTWIAVFQG YACDPTPEGI ATAMTRTVVY SS LCVLGFD FVLTAVMFGG I UniProtKB: Intermembrane phospholipid transport system permease protein MlaE |
-分子 #2: Anti-sigma factor antagonist
| 分子 | 名称: Anti-sigma factor antagonist / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Acinetobacter baumannii (バクテリア) Acinetobacter baumannii (バクテリア) |
| 分子量 | 理論値: 10.874672 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MVQYLNQELV VSGKIDFENA EQQYQAGLAI IKKQTSFPLI VDLKQLEHGN TLALAVLVQW LRQTPQKSGL HFKNVPEKML KIIQACHLQ EDLHLV UniProtKB: Anti-sigma factor antagonist |
-分子 #3: MCE family protein
| 分子 | 名称: MCE family protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Acinetobacter baumannii (バクテリア) Acinetobacter baumannii (バクテリア) |
| 分子量 | 理論値: 24.135322 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKSRTSELAV GIFVIIFGIA LFFLAMKVSG LVGTNLSDGY TMKAQFDNVN GLKPRAKVTM SGVTIGRVDS ITLDPVTRLA TVTFDLDGK LTSFNAEQLK EVQKNALDEL RYSSDYTQAT PAQQKTMEQQ LISNMNSITS IDEDAYIMVA TNGLLGEKYL K IVPGGGLN ...文字列: MKSRTSELAV GIFVIIFGIA LFFLAMKVSG LVGTNLSDGY TMKAQFDNVN GLKPRAKVTM SGVTIGRVDS ITLDPVTRLA TVTFDLDGK LTSFNAEQLK EVQKNALDEL RYSSDYTQAT PAQQKTMEQQ LISNMNSITS IDEDAYIMVA TNGLLGEKYL K IVPGGGLN YLKRGDTISN TQGTMDLEDL ISKFITGGGA GKVAAGSSSA EEKAPASTDS SAQPSFVE UniProtKB: ABC transporter periplasmic substrate-binding protein |
-分子 #4: ABC transporter ATP-binding protein
| 分子 | 名称: ABC transporter ATP-binding protein / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Acinetobacter baumannii (バクテリア) Acinetobacter baumannii (バクテリア) |
| 分子量 | 理論値: 30.367848 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MIAIMNNKTP LSTQSLIEVK NLSFNRGERV IYDNISLNIR RGQITAIMGP SGTGKTTLLR LIGGQLVPDQ GEVLLDGKDI AQMSRQELF AARARMGMLF QSGALFTDMS VYENVAFPIR AHTKLSENLI AELVALKLES VGLRGTEQLM PTELSGGMNR R VALARAIA ...文字列: MIAIMNNKTP LSTQSLIEVK NLSFNRGERV IYDNISLNIR RGQITAIMGP SGTGKTTLLR LIGGQLVPDQ GEVLLDGKDI AQMSRQELF AARARMGMLF QSGALFTDMS VYENVAFPIR AHTKLSENLI AELVALKLES VGLRGTEQLM PTELSGGMNR R VALARAIA LDPDLIMYDE PFAGQDPIVK GVLTRLIRSL REALDLTTII VSHDVPETLS IADYIYVVAE GKIQGEGTPE EL QAYASPF VKQFLTGSAE GPVEYQFSHQ AYLDNEVRP UniProtKB: ABC transporter ATP-binding protein |
-分子 #5: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 5 / コピー数: 2 / 式: ANP |
|---|---|
| 分子量 | 理論値: 506.196 Da |
| Chemical component information |  ChemComp-ANP: |
-分子 #6: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 6 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 47.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.9 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 93295 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)