+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10725 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | OP protein cage, complex 2 | |||||||||
 マップデータ マップデータ | OP protein cage, complex 2 | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Salmonella enterica (サルモネラ菌) Salmonella enterica (サルモネラ菌) | |||||||||
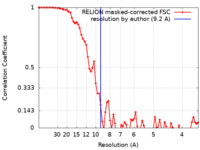
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 9.2 Å | |||||||||
 データ登録者 データ登録者 | Edwardson TGW / Tetter S / Hilvert D | |||||||||
| 資金援助 |  スイス, 2件 スイス, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Two-tier supramolecular encapsulation of small molecules in a protein cage. 著者: Thomas G W Edwardson / Stephan Tetter / Donald Hilvert /  要旨: Expanding protein design to include other molecular building blocks has the potential to increase structural complexity and practical utility. Nature often employs hybrid systems, such as clathrin- ...Expanding protein design to include other molecular building blocks has the potential to increase structural complexity and practical utility. Nature often employs hybrid systems, such as clathrin-coated vesicles, lipid droplets, and lipoproteins, which combine biopolymers and lipids to transport a broader range of cargo molecules. To recapitulate the structure and function of such composite compartments, we devised a supramolecular strategy that enables porous protein cages to encapsulate poorly water-soluble small molecule cargo through templated formation of a hydrophobic surfactant-based core. These lipoprotein-like complexes protect their cargo from sequestration by serum proteins and enhance the cellular uptake of fluorescent probes and cytotoxic drugs. This design concept could be applied to other protein cages, surfactant mixtures, and cargo molecules to generate unique hybrid architectures and functional capabilities. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10725.map.gz emd_10725.map.gz | 27.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10725-v30.xml emd-10725-v30.xml emd-10725.xml emd-10725.xml | 17.2 KB 17.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10725_fsc.xml emd_10725_fsc.xml | 7.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10725.png emd_10725.png | 114.4 KB | ||
| マスクデータ |  emd_10725_msk_1.map emd_10725_msk_1.map | 30.5 MB |  マスクマップ マスクマップ | |
| その他 |  emd_10725_half_map_1.map.gz emd_10725_half_map_1.map.gz emd_10725_half_map_2.map.gz emd_10725_half_map_2.map.gz | 20.9 MB 20.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10725 http://ftp.pdbj.org/pub/emdb/structures/EMD-10725 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10725 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10725 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10725_validation.pdf.gz emd_10725_validation.pdf.gz | 339.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10725_full_validation.pdf.gz emd_10725_full_validation.pdf.gz | 338.5 KB | 表示 | |
| XML形式データ |  emd_10725_validation.xml.gz emd_10725_validation.xml.gz | 12.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10725 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10725 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10725 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10725 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10725.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10725.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | OP protein cage, complex 2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.8 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
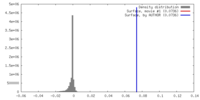
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10725_msk_1.map emd_10725_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
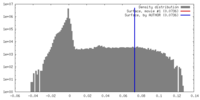
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_10725_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
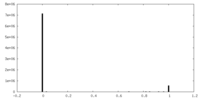
| 投影像・断面図 |
| ||||||||||||
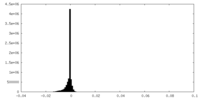
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_10725_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : OP protein cage
| 全体 | 名称: OP protein cage |
|---|---|
| 要素 |
|
-超分子 #1: OP protein cage
| 超分子 | 名称: OP protein cage / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Positively supercharged variant of the computationally designed cage protein O3-33 |
|---|---|
| 由来(天然) | 生物種:  Salmonella enterica (サルモネラ菌) Salmonella enterica (サルモネラ菌) |
| 組換発現 | 生物種:  |
-分子 #1: OP protein cage, complex 2
| 分子 | 名称: OP protein cage, complex 2 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Salmonella enterica (サルモネラ菌) Salmonella enterica (サルモネラ菌) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSQAIGILEL RSIAAGMELG DAMLKSANVD LLVSKTISRG KFLLMLGGDI GAIQQAIETG TSQAGRLLVD SLVLANIHPS VLPAISGLNS VDKRQAVGIV ETRSVAACIS AADRAVKGSN VTLVRVHMAR GIGGKCYMVV AGDVSDVALA VTVASSSAGA YGRLVYASLI ...文字列: MSQAIGILEL RSIAAGMELG DAMLKSANVD LLVSKTISRG KFLLMLGGDI GAIQQAIETG TSQAGRLLVD SLVLANIHPS VLPAISGLNS VDKRQAVGIV ETRSVAACIS AADRAVKGSN VTLVRVHMAR GIGGKCYMVV AGDVSDVALA VTVASSSAGA YGRLVYASLI PRPHEAMWRQ MVEGLEHHHH HH |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 詳細: 25 mM Tris-HCl, 200 mM NaCl, 5 mM EDTA, pH 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 22 K / 詳細: blot for 12 s, 25 blot strength, before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - 画像ごとのフレーム数: 1-7 / 撮影したグリッド数: 1 / 実像数: 127 / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): -3.3 µm / 最小 デフォーカス(公称値): -1.8 µm / 倍率(公称値): 62000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Correlation coefficient |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)