+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10632 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of glutamate transporter homologue GltTk in the presence of TBOA inhibitor | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | AMINO ACID TRANSPORTER / ASPARTATE TRANSPORT / GLUTAMATE TRANSPORTER HOMOLOGUE / TRANSPORT PROTEIN / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Thermococcus kodakarensis KOD1 (古細菌) / Thermococcus kodakarensis KOD1 (古細菌) /   Thermococcus kodakarensis (strain ATCC BAA-918 / JCM 12380 / KOD1) (古細菌) Thermococcus kodakarensis (strain ATCC BAA-918 / JCM 12380 / KOD1) (古細菌) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.47 Å | |||||||||
 データ登録者 データ登録者 | Arkhipova V / Slotboom DJ | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Structural ensemble of a glutamate transporter homologue in lipid nanodisc environment. 著者: Valentina Arkhipova / Albert Guskov / Dirk J Slotboom /   要旨: Glutamate transporters are cation-coupled secondary active membrane transporters that clear the neurotransmitter L-glutamate from the synaptic cleft. These transporters are homotrimers, with each ...Glutamate transporters are cation-coupled secondary active membrane transporters that clear the neurotransmitter L-glutamate from the synaptic cleft. These transporters are homotrimers, with each protomer functioning independently by an elevator-type mechanism, in which a mobile transport domain alternates between inward- and outward-oriented states. Using single-particle cryo-EM we have determined five structures of the glutamate transporter homologue Glt, a Na- L-aspartate symporter, embedded in lipid nanodiscs. Dependent on the substrate concentrations used, the protomers of the trimer adopt a variety of asymmetrical conformations, consistent with the independent movement. Six of the 15 resolved protomers are in a hitherto elusive state of the transport cycle in which the inward-facing transporters are loaded with Na ions. These structures explain how substrate-leakage is prevented - a strict requirement for coupled transport. The belt protein of the lipid nanodiscs bends around the inward oriented protomers, suggesting that membrane deformations occur during transport. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10632.map.gz emd_10632.map.gz | 32.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10632-v30.xml emd-10632-v30.xml emd-10632.xml emd-10632.xml | 14 KB 14 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_10632.png emd_10632.png | 103.7 KB | ||
| Filedesc metadata |  emd-10632.cif.gz emd-10632.cif.gz | 5.8 KB | ||
| その他 |  emd_10632_additional.map.gz emd_10632_additional.map.gz | 57 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10632 http://ftp.pdbj.org/pub/emdb/structures/EMD-10632 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10632 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10632 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10632_validation.pdf.gz emd_10632_validation.pdf.gz | 477.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10632_full_validation.pdf.gz emd_10632_full_validation.pdf.gz | 476.6 KB | 表示 | |
| XML形式データ |  emd_10632_validation.xml.gz emd_10632_validation.xml.gz | 6.2 KB | 表示 | |
| CIF形式データ |  emd_10632_validation.cif.gz emd_10632_validation.cif.gz | 7.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10632 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10632 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10632 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10632 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10632.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10632.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.012 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: sharpened map
| ファイル | emd_10632_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
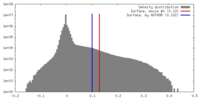
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Trimer of GltTk
| 全体 | 名称: Trimer of GltTk |
|---|---|
| 要素 |
|
-超分子 #1: Trimer of GltTk
| 超分子 | 名称: Trimer of GltTk / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Thermococcus kodakarensis KOD1 (古細菌) Thermococcus kodakarensis KOD1 (古細菌) |
| 分子量 | 理論値: 136 KDa |
-分子 #1: Proton/glutamate symporter, SDF family
| 分子 | 名称: Proton/glutamate symporter, SDF family / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Thermococcus kodakarensis (strain ATCC BAA-918 / JCM 12380 / KOD1) (古細菌) Thermococcus kodakarensis (strain ATCC BAA-918 / JCM 12380 / KOD1) (古細菌) |
| 分子量 | 理論値: 45.580203 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGKSLLRRYL DYPVLWKILW GLVLGAVFGL IAGHFGYAGA VKTYIKPFGD LFVRLLKMLV MPIVLASLVV GAASISPARL GRVGVKIVV YYLATSAMAV FFGLIVGRLF NVGANVNLGS GTGKAIEAQP PSLVQTLLNI VPTNPFASLA KGEVLPVIFF A IILGIAIT ...文字列: MGKSLLRRYL DYPVLWKILW GLVLGAVFGL IAGHFGYAGA VKTYIKPFGD LFVRLLKMLV MPIVLASLVV GAASISPARL GRVGVKIVV YYLATSAMAV FFGLIVGRLF NVGANVNLGS GTGKAIEAQP PSLVQTLLNI VPTNPFASLA KGEVLPVIFF A IILGIAIT YLMNRNEERV RKSAETLLRV FDGLAEAMYL IVGGVMQYAP IGVFALIAYV MAEQGVRVVG PLAKVVGAVY TG LFLQIVI TYFILLKVFG IDPIKFIRKA KDAMITAFVT RSSSGTLPVT MRVAEEEMGV DKGIFSFTLP LGATINMDGT ALY QGVTVL FVANAIGHPL TLGQQLVVVL TAVLASIGTA GVPGAGAIML AMVLQSVGLD LTPGSPVALA YAMILGIDAI LDMG RTMVN VTGDLAGTVI VAKTEKELDE SKWIS UniProtKB: Proton/glutamate symporter, SDF family |
-分子 #2: (3S)-3-(BENZYLOXY)-L-ASPARTIC ACID
| 分子 | 名称: (3S)-3-(BENZYLOXY)-L-ASPARTIC ACID / タイプ: ligand / ID: 2 / コピー数: 2 / 式: TB1 |
|---|---|
| 分子量 | 理論値: 239.225 Da |
| Chemical component information | 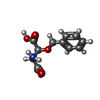 ChemComp-TB1: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / 平均電子線量: 53.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)