登録情報 データベース : EMDB / ID : EMD-0874タイトル The cryo-EM structure of E. coli CueR transcription activation complex 複合体 : Escherichia coli CueR transcription activation complexタンパク質・ペプチド : x 6種DNA : x 2種RNA : x 1種リガンド : x 3種 / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
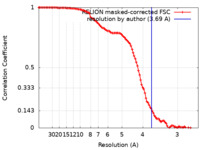
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Escherichia coli K-12 (大腸菌) / Escherichia coli (strain K12) (大腸菌) / synthetic construct (人工物) 手法 / / 解像度 : 3.69 Å Fang CL / Zhang Y 資金援助 Organization Grant number 国 National Natural Science Foundation of China 31822001
ジャーナル : Nat Chem Biol / 年 : 2021タイトル : CueR activates transcription through a DNA distortion mechanism.著者 : Chengli Fang / Steven J Philips / Xiaoxian Wu / Kui Chen / Jing Shi / Liqiang Shen / Juncao Xu / Yu Feng / Thomas V O'Halloran / Yu Zhang / 要旨 : The MerR-family transcription factors (TFs) are a large group of bacterial proteins responding to cellular metal ions and multiple antibiotics by binding within central RNA polymerase-binding regions ... The MerR-family transcription factors (TFs) are a large group of bacterial proteins responding to cellular metal ions and multiple antibiotics by binding within central RNA polymerase-binding regions of a promoter. While most TFs alter transcription through protein-protein interactions, MerR TFs are capable of reshaping promoter DNA. To address the question of which mechanism prevails, we determined two cryo-EM structures of transcription activation complexes (TAC) comprising Escherichia coli CueR (a prototype MerR TF), RNAP holoenzyme and promoter DNA. The structures reveal that this TF promotes productive promoter-polymerase association without canonical protein-protein contacts seen between other activator proteins and RNAP. Instead, CueR realigns the key promoter elements in the transcription activation complex by clamp-like protein-DNA interactions: these induce four distinct kinks that ultimately position the -10 element for formation of the transcription bubble. These structural and biochemical results provide strong support for the DNA distortion paradigm of allosteric transcriptional control by MerR TFs. 履歴 登録 2019年11月21日 - ヘッダ(付随情報) 公開 2020年9月30日 - マップ公開 2020年9月30日 - 更新 2024年3月27日 - 現状 2024年3月27日 処理サイト : PDBj / 状態 : 公開
すべて表示 表示を減らす
 万見
万見 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報

 データ登録者
データ登録者 中国, 1件
中国, 1件  引用
引用 ジャーナル: Nat Chem Biol / 年: 2021
ジャーナル: Nat Chem Biol / 年: 2021

 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_0874.map.gz
emd_0874.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-0874-v30.xml
emd-0874-v30.xml emd-0874.xml
emd-0874.xml EMDBヘッダ
EMDBヘッダ emd_0874_fsc.xml
emd_0874_fsc.xml FSCデータファイル
FSCデータファイル emd_0874.png
emd_0874.png emd_0874_msk_1.map
emd_0874_msk_1.map マスクマップ
マスクマップ emd-0874.cif.gz
emd-0874.cif.gz emd_0874_half_map_1.map.gz
emd_0874_half_map_1.map.gz emd_0874_half_map_2.map.gz
emd_0874_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-0874
http://ftp.pdbj.org/pub/emdb/structures/EMD-0874 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0874
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0874 emd_0874_validation.pdf.gz
emd_0874_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_0874_full_validation.pdf.gz
emd_0874_full_validation.pdf.gz emd_0874_validation.xml.gz
emd_0874_validation.xml.gz emd_0874_validation.cif.gz
emd_0874_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0874
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0874 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0874
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0874 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_0874.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_0874.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_0874_msk_1.map
emd_0874_msk_1.map 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー




















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)