+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0742 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 277 K cryoEM structure of Sso-KARI in complex with Mg2+, NADH and CPD | |||||||||
 マップデータ マップデータ | Sso-KARI dodecameric enzyme in complex with Mg2 , NADH and CPD, and cryoEM sample was prepared at 277K. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Complex / ISOMERASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ketol-acid reductoisomerase (NADP+) / ketol-acid reductoisomerase activity / L-valine biosynthetic process / isoleucine biosynthetic process / isomerase activity / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |   Saccharolobus solfataricus (古細菌) Saccharolobus solfataricus (古細菌) | |||||||||
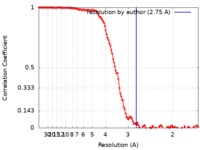
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.75 Å | |||||||||
 データ登録者 データ登録者 | Chen CY / Chang YC / Lin BL / Huang CH / Tsai MD | |||||||||
 引用 引用 |  ジャーナル: J Am Chem Soc / 年: 2019 ジャーナル: J Am Chem Soc / 年: 2019タイトル: Temperature-Resolved Cryo-EM Uncovers Structural Bases of Temperature-Dependent Enzyme Functions. 著者: Chin-Yu Chen / Yuan-Chih Chang / Bo-Lin Lin / Chun-Hsiang Huang / Ming-Daw Tsai /  要旨: Protein functions are temperature-dependent, but protein structures are usually solved at a single (often low) temperature because of limitations on the conditions of crystal growth or protein ...Protein functions are temperature-dependent, but protein structures are usually solved at a single (often low) temperature because of limitations on the conditions of crystal growth or protein vitrification. Here we demonstrate the feasibility of solving cryo-EM structures of proteins vitrified at high temperatures, solve 12 structures of an archaeal ketol-acid reductoisomerase (KARI) vitrified at 4-70 °C, and show that structures of both the Mg form (KARI:2Mg) and its ternary complex (KARI:2Mg:NADH:inhibitor) are temperature-dependent in correlation with the temperature dependence of enzyme activity. Furthermore, structural analyses led to dissection of the induced-fit mechanism into ligand-induced and temperature-induced effects and to capture of temperature-resolved intermediates of the temperature-induced conformational change. The results also suggest that it is preferable to solve cryo-EM structures of protein complexes at functional temperatures. These studies should greatly expand the landscapes of protein structure-function relationships and enhance the mechanistic analysis of enzymatic functions. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0742.map.gz emd_0742.map.gz | 154.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0742-v30.xml emd-0742-v30.xml emd-0742.xml emd-0742.xml | 10.4 KB 10.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0742_fsc.xml emd_0742_fsc.xml | 14.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0742.png emd_0742.png | 212 KB | ||
| Filedesc metadata |  emd-0742.cif.gz emd-0742.cif.gz | 5.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0742 http://ftp.pdbj.org/pub/emdb/structures/EMD-0742 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0742 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0742 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6kpaMC  0740C  0743C  0746C  0747C  0748C  0749C  0750C  0751C  0752C  0753C  0754C  6kouC  6kpeC  6kphC  6kpiC  6kpjC  6kpkC  6kq4C  6kq8C  6kqjC  6kqkC  6kqoC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0742.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0742.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sso-KARI dodecameric enzyme in complex with Mg2 , NADH and CPD, and cryoEM sample was prepared at 277K. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.84 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : KARI-Mg2+/NADH/CPD complex
| 全体 | 名称: KARI-Mg2+/NADH/CPD complex |
|---|---|
| 要素 |
|
-超分子 #1: KARI-Mg2+/NADH/CPD complex
| 超分子 | 名称: KARI-Mg2+/NADH/CPD complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Saccharolobus solfataricus (古細菌) Saccharolobus solfataricus (古細菌) |
-分子 #1: Ketol-acid reductoisomerase
| 分子 | 名称: Ketol-acid reductoisomerase / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharolobus solfataricus (古細菌) Saccharolobus solfataricus (古細菌) |
| 分子量 | 理論値: 37.229855 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDKTVLDANL DPLKGKTIGV IGYGNQGRVQ ATIMRENGLN VIVGNVKDKY YELAKKEGFE VYEIDEAVRR SDVALLLIPD EVMKEVYEK KIAPVLQGKK EFVLDFASGY NVAFGLIRPP KSVDTIMVAP RMVGEGIMDL HKQGKGYPVL LGVKQDASGK A WDYAKAIA ...文字列: MDKTVLDANL DPLKGKTIGV IGYGNQGRVQ ATIMRENGLN VIVGNVKDKY YELAKKEGFE VYEIDEAVRR SDVALLLIPD EVMKEVYEK KIAPVLQGKK EFVLDFASGY NVAFGLIRPP KSVDTIMVAP RMVGEGIMDL HKQGKGYPVL LGVKQDASGK A WDYAKAIA KGIGAIPGGI AVISSFEEEA LLDLMSEHTW VPILFGAIKA CYDIAVKEYG VSPEAALLEF YASGELAEIA RL IAEEGIF NQMVHHSTTS QYGTLTRMFK YYDVVRRIVE NEAKYIWDGS FAKEWSLEQQ AGYPVFYRLW ELATQSEMAK AEK ELYKLL GRKVKND UniProtKB: Ketol-acid reductoisomerase |
-分子 #2: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 2 / コピー数: 24 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #3: 1,4-DIHYDRONICOTINAMIDE ADENINE DINUCLEOTIDE
| 分子 | 名称: 1,4-DIHYDRONICOTINAMIDE ADENINE DINUCLEOTIDE / タイプ: ligand / ID: 3 / コピー数: 12 / 式: NAI |
|---|---|
| 分子量 | 理論値: 665.441 Da |
| Chemical component information |  ChemComp-NAI: |
-分子 #4: cyclopropane-1,1-dicarboxylic acid
| 分子 | 名称: cyclopropane-1,1-dicarboxylic acid / タイプ: ligand / ID: 4 / コピー数: 12 / 式: 9TY |
|---|---|
| 分子量 | 理論値: 130.099 Da |
| Chemical component information | 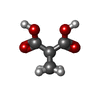 ChemComp-9TY: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 / 詳細: 20 mM Tris-Cl, pH 7.5, 50 mM NaCl and 5 mM MgCl2 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 平均電子線量: 52.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)