+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0529 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Purified ribosome - A complete data processing workflow for CryoET and subtomogram averaging | |||||||||
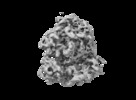
 マップデータ マップデータ | Averaged structure of purified ribosome from EMPIAR-10064 dataset, after subtilt refinement in EMAN2. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
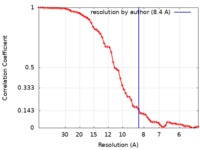
| 手法 | 電子線トモグラフィー法 / クライオ電子顕微鏡法 / 解像度: 8.4 Å | |||||||||
 データ登録者 データ登録者 | Chen M / Bell JM / Ludtke SJ | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Methods / 年: 2019 ジャーナル: Nat Methods / 年: 2019タイトル: A complete data processing workflow for cryo-ET and subtomogram averaging. 著者: Muyuan Chen / James M Bell / Xiaodong Shi / Stella Y Sun / Zhao Wang / Steven J Ludtke /   要旨: Electron cryotomography is currently the only method capable of visualizing cells in three dimensions at nanometer resolutions. While modern instruments produce massive amounts of tomography data ...Electron cryotomography is currently the only method capable of visualizing cells in three dimensions at nanometer resolutions. While modern instruments produce massive amounts of tomography data containing extremely rich structural information, data processing is very labor intensive and the results are often limited by the skills of the personnel rather than the data. We present an integrated workflow that covers the entire tomography data processing pipeline, from automated tilt series alignment to subnanometer resolution subtomogram averaging. Resolution enhancement is made possible through the use of per-particle per-tilt contrast transfer function correction and alignment. The workflow greatly reduces human bias, increases throughput and more closely approaches data-limited resolution for subtomogram averaging in both purified macromolecules and cells. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0529.map.gz emd_0529.map.gz | 4.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0529-v30.xml emd-0529-v30.xml emd-0529.xml emd-0529.xml | 10.6 KB 10.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0529_fsc.xml emd_0529_fsc.xml | 6.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0529.png emd_0529.png | 85.9 KB | ||
| その他 |  emd_0529_additional.map.gz emd_0529_additional.map.gz | 21 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0529 http://ftp.pdbj.org/pub/emdb/structures/EMD-0529 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0529 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0529 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0529_validation.pdf.gz emd_0529_validation.pdf.gz | 79 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0529_full_validation.pdf.gz emd_0529_full_validation.pdf.gz | 78.1 KB | 表示 | |
| XML形式データ |  emd_0529_validation.xml.gz emd_0529_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0529 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0529 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0529 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0529 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0529.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0529.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Averaged structure of purified ribosome from EMPIAR-10064 dataset, after subtilt refinement in EMAN2. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.62 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
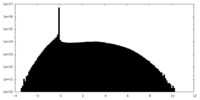
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Averaged structure after subtomogram refinement but before subtilt...
| ファイル | emd_0529_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Averaged structure after subtomogram refinement but before subtilt refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ribosome from EMPIAR-10064, MixedCTEM dataset
| 全体 | 名称: Ribosome from EMPIAR-10064, MixedCTEM dataset |
|---|---|
| 要素 |
|
-超分子 #1: Ribosome from EMPIAR-10064, MixedCTEM dataset
| 超分子 | 名称: Ribosome from EMPIAR-10064, MixedCTEM dataset / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線トモグラフィー法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE |
| 切片作成 | その他: NO SECTIONING |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)