+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 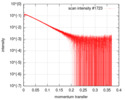 |
|---|---|
 試料 試料 | Truncated Apolipoprotein E4 (1-191) bound to 330 µg/mL heparin
|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報lipid transport involved in lipid storage / intermediate-density lipoprotein particle clearance / positive regulation of lipid transport across blood-brain barrier / regulation of cellular response to very-low-density lipoprotein particle stimulus / metal chelating activity / triglyceride-rich lipoprotein particle clearance / discoidal high-density lipoprotein particle / lipoprotein particle / negative regulation of triglyceride metabolic process / negative regulation of cholesterol biosynthetic process ...lipid transport involved in lipid storage / intermediate-density lipoprotein particle clearance / positive regulation of lipid transport across blood-brain barrier / regulation of cellular response to very-low-density lipoprotein particle stimulus / metal chelating activity / triglyceride-rich lipoprotein particle clearance / discoidal high-density lipoprotein particle / lipoprotein particle / negative regulation of triglyceride metabolic process / negative regulation of cholesterol biosynthetic process / regulation of amyloid-beta clearance / maintenance of location in cell / positive regulation of lipoprotein transport / Transcriptional regulation by the AP-2 (TFAP2) family of transcription factors / chylomicron remnant clearance / chylomicron remnant / intermediate-density lipoprotein particle / acylglycerol homeostasis / NMDA glutamate receptor clustering / very-low-density lipoprotein particle remodeling / phosphatidylcholine-sterol O-acyltransferase activator activity / response to caloric restriction / Chylomicron clearance / positive regulation of phospholipid efflux / very-low-density lipoprotein particle clearance / Chylomicron remodeling / lipid transporter activity / cellular response to lipoprotein particle stimulus / positive regulation of low-density lipoprotein particle receptor catabolic process / regulation of amyloid fibril formation / Chylomicron assembly / high-density lipoprotein particle clearance / chylomicron / phospholipid efflux / regulation of protein metabolic process / very-low-density lipoprotein particle receptor binding / high-density lipoprotein particle remodeling / lipoprotein catabolic process / AMPA glutamate receptor clustering / melanosome organization / positive regulation of cholesterol metabolic process / multivesicular body, internal vesicle / regulation of behavioral fear response / reverse cholesterol transport / positive regulation of amyloid-beta clearance / host-mediated activation of viral process / high-density lipoprotein particle assembly / low-density lipoprotein particle / lipoprotein biosynthetic process / cholesterol transfer activity / high-density lipoprotein particle / protein import / very-low-density lipoprotein particle / cholesterol catabolic process / low-density lipoprotein particle remodeling / heparan sulfate proteoglycan binding / amyloid precursor protein metabolic process / negative regulation of amyloid fibril formation / regulation of amyloid precursor protein catabolic process / positive regulation of membrane protein ectodomain proteolysis / regulation of Cdc42 protein signal transduction / synaptic transmission, cholinergic / HDL remodeling / negative regulation of endothelial cell migration / cholesterol efflux / regulation of cholesterol metabolic process / negative regulation of protein metabolic process / regulation of axon extension / triglyceride homeostasis / artery morphogenesis / Scavenging by Class A Receptors / triglyceride metabolic process / low-density lipoprotein particle receptor binding / positive regulation of amyloid fibril formation / regulation of innate immune response / virion assembly / positive regulation of dendritic spine development / negative regulation of endothelial cell proliferation / negative regulation of amyloid-beta formation / locomotory exploration behavior / antioxidant activity / response to dietary excess / lipoprotein particle binding / negative regulation of MAP kinase activity / negative regulation of blood vessel endothelial cell migration / positive regulation of endocytosis / negative regulation of long-term synaptic potentiation / negative regulation of platelet activation / positive regulation of dendritic spine maintenance / negative regulation of blood coagulation / positive regulation of cholesterol efflux / regulation of neuronal synaptic plasticity / long-term memory / negative regulation of protein secretion / fatty acid homeostasis / long-chain fatty acid transport / synaptic cleft / regulation of protein-containing complex assembly / intracellular transport / positive regulation of lipid biosynthetic process 類似検索 - 分子機能 |
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
 登録者 登録者 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-モデル
- 試料
試料
 試料 試料 | 名称: Truncated Apolipoprotein E4 (1-191) bound to 330 µg/mL heparin 試料濃度: 4 mg/ml / Entity id: 1633 / 1635 |
|---|---|
| バッファ | 名称: 20 mM HEPES, 300 mM NaCl, 1 mM TCEP / pH: 8 |
| 要素 #1633 | タイプ: other / 記述: Heparin / 分子量: 15 / 分子数: 1 配列: (C12H19NO20S3)n |
| 要素 #1635 | 名称: ApoE4 / タイプ: protein / 記述: Apolipoprotein E4 (1-191) / 分子量: 22.605 / 分子数: 1 / 由来: Homo sapiens / 参照: UniProt: P02649 配列: GPHMKVEQAV ETEPEPELRQ QTEWQSGQRW ELALGRFWDY LRWVQTLSEQ VQEELLSSQV TQELRALMDE TMKELKAYKS ELEEQLTPVA EETRARLSKE LQAAQARLGA DMEDVRGRLV QYRGEVQAML GQSTEELRVR LASHLRKLRK RLLRDADDLQ KRLAVYQAGA ...配列: GPHMKVEQAV ETEPEPELRQ QTEWQSGQRW ELALGRFWDY LRWVQTLSEQ VQEELLSSQV TQELRALMDE TMKELKAYKS ELEEQLTPVA EETRARLSKE LQAAQARLGA DMEDVRGRLV QYRGEVQAML GQSTEELRVR LASHLRKLRK RLLRDADDLQ KRLAVYQAGA REGAERGLSA IRERLGPLVE QGRVR |
-実験情報
| ビーム | 設備名称: Diamond Light Source B21 / 地域: Didcot / 国: UK  / 形状: 1 x 5 mm / 線源: X-ray synchrotron / 波長: 0.1 Å / スペクトロメータ・検出器間距離: 4.036 mm / 形状: 1 x 5 mm / 線源: X-ray synchrotron / 波長: 0.1 Å / スペクトロメータ・検出器間距離: 4.036 mm | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 2M | |||||||||||||||||||||
| スキャン | 測定日: 2018年5月3日 / 保管温度: 20 °C / セル温度: 20 °C / 照射時間: 1 sec. / フレーム数: 28 / 単位: 1/A /
| |||||||||||||||||||||
| 距離分布関数 P(R) |
| |||||||||||||||||||||
| 結果 | コメント: ApoE4 (1-191) + 330 µg/mL heparin. Molecular weight estimates are inaccurate due to heparin heterogeneity.
|
 ムービー
ムービー コントローラー
コントローラー



 SASDGU7
SASDGU7











