+ Open data
Open data
- Basic information
Basic information
| Entry | 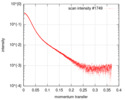 |
|---|---|
 Sample Sample | Apolipoprotein E4 (K143A K146A) mutant bound to 37 µM Suramin
|
| Function / homology |  Function and homology information Function and homology informationlipid transport involved in lipid storage / intermediate-density lipoprotein particle clearance / positive regulation of lipid transport across blood-brain barrier / regulation of cellular response to very-low-density lipoprotein particle stimulus / metal chelating activity / triglyceride-rich lipoprotein particle clearance / discoidal high-density lipoprotein particle / lipoprotein particle / negative regulation of triglyceride metabolic process / maintenance of location in cell ...lipid transport involved in lipid storage / intermediate-density lipoprotein particle clearance / positive regulation of lipid transport across blood-brain barrier / regulation of cellular response to very-low-density lipoprotein particle stimulus / metal chelating activity / triglyceride-rich lipoprotein particle clearance / discoidal high-density lipoprotein particle / lipoprotein particle / negative regulation of triglyceride metabolic process / maintenance of location in cell / regulation of amyloid-beta clearance / negative regulation of cholesterol biosynthetic process / positive regulation of lipoprotein transport / Transcriptional regulation by the AP-2 (TFAP2) family of transcription factors / chylomicron remnant clearance / chylomicron remnant / intermediate-density lipoprotein particle / acylglycerol homeostasis / NMDA glutamate receptor clustering / very-low-density lipoprotein particle remodeling / Chylomicron clearance / phosphatidylcholine-sterol O-acyltransferase activator activity / positive regulation of phospholipid efflux / Chylomicron remodeling / positive regulation of low-density lipoprotein particle receptor catabolic process / response to caloric restriction / cellular response to lipoprotein particle stimulus / very-low-density lipoprotein particle clearance / lipid transporter activity / Chylomicron assembly / high-density lipoprotein particle clearance / phospholipid efflux / chylomicron / regulation of protein metabolic process / very-low-density lipoprotein particle receptor binding / regulation of amyloid fibril formation / lipoprotein catabolic process / AMPA glutamate receptor clustering / high-density lipoprotein particle remodeling / melanosome organization / positive regulation of cholesterol metabolic process / multivesicular body, internal vesicle / regulation of behavioral fear response / reverse cholesterol transport / positive regulation of amyloid-beta clearance / high-density lipoprotein particle assembly / host-mediated activation of viral process / low-density lipoprotein particle / lipoprotein biosynthetic process / cholesterol transfer activity / protein import / high-density lipoprotein particle / very-low-density lipoprotein particle / cholesterol catabolic process / heparan sulfate proteoglycan binding / low-density lipoprotein particle remodeling / amyloid precursor protein metabolic process / negative regulation of amyloid fibril formation / regulation of Cdc42 protein signal transduction / positive regulation of membrane protein ectodomain proteolysis / synaptic transmission, cholinergic / regulation of amyloid precursor protein catabolic process / HDL remodeling / negative regulation of endothelial cell migration / cholesterol efflux / regulation of cholesterol metabolic process / artery morphogenesis / regulation of axon extension / negative regulation of protein metabolic process / Scavenging by Class A Receptors / triglyceride homeostasis / triglyceride metabolic process / low-density lipoprotein particle receptor binding / positive regulation of amyloid fibril formation / regulation of innate immune response / virion assembly / negative regulation of endothelial cell proliferation / positive regulation of dendritic spine development / response to dietary excess / antioxidant activity / negative regulation of MAP kinase activity / negative regulation of amyloid-beta formation / lipoprotein particle binding / locomotory exploration behavior / negative regulation of long-term synaptic potentiation / negative regulation of blood vessel endothelial cell migration / positive regulation of endocytosis / negative regulation of platelet activation / negative regulation of blood coagulation / positive regulation of dendritic spine maintenance / regulation of neuronal synaptic plasticity / positive regulation of cholesterol efflux / negative regulation of protein secretion / fatty acid homeostasis / long-term memory / regulation of protein-containing complex assembly / synaptic cleft / long-chain fatty acid transport / intracellular transport / positive regulation of lipid biosynthetic process Similarity search - Function |
| Biological species |  Homo sapiens (human) Homo sapiens (human) |
 Contact author Contact author |
|
- Structure visualization
Structure visualization
- Downloads & links
Downloads & links
-Data source
| SASBDB page |  SASDGN8 SASDGN8 |
|---|
-Related structure data
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
|---|
- External links
External links
| Related items in Molecule of the Month |
|---|
-Models
- Sample
Sample
 Sample Sample | Name: Apolipoprotein E4 (K143A K146A) mutant bound to 37 µM Suramin Specimen concentration: 4 mg/ml / Entity id: 1634 / 1682 |
|---|---|
| Buffer | Name: 20 mM HEPES, 300 mM NaCl, 1 mM TCEP / pH: 8 |
| Entity #1634 | Name: ApoE4 (K143A K146A) / Type: protein / Description: Apolipoprotein E4 (K143A K146A) mutant / Formula weight: 34.598 / Num. of mol.: 4 / Source: Homo sapiens / References: UniProt: P02649 Sequence: GPHMKVEQAV ETEPEPELRQ QTEWQSGQRW ELALGRFWDY LRWVQTLSEQ VQEELLSSQV TQELRALMDE TMKELKAYKS ELEEQLTPVA EETRARLSKE LQAAQARLGA DMEDVRGRLV QYRGEVQAML GQSTEELRVR LASHLRALRA RLLRDADDLQ KRLAVYQAGA ...Sequence: GPHMKVEQAV ETEPEPELRQ QTEWQSGQRW ELALGRFWDY LRWVQTLSEQ VQEELLSSQV TQELRALMDE TMKELKAYKS ELEEQLTPVA EETRARLSKE LQAAQARLGA DMEDVRGRLV QYRGEVQAML GQSTEELRVR LASHLRALRA RLLRDADDLQ KRLAVYQAGA REGAERGLSA IRERLGPLVE QGRVRAATVG SLAGQPLQER AQAWGERLRA RMEEMGSRTR DRLDEVKEQV AEVRAKLEEQ AQQIRLQAEA FQARLKSWFE PLVEDMQRQW AGLVEKVQAA VGTSAAPVPS DNH |
| Entity #1682 | Type: other / Description: Suramin / Formula weight: 1.297 / Num. of mol.: 1 Sequence: C51H40N6O2 3S6 |
-Experimental information
| Beam | Instrument name: Diamond Light Source B21 / City: Didcot / 国: UK  / Shape: 1 x 5 mm / Type of source: X-ray synchrotron / Wavelength: 0.1 Å / Dist. spec. to detc.: 4.036 mm / Shape: 1 x 5 mm / Type of source: X-ray synchrotron / Wavelength: 0.1 Å / Dist. spec. to detc.: 4.036 mm | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Detector | Name: Pilatus 2M | |||||||||||||||||||||
| Scan | Measurement date: Jul 21, 2018 / Storage temperature: 20 °C / Cell temperature: 20 °C / Exposure time: 1 sec. / Number of frames: 28 / Unit: 1/A /
| |||||||||||||||||||||
| Distance distribution function P(R) |
| |||||||||||||||||||||
| Result |
|
 Movie
Movie Controller
Controller















