+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: SASBDB / ID: SASDG47 |
|---|---|
 試料 試料 | 2:4 heterohexamer of pUL7 and pUL51 from herpes simplex virus 1
|
| 機能・相同性 | Herpesvirus UL51 / Herpesvirus UL51 protein / Herpesvirus UL7-like / Herpesvirus UL7 like / viral tegument / host cell Golgi apparatus / UL7 / UL51 機能・相同性情報 機能・相同性情報 |
| 生物種 |   Human alphaherpesvirus 1 strain KOS (ヘルペスウイルス) Human alphaherpesvirus 1 strain KOS (ヘルペスウイルス) |
 登録者 登録者 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-モデル
| モデル #3903 | 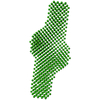 タイプ: dummy / ソフトウェア: (SUPCOMB 23 (r10552)) / ダミー原子の半径: 3.75 A / 対称性: P2 コメント: Refined DAMMIN model of the HSV-1 pUL7:pUL51 2:4 heterohexamer カイ2乗値: 0.910 / P-value: 0.805657 |
|---|---|
| モデル #3904 |  タイプ: dummy / ソフトウェア: (2.3i) / ダミー原子の半径: 1.90 A / 対称性: P2 コメント: Representative GASBOR model of the HSV-1 pUL7:pUL51 2:4 heterohexamer カイ2乗値: 1.00 / P-value: 0.363345 |
- 試料
試料
 試料 試料 | 名称: 2:4 heterohexamer of pUL7 and pUL51 from herpes simplex virus 1 試料濃度: 8 mg/ml / Entity id: 1941 / 1942 |
|---|---|
| バッファ | 名称: 20 mM HEPES, 200 mM NaCl, 3% (v/v) glycerol, 1 mM DTT pH: 7.5 |
| 要素 #1941 | 名称: pUL7 / タイプ: protein / 記述: Tegument protein UL7 / 分子量: 33.907 / 分子数: 2 / 由来: Human alphaherpesvirus 1 strain KOS / 参照: UniProt: A0A110B4Q7 配列: MAAATADDEG SAATILKQAI AGDRSLVEAA EAISQQTLLR LACEVRQVGD RQPRFTATSI ARVDVAPGCR LRFVLDGSPE DAYVTSEDYF KRCCGQSSYR GFAVAVLTAN EDHVHSLAVP PLVLLHRFSL FNPRDLLDFE LACLLMYLEN CPRSHATPST FAKVLAWLGV ...配列: MAAATADDEG SAATILKQAI AGDRSLVEAA EAISQQTLLR LACEVRQVGD RQPRFTATSI ARVDVAPGCR LRFVLDGSPE DAYVTSEDYF KRCCGQSSYR GFAVAVLTAN EDHVHSLAVP PLVLLHRFSL FNPRDLLDFE LACLLMYLEN CPRSHATPST FAKVLAWLGV AGRRTSPFER VRCLFLRSCH WVLNTLMFMV HVKPFDDEFV LPHWYMARYL LANNPPPVLS ALFCATPTSS SFRLPGPPPR SDCVAYNPAG IMGSCWASEE VRAPLVYWWL SETPKRQTSS LFYQFCGSLE VLFQ |
| 要素 #1942 | 名称: pUL51 / タイプ: protein / 記述: Tegument protein UL51 / 分子量: 25.448 / 分子数: 4 / 由来: Human alphaherpesvirus 1 strain KOS / 参照: UniProt: D3YPL0 配列: MASLLGAISG WGARPEEQYE MIRAAVPPSE AEPRLQEALV VVNALLPAPI TLDDALGSLD DTRRLVKARA LARTYHACMV NLERLARHHP GLEAPTIDGA VAAHQDKMRR LADTCMATIL QMYMSVGAAD KSADVLVSQA IRSMAESDVV MEDVAIAERA LGLSAFGVAG ...配列: MASLLGAISG WGARPEEQYE MIRAAVPPSE AEPRLQEALV VVNALLPAPI TLDDALGSLD DTRRLVKARA LARTYHACMV NLERLARHHP GLEAPTIDGA VAAHQDKMRR LADTCMATIL QMYMSVGAAD KSADVLVSQA IRSMAESDVV MEDVAIAERA LGLSAFGVAG GTRSGGLGVT EAPSLGHPHT PPPEVTLAPA ARNGDALPDP KPESCPRVSV PRPTASPTAP RPGPSRAAPC VLGQ |
-実験情報
| ビーム | 設備名称: PETRA III EMBL P12 / 地域: Hamburg / 国: Germany  / 線源: X-ray synchrotron / 波長: 0.124 Å / スペクトロメータ・検出器間距離: 3 mm / 線源: X-ray synchrotron / 波長: 0.124 Å / スペクトロメータ・検出器間距離: 3 mm | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 6M | ||||||||||||||||||||||||||||||
| スキャン | 測定日: 2019年5月16日 / セル温度: 20.2 °C / 照射時間: 0.995 sec. / フレーム数: 48 / 単位: 1/nm /
| ||||||||||||||||||||||||||||||
| 距離分布関数 P(R) |
| ||||||||||||||||||||||||||||||
| 結果 | コメント: The quoted experimental molecular mass, that corresponds to a 2:4 pUL7:pUL51 heterohexamer, was determined from SEC-MALLS-RI measurements performed in parallel to the SEC-SAXS. The SEC- ...コメント: The quoted experimental molecular mass, that corresponds to a 2:4 pUL7:pUL51 heterohexamer, was determined from SEC-MALLS-RI measurements performed in parallel to the SEC-SAXS. The SEC-SAXS data, Rg correlations through the dimer elution peak as well as the the SEC-MALLS-RI data are made available in the full-entry zip archive. The GASBORMX model displayed in this entry represents a 100 % volume fraction of the heterohexamer in solution that has been separated from a 1:2 pUL7:pUL51 heterotrimer (refer to SASBDB entry SASDG57).
|
 ムービー
ムービー コントローラー
コントローラー



 SASDG47
SASDG47