| ビーム | 設備名称: Cornell High Energy Synchrotron Source (CHESS) G1
地域: Ithaca, NY / 国: USA  / 線源: X-ray synchrotron / 波長: 0.124612 Å / スペクトロメータ・検出器間距離: 1.52109 mm / 線源: X-ray synchrotron / 波長: 0.124612 Å / スペクトロメータ・検出器間距離: 1.52109 mm |
|---|
| 検出器 | 名称: Pilatus 200K / タイプ: Pilatus / Pixsize x: 172 mm |
|---|
| スキャン | タイトル: Herpes simplex virus 1 tegument protein UL11 / 測定日: 2017年6月4日 / 保管温度: 4 °C / セル温度: 25 °C / 照射時間: 2 sec. / フレーム数: 497 / 単位: 1/A / |
|---|
| 距離分布関数 P(R) | ソフトウェア P(R): GNOM 5.0 / ポイント数: 480 / | Min | Max |
|---|
| Q | 0.007412 | 0.280191 |
|---|
| P(R) point | 1 | 480 |
|---|
| R | 0 | 120 |
|---|
|
|---|
| 結果 | カーブのタイプ: sec / | 実験値 | Porod |
|---|
| 分子量 | 15.8 kDa | 18 kDa |
|---|
| 体積 | - | 29.5 nm3 |
|---|
| P(R) | Guinier | Guinier error |
|---|
| 前方散乱 I0 | 0.004866 | 0.00477 | 4.1E-5 |
|---|
| 慣性半径, Rg | 2.63 nm | 2.429 nm | 0.2 |
|---|
| Min | Max |
|---|
| D | - | 12 |
|---|
| Guinier point | 9 | 81 |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報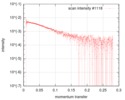
 試料
試料 機能・相同性情報
機能・相同性情報 Human herpesvirus 1 (strain 17) (ヘルペスウイルス)
Human herpesvirus 1 (strain 17) (ヘルペスウイルス) 登録者
登録者 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク 試料
試料 試料
試料 / 線源: X-ray synchrotron / 波長: 0.124612 Å / スペクトロメータ・検出器間距離: 1.52109 mm
/ 線源: X-ray synchrotron / 波長: 0.124612 Å / スペクトロメータ・検出器間距離: 1.52109 mm ムービー
ムービー コントローラー
コントローラー



 SASDEX4
SASDEX4