+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  |
|---|---|
 試料 試料 | Cell wall synthesis protein Wag31: Polymer tbWag31 T73A
|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報peptidoglycan-based cell wall biogenesis / regulation of growth rate / cell pole / peptidoglycan-based cell wall / regulation of cell shape / protein stabilization / cell division / plasma membrane / cytoplasm 類似検索 - 分子機能 |
| 生物種 |  Mycobacterium tuberculosis (strain ATCC 25618 / H37Rv) (結核菌) Mycobacterium tuberculosis (strain ATCC 25618 / H37Rv) (結核菌) |
 引用 引用 |  日付: 2018 Aug 18 日付: 2018 Aug 18タイトル: Higher order assembling of the mycobacterial essential polar growth factor DivIVA/Wag31 著者: Choukate K / Gupta A / Basu B / Virk K / Ganguli M |
 登録者 登録者 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-モデル
- 試料
試料
 試料 試料 | 名称: Cell wall synthesis protein Wag31: Polymer tbWag31 T73A 試料濃度: 2.7 mg/ml |
|---|---|
| バッファ | 名称: 50mM Tris pH7.5, 300mM NaCl, 10% Glycerol, 1mM EDTA (ethylene diamine tetra acetic acid), 5mM β-mercaptoethanol (BME) pH: 7.5 |
| 要素 #1213 | タイプ: protein / 記述: Cell wall synthesis protein Wag31 / 分子量: 29.922 由来: Mycobacterium tuberculosis (strain ATCC 25618 / H37Rv) 参照: UniProt: P9WMU1 配列: MHHHHHHENL YFQGPLTPAD VHNVAFSKPP IGKRGYNEDE VDAFLDLVEN ELTRLIEENS DLRQRINELD QELAAGGGAG VTPQAAQAIP AYEPEPGKPA PAAVSAGMNE EQALKAARVL SLAQDTADRL TNTAKAESDK MLADARANAE QILGEARHTA DATVAEARQR ...配列: MHHHHHHENL YFQGPLTPAD VHNVAFSKPP IGKRGYNEDE VDAFLDLVEN ELTRLIEENS DLRQRINELD QELAAGGGAG VTPQAAQAIP AYEPEPGKPA PAAVSAGMNE EQALKAARVL SLAQDTADRL TNTAKAESDK MLADARANAE QILGEARHTA DATVAEARQR ADAMLADAQS RSEAQLRQAQ EKADALQADA ERKHSEIMGT INQQRAVLEG RLEQLRTFER EYRTRLKTYL ESQLEELGQR GSAAPVDSNA DAGGFDQFNR GKN |
-実験情報
| ビーム | 設備名称: ESRF BM29 / 地域: Grenoble / 国: France  / 線源: X-ray synchrotron / 波長: 0.1 Å / スペクトロメータ・検出器間距離: 2.87 mm / 線源: X-ray synchrotron / 波長: 0.1 Å / スペクトロメータ・検出器間距離: 2.87 mm | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 1M / タイプ: Dectris / Pixsize x: 172 mm | ||||||||||||||||||||||||
| スキャン | 測定日: 2017年12月15日 / 保管温度: 4 °C / セル温度: 4 °C / 照射時間: 1 sec. / フレーム数: 10 / 単位: 1/nm /
| ||||||||||||||||||||||||
| 距離分布関数 P(R) |
| ||||||||||||||||||||||||
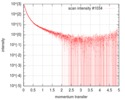
| 結果 | コメント: The cell wall synthesis protein Wag31: Polymer tbWag31 T73A forms a large super-macromolecular assembly (supported by atomic force microscopy, AFM) and as a result there is a very ...コメント: The cell wall synthesis protein Wag31: Polymer tbWag31 T73A forms a large super-macromolecular assembly (supported by atomic force microscopy, AFM) and as a result there is a very limited angular range to estimate the radius of gyration, Rg, using the Guinier approximation. In addition, the quoted monomer molecular weight does not apply to this sample. The probable distribution of real-space scattering pair vector lengths (p(r) vs r profile or PDDF) represents the cross-sectional pair distribution function for rod-like particle (option 4 in GNOM) and the subsequent data validation tools for this entry are based on the corresponding cross section parameters (Rg of cross section and Dmax of cross section). The I(s) is recorded in a relative scale in kDa units assuming a 1 mg/ml sample concentration. However the experimental concentration was found to be 2.7 mg/ml (assessed using a Bradford assay).
|
 ムービー
ムービー コントローラー
コントローラー



 SASDEC2
SASDEC2