+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: SASBDB / ID: SASDDY4 |
|---|---|
 試料 試料 | Protein sex-lethal mutant with 10GS-linker
|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報sex determination, primary response to X:A ratio / germarium-derived cystoblast division / epithelium regeneration / somatic sex determination / female germ-line sex determination / oocyte differentiation / female sex determination / imaginal disc growth / sex determination / regulation of stem cell division ...sex determination, primary response to X:A ratio / germarium-derived cystoblast division / epithelium regeneration / somatic sex determination / female germ-line sex determination / oocyte differentiation / female sex determination / imaginal disc growth / sex determination / regulation of stem cell division / poly-pyrimidine tract binding / negative regulation of RNA export from nucleus / sex-chromosome dosage compensation / sex differentiation / alternative mRNA splicing, via spliceosome / poly(A) binding / pre-mRNA binding / regulation of mRNA splicing, via spliceosome / positive regulation of smoothened signaling pathway / reciprocal meiotic recombination / poly(U) RNA binding / oogenesis / regulation of alternative mRNA splicing, via spliceosome / negative regulation of mRNA splicing, via spliceosome / negative regulation of translational initiation / mRNA regulatory element binding translation repressor activity / positive regulation of RNA splicing / mRNA 3'-UTR binding / mRNA 5'-UTR binding / negative regulation of translation / protein stabilization / ribonucleoprotein complex / mRNA binding / protein-containing complex / RNA binding / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 |
| 生物種 |  |
 引用 引用 |  ジャーナル: ACS Comb Sci / 年: 2018 ジャーナル: ACS Comb Sci / 年: 2018タイトル: A General Small-Angle X-ray Scattering-Based Screening Protocol Validated for Protein-RNA Interactions. 著者: Po-Chia Chen / Pawel Masiewicz / Vladimir Rybin / Dmitri Svergun / Janosch Hennig /  要旨: We present a screening protocol utilizing small-angle X-ray scattering (SAXS) to obtain structural information on biomolecular interactions independent of prior knowledge, so as to complement ...We present a screening protocol utilizing small-angle X-ray scattering (SAXS) to obtain structural information on biomolecular interactions independent of prior knowledge, so as to complement affinity-based screening and provide leads for further exploration. This protocol categorizes ligand titrations by computing pairwise agreement between curves, and separately estimates affinities by quantifying complex formation as a departure from the linear sum properties of solution SAXS. The protocol is validated by sparse sequence search around the native poly uridine RNA motifs of the two-RRM domain Sex-lethal protein (Sxl). The screening of 35 RNA motifs between 4 to 10 nucleotides reveals a strong variation of resulting complexes, revealed to be preference-switching between 1:1 and 2:2 binding stoichiometries upon addition of structural modeling. Validation of select sequences in isothermal calorimetry and NMR titration retrieves domain-specific roles and function of a guanine anchor. These findings reinforce the suitability of SAXS as a complement in lead identification. |
 登録者 登録者 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-モデル
| モデル #1866 |  タイプ: mix / ソフトウェア: (2.1) / ダミー原子の半径: 1.90 A / カイ2乗値: 0.679 / P-value: 0.675072  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
|---|---|
| モデル #1867 |  タイプ: mix / ソフトウェア: (2.1) / ダミー原子の半径: 1.90 A / カイ2乗値: 0.679 / P-value: 0.675072  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
| モデル #1868 |  タイプ: mix / ソフトウェア: (2.1) / ダミー原子の半径: 1.90 A / カイ2乗値: 0.679 / P-value: 0.675072  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
| モデル #1869 |  タイプ: mix / ソフトウェア: (2.1) / ダミー原子の半径: 1.90 A / カイ2乗値: 0.679 / P-value: 0.675072  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
| モデル #1870 |  タイプ: mix / ソフトウェア: (2.1) / ダミー原子の半径: 1.90 A / カイ2乗値: 0.679 / P-value: 0.675072  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
- 試料
試料
 試料 試料 | 名称: Protein sex-lethal mutant with 10GS-linker / 試料濃度: 1 mg/ml |
|---|---|
| バッファ | 名称: 10 mM KP, 50 mM NaCl, 10 mM DTT / pH: 6 |
| 要素 #1006 | 名称: Sxl10GS / タイプ: protein / 記述: Protein sex-lethal mutant / 分子量: 20.432 / 分子数: 1 / 由来: Drosophila melanogaster / 参照: UniProt: P19339 配列: GAMASNTNLI VNYLPQDMTD RELYALFRAI GPINTCRIMR DYKTGYSFGY AFVDFTSEMD SQRAIKVLNG ITVRNKRLKV SYARPGGGSG SGGGGSGESI KDTNLYVTNL PRTITDDQLD TIFGKYGSIV QKNILRDKLT GRPRGVAFVR YNKREEAQEA ISALNNVIPE GGSQPLSVRL AEEHGK |
-実験情報
| ビーム | 設備名称: ESRF BM29 / 地域: Grenoble / 国: France  / 線源: X-ray synchrotron / 波長: 0.09919 Å / スペクトロメータ・検出器間距離: 2.867 mm / 線源: X-ray synchrotron / 波長: 0.09919 Å / スペクトロメータ・検出器間距離: 2.867 mm | |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 1M | |||||||||||||||||||||||||||||||||
| スキャン |
| |||||||||||||||||||||||||||||||||
| 距離分布関数 P(R) |
| |||||||||||||||||||||||||||||||||
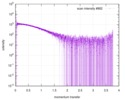
| 結果 |
|
 ムービー
ムービー コントローラー
コントローラー


 SASDDY4
SASDDY4