+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: SASBDB / ID: SASDBL2 |
|---|---|
 試料 試料 | MBP-PICK1 fusion
|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報membrane curvature sensor activity / glial cell development / neuronal ion channel clustering / Arp2/3 complex binding / cellular response to decreased oxygen levels / Trafficking of GluR2-containing AMPA receptors / regulation of Arp2/3 complex-mediated actin nucleation / negative regulation of Arp2/3 complex-mediated actin nucleation / monoamine transport / dendritic spine organization ...membrane curvature sensor activity / glial cell development / neuronal ion channel clustering / Arp2/3 complex binding / cellular response to decreased oxygen levels / Trafficking of GluR2-containing AMPA receptors / regulation of Arp2/3 complex-mediated actin nucleation / negative regulation of Arp2/3 complex-mediated actin nucleation / monoamine transport / dendritic spine organization / long-term synaptic depression / dendritic spine maintenance / receptor clustering / positive regulation of receptor internalization / cellular response to glucose starvation / regulation of insulin secretion / protein kinase C binding / trans-Golgi network membrane / Cell surface interactions at the vascular wall / intracellular protein transport / G protein-coupled receptor binding / phospholipid binding / endocytic vesicle membrane / actin filament binding / synaptic vesicle / presynaptic membrane / cytoskeleton / neuron projection / protein phosphorylation / postsynaptic density / protein domain specific binding / signaling receptor binding / synapse / perinuclear region of cytoplasm / Golgi apparatus / metal ion binding / identical protein binding / plasma membrane / cytosol / cytoplasm 類似検索 - 分子機能 |
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
 引用 引用 |  ジャーナル: Mol Biol Cell / 年: 2015 ジャーナル: Mol Biol Cell / 年: 2015タイトル: PICK1 is implicated in organelle motility in an Arp2/3 complex-independent manner. 著者: Yadaiah Madasu / Changsong Yang / Malgorzata Boczkowska / Kelley A Bethoney / Adam Zwolak / Grzegorz Rebowski / Tatyana Svitkina / Roberto Dominguez /  要旨: PICK1 is a modular scaffold implicated in synaptic receptor trafficking. It features a PDZ domain, a BAR domain, and an acidic C-terminal tail (ACT). Analysis by small- angle x-ray scattering ...PICK1 is a modular scaffold implicated in synaptic receptor trafficking. It features a PDZ domain, a BAR domain, and an acidic C-terminal tail (ACT). Analysis by small- angle x-ray scattering suggests a structural model that places the receptor-binding site of the PDZ domain and membrane-binding surfaces of the BAR and PDZ domains adjacent to each other on the concave side of the banana-shaped PICK1 dimer. In the model, the ACT of one subunit of the dimer interacts with the PDZ and BAR domains of the other subunit, possibly accounting for autoinhibition. Consistently, full-length PICK1 shows diffuse cytoplasmic localization, but it clusters on vesicle-like structures that colocalize with the trans-Golgi network marker TGN38 upon deletion of either the ACT or PDZ domain. This localization is driven by the BAR domain. Live-cell imaging further reveals that PICK1-associated vesicles undergo fast, nondirectional motility in an F-actin-dependent manner, but deleting the ACT dramatically reduces vesicle speed. Thus the ACT links PICK1-associated vesicles to a motility factor, likely myosin, but, contrary to previous reports, PICK1 neither binds nor inhibits Arp2/3 complex. |
 登録者 登録者 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-モデル
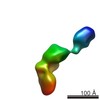
| モデル #402 |  タイプ: dummy / ソフトウェア: crysol / ダミー原子の半径: 1.90 A / カイ2乗値: 1.1881  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
|---|
- 試料
試料
 試料 試料 | 名称: MBP-PICK1 fusion / 試料濃度: 0.46-7.50 |
|---|---|
| バッファ | 名称: 50 mM Tris / pH: 7.5 / 組成: 300 mM NaCl, 1 mM maltose, 1 mM EGTA, 2 mM DTT |
| 要素 #266 | 名称: MBP-PICK1 / タイプ: protein 記述: Maltose Binding Protein fused to Protein Interacting with C kinase 1 分子量: 87.089 / 分子数: 2 / 由来: Homo sapiens / 参照: UniProt: Q9NRD5 配列: MKIEEGKLVI WINGDKGYNG LAEVGKKFEK DTGIKVTVEH PDKLEEKFPQ VAATGDGPDI IFWAHDRFGG YAQSGLLAEI TPDKAFQDKL YPFTWDAVRY NGKLIAYPIA VEALSLIYNK DLLPNPPKTW EEIPALDKEL KAKGKSALMF NLQEPYFTWP LIAADGGYAF ...配列: MKIEEGKLVI WINGDKGYNG LAEVGKKFEK DTGIKVTVEH PDKLEEKFPQ VAATGDGPDI IFWAHDRFGG YAQSGLLAEI TPDKAFQDKL YPFTWDAVRY NGKLIAYPIA VEALSLIYNK DLLPNPPKTW EEIPALDKEL KAKGKSALMF NLQEPYFTWP LIAADGGYAF KYENGKYDIK DVGVDNAGAK AGLTFLVDLI KNKHMNADTD YSIAEAAFNK GETAMTINGP WAWSNIDTSK VNYGVTVLPT FKGQPSKPFV GVLSAGINAA SPNKELAKEF LENYLLTDEG LEAVNKDKPL GAVALKSYEE ELAKDPRIAA TMENAQKGEI MPNIPQMSAF WYAVRTAVIN AASGRQTVDA ALAAAQTNAA AMFADLDYDI EEDKLGIPTV PGKVTLQKDA QNLIGISIGG GAQYCPCLYI VQVFDNTPAA LDGTVAAGDE ITGVNGRSIK GKTKVEVAKM IQEVKGEVTI HYNKLQADPK QGMSLDIVLK KVKHRLVENM SSGTADALGL SRAILCNDGL VKRLEELERT AELYKGMTEH TKNLLRAFYE LSQTHRAFGD VFSVIGVREP QPAASEAFVK FADAHRSIEK FGIRLLKTIK PMLTDLNTYL NKAIPDTRLT IKKYLDVKFE YLSYCLKVKE MDDEEYSCIA LGEPLYRVST GNYEYRLILR CRQEARARFS QMRKDVLEKM ELLDQKHVQD IVFQLQRLVS TMSKYYNDCY AVLRDADVFP IEVDLAHTTL AYGLNQEEFT DGEEEEEEED TAAGEPSRDT RGAAGPLDKG GSWCDS |
-実験情報
| ビーム | 設備名称: Cornell High Energy Synchrotron Source (CHESS) G1 地域: Ithaca, NY / 国: USA  / 線源: X-ray synchrotron / 線源: X-ray synchrotron | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Finger Lakes CCD / タイプ: CCD | ||||||||||||||||||
| スキャン |
| ||||||||||||||||||
| 距離分布関数 P(R) |
| ||||||||||||||||||
| 結果 | コメント: This dataset was collected at a concentration of 3.75 mg/ml.
|
 ムービー
ムービー コントローラー
コントローラー


 SASDBL2
SASDBL2