+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 9h8t | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the atovaquone-inhibited Complex III from the Chlorocebus sabaeus respirasome | |||||||||||||||||||||||||||
 要素 要素 |
| |||||||||||||||||||||||||||
 キーワード キーワード | ELECTRON TRANSPORT / complex III / respirasome / mitochondria / green african monkey / atovaquone | |||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報subthalamus development / pons development / cerebellar Purkinje cell layer development / mitochondrial respiratory chain complex III assembly / pyramidal neuron development / thalamus development / respiratory chain complex III / quinol-cytochrome-c reductase / quinol-cytochrome-c reductase activity / mitochondrial electron transport, ubiquinol to cytochrome c ...subthalamus development / pons development / cerebellar Purkinje cell layer development / mitochondrial respiratory chain complex III assembly / pyramidal neuron development / thalamus development / respiratory chain complex III / quinol-cytochrome-c reductase / quinol-cytochrome-c reductase activity / mitochondrial electron transport, ubiquinol to cytochrome c / hypothalamus development / midbrain development / hippocampus development / respiratory electron transport chain / metalloendopeptidase activity / 2 iron, 2 sulfur cluster binding / electron transfer activity / mitochondrial inner membrane / heme binding / proteolysis / nucleoplasm / metal ion binding 類似検索 - 分子機能 | |||||||||||||||||||||||||||
| 生物種 |  Chlorocebus sabaeus (オナガザル) Chlorocebus sabaeus (オナガザル) | |||||||||||||||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.67 Å | |||||||||||||||||||||||||||
 データ登録者 データ登録者 | Maclean, A. / Muhleip, A. | |||||||||||||||||||||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2025 ジャーナル: Nat Struct Mol Biol / 年: 2025タイトル: Structure, assembly and inhibition of the Toxoplasma gondii respiratory chain supercomplex. 著者: Andrew E MacLean / Shikha Shikha / Mariana Ferreira Silva / Max J Gramelspacher / Aaron Nilsen / Katherine M Liebman / Sovitj Pou / Rolf W Winter / Amit Meir / Michael K Riscoe / J Stone ...著者: Andrew E MacLean / Shikha Shikha / Mariana Ferreira Silva / Max J Gramelspacher / Aaron Nilsen / Katherine M Liebman / Sovitj Pou / Rolf W Winter / Amit Meir / Michael K Riscoe / J Stone Doggett / Lilach Sheiner / Alexander Mühleip /    要旨: The apicomplexan mitochondrial electron transport chain is essential for parasite survival and displays a divergent subunit composition. Here we report cryo-electron microscopy structures of an ...The apicomplexan mitochondrial electron transport chain is essential for parasite survival and displays a divergent subunit composition. Here we report cryo-electron microscopy structures of an apicomplexan III-IV supercomplex and of the drug target complex III. The supercomplex structure reveals how clade-specific subunits form an apicomplexan-conserved III-IV interface with a unique, kinked architecture, suggesting that supercomplexes evolved independently in different eukaryotic lineages. A knockout resulting in supercomplex disassembly challenges the proposed role of III-IV in electron transfer efficiency as suggested for mammals. Nevertheless, knockout analysis indicates that III-IV is critical for parasite fitness. The complexes from the model parasite Toxoplasma gondii were inhibited with the antimalarial atovaquone, revealing interactions underpinning species specificity. They were also inhibited with endochin-like quinolone (ELQ)-300, an inhibitor in late-stage preclinical development. Notably, in the apicomplexan binding site, ELQ-300 is flipped compared with related compounds in the mammalian enzyme. On the basis of the binding modes and parasite-specific interactions discovered, we designed more potent ELQs with subnanomolar activity against T. gondii. Our findings reveal critical evolutionary differences in the role of supercomplexes in mitochondrial biology and provide insight into cytochrome b inhibition, informing future drug discovery. | |||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  9h8t.cif.gz 9h8t.cif.gz | 1.5 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb9h8t.ent.gz pdb9h8t.ent.gz | 1.3 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  9h8t.json.gz 9h8t.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  9h8t_validation.pdf.gz 9h8t_validation.pdf.gz | 2.6 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  9h8t_full_validation.pdf.gz 9h8t_full_validation.pdf.gz | 2.7 MB | 表示 | |
| XML形式データ |  9h8t_validation.xml.gz 9h8t_validation.xml.gz | 125.5 KB | 表示 | |
| CIF形式データ |  9h8t_validation.cif.gz 9h8t_validation.cif.gz | 193.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/h8/9h8t https://data.pdbj.org/pub/pdb/validation_reports/h8/9h8t ftp://data.pdbj.org/pub/pdb/validation_reports/h8/9h8t ftp://data.pdbj.org/pub/pdb/validation_reports/h8/9h8t | HTTPS FTP |
-関連構造データ
| 関連構造データ |  51939MC  9g9tC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 4種, 8分子 DdEeJjHh
| #1: タンパク質 | 分子量: 35268.703 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)  Chlorocebus sabaeus (オナガザル) / 参照: UniProt: A0A0D9R8S7 Chlorocebus sabaeus (オナガザル) / 参照: UniProt: A0A0D9R8S7#5: タンパク質 | 分子量: 7320.473 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)  Chlorocebus sabaeus (オナガザル) / 参照: UniProt: A0A0D9RB67 Chlorocebus sabaeus (オナガザル) / 参照: UniProt: A0A0D9RB67#8: タンパク質 | 分子量: 43070.770 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)  Chlorocebus sabaeus (オナガザル) / 参照: UniProt: Q1AL63 Chlorocebus sabaeus (オナガザル) / 参照: UniProt: Q1AL63#10: タンパク質 | 分子量: 6623.710 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)  Chlorocebus sabaeus (オナガザル) Chlorocebus sabaeus (オナガザル)発現宿主:  Chlorocebus sabaeus (オナガザル) Chlorocebus sabaeus (オナガザル) |
|---|
-Cytochrome b-c1 complex subunit ... , 4種, 9分子 ATaGgCcFf
| #2: タンパク質 | 分子量: 29817.166 Da / 分子数: 3 / 由来タイプ: 天然 / 由来: (天然)  Chlorocebus sabaeus (オナガザル) / 参照: UniProt: A0A0D9QXW2, quinol-cytochrome-c reductase Chlorocebus sabaeus (オナガザル) / 参照: UniProt: A0A0D9QXW2, quinol-cytochrome-c reductase#3: タンパク質 | 分子量: 13540.450 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)  Chlorocebus sabaeus (オナガザル) / 参照: UniProt: A0A0D9RJ49 Chlorocebus sabaeus (オナガザル) / 参照: UniProt: A0A0D9RJ49#4: タンパク質 | 分子量: 9748.262 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)  Chlorocebus sabaeus (オナガザル) / 参照: UniProt: A0A0D9RML3 Chlorocebus sabaeus (オナガザル) / 参照: UniProt: A0A0D9RML3#6: タンパク質 | 分子量: 10803.954 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)  Chlorocebus sabaeus (オナガザル) / 参照: UniProt: A0A0D9S9I7 Chlorocebus sabaeus (オナガザル) / 参照: UniProt: A0A0D9S9I7 |
|---|
-Ubiquinol-cytochrome c reductase core protein ... , 2種, 4分子 KkLl
| #7: タンパク質 | 分子量: 48592.984 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)  Chlorocebus sabaeus (オナガザル) / 参照: UniProt: A0A0D9R493 Chlorocebus sabaeus (オナガザル) / 参照: UniProt: A0A0D9R493#9: タンパク質 | 分子量: 52885.816 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)  Chlorocebus sabaeus (オナガザル) Chlorocebus sabaeus (オナガザル) |
|---|
-非ポリマー , 10種, 40分子 




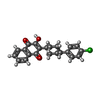

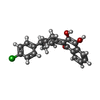











| #11: 化合物 | | #12: 化合物 | #13: 化合物 | #14: 化合物 | ChemComp-CDL / #15: 化合物 | #16: 化合物 | #17: 化合物 | ChemComp-PEE / #18: 化合物 | #19: 化合物 | ChemComp-HEM / #20: 水 | ChemComp-HOH / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|---|
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: mitochondrial respirasome from Chlorocebus sabaeus Vero cells inhibited by atovaquone タイプ: COMPLEX / Entity ID: #1-#10 / 由来: NATURAL |
|---|---|
| 分子量 | 実験値: NO |
| 由来(天然) | 生物種:  Chlorocebus sabaeus (オナガザル) / 株: Vero cell line Chlorocebus sabaeus (オナガザル) / 株: Vero cell line |
| 緩衝液 | pH: 7.4 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 301 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1600 nm / 最小 デフォーカス(公称値): 600 nm / Cs: 2.7 mm |
| 撮影 | 電子線照射量: 36 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) |
- 解析
解析
| EMソフトウェア | 名称: PHENIX / カテゴリ: モデル精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 2.67 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 637526 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー




 PDBj
PDBj























