+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8wcl | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | FCP pentamer in Chaetoceros gracilis | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | PHOTOSYNTHESIS / FCP pentamer in Chaetoceros gracilis | ||||||
| 機能・相同性 | Chem-A86 / CHLOROPHYLL A / Chlorophyll c1 / Chlorophyll c2 / 1,2-DISTEAROYL-MONOGALACTOSYL-DIGLYCERIDE 機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  Chaetoceros neogracilis (珪藻) Chaetoceros neogracilis (珪藻) | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.65 Å | ||||||
 データ登録者 データ登録者 | Feng, Y. / Li, Z. / Zhou, C. / Liu, C. / Shen, J.-R. / Wang, W. | ||||||
| 資金援助 |  中国, 1件 中国, 1件
| ||||||
 引用 引用 |  ジャーナル: Plant Commun / 年: 2024 ジャーナル: Plant Commun / 年: 2024タイトル: Structural and spectroscopic insights into fucoxanthin chlorophyll a/c-binding proteins of diatoms in diverse oligomeric states. 著者: Cuicui Zhou / Yue Feng / Zhenhua Li / Lili Shen / Xiaoyi Li / Yumei Wang / Guangye Han / Tingyun Kuang / Cheng Liu / Jian-Ren Shen / Wenda Wang /   要旨: Diatoms, a group of prevalent marine algae, contribute significantly to global primary productivity. Their substantial biomass is linked to enhanced absorption of blue-green light underwater, ...Diatoms, a group of prevalent marine algae, contribute significantly to global primary productivity. Their substantial biomass is linked to enhanced absorption of blue-green light underwater, facilitated by fucoxanthin chlorophyll (Chl) a/c-binding proteins (FCPs), which exhibit oligomeric diversity across diatom species. Using mild clear native PAGE analysis of solubilized thylakoid membranes, we displayed monomeric, dimeric, trimeric, tetrameric, and pentameric FCPs in diatoms. Mass spectrometry analysis revealed that each oligomeric FCP has a specific protein composition, and together they constitute a large Lhcf family of FCP antennas. In addition, we resolved the structures of the Thalassiosira pseudonana FCP (Tp-FCP) homotrimer and the Chaetoceros gracilis FCP (Cg-FCP) pentamer by cryoelectron microscopy at 2.73-Å and 2.65-Å resolution, respectively. The distinct pigment compositions and organizations of various oligomeric FCPs affect their blue-green light-harvesting, excitation energy transfer pathways. Compared with dimeric and trimeric FCPs, the Cg-FCP tetramer and Cg-FCP pentamer exhibit stronger absorption by Chl c, redshifted and broader Chl a fluorescence emission, and more robust circular dichroism signals originating from Chl a-carotenoid dimers. These spectroscopic characteristics indicate that Chl a molecules in the Cg-FCP tetramer and Cg-FCP pentamer are more heterogeneous than in both dimers and the Tp-FCP trimer. The structural and spectroscopic insights provided by this study contribute to a better understanding of the mechanisms that empower diatoms to adapt to fluctuating light environments. | ||||||
| 履歴 |
|
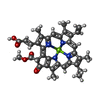
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8wcl.cif.gz 8wcl.cif.gz | 251.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8wcl.ent.gz pdb8wcl.ent.gz | 219.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8wcl.json.gz 8wcl.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8wcl_validation.pdf.gz 8wcl_validation.pdf.gz | 5.1 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8wcl_full_validation.pdf.gz 8wcl_full_validation.pdf.gz | 5.3 MB | 表示 | |
| XML形式データ |  8wcl_validation.xml.gz 8wcl_validation.xml.gz | 85.1 KB | 表示 | |
| CIF形式データ |  8wcl_validation.cif.gz 8wcl_validation.cif.gz | 102.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/wc/8wcl https://data.pdbj.org/pub/pdb/validation_reports/wc/8wcl ftp://data.pdbj.org/pub/pdb/validation_reports/wc/8wcl ftp://data.pdbj.org/pub/pdb/validation_reports/wc/8wcl | HTTPS FTP |
-関連構造データ
| 関連構造データ |  37442MC  8jp3C  8wckC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-Chlorophyll a/c-binding protein ... , 2種, 2分子 67
| #1: タンパク質 | 分子量: 22758.740 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Chaetoceros neogracilis (珪藻) Chaetoceros neogracilis (珪藻) |
|---|---|
| #4: タンパク質 | 分子量: 22298.115 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Chaetoceros neogracilis (珪藻) Chaetoceros neogracilis (珪藻) |
-タンパク質 , 2種, 3分子 859
| #2: タンパク質 | 分子量: 22098.182 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)  Chaetoceros neogracilis (珪藻) Chaetoceros neogracilis (珪藻)#3: タンパク質 | | 分子量: 21143.453 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Chaetoceros neogracilis (珪藻) Chaetoceros neogracilis (珪藻) |
|---|
-糖 , 1種, 5分子 
| #9: 糖 | ChemComp-LMT / |
|---|
-非ポリマー , 5種, 79分子 







| #5: 化合物 | ChemComp-A86 / ( #6: 化合物 | ChemComp-CLA / #7: 化合物 | ChemComp-KC2 / #8: 化合物 | ChemComp-KC1 / #10: 化合物 | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: FCP pentamer in Chaetoceros gracilis / タイプ: COMPLEX / Entity ID: #1, #3, #2, #4 / 由来: NATURAL |
|---|---|
| 由来(天然) | 生物種:  Chaetoceros neogracilis (珪藻) Chaetoceros neogracilis (珪藻) |
| 緩衝液 | pH: 6.5 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 1000 nm |
| 撮影 | 電子線照射量: 60 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) |
- 解析
解析
| EMソフトウェア | 名称: PHENIX / バージョン: 1.19.2_4158: / カテゴリ: モデル精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 2.65 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 681510 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー





 PDBj
PDBj










