+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8u53 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Mechanically activated ion channel OSCA3.1 in nanodiscs | ||||||
 要素 要素 | CSC1-like protein ERD4 | ||||||
 キーワード キーワード | MEMBRANE PROTEIN / Mechanically activated ion channel | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報plasmodesma / plant-type vacuole / calcium-activated cation channel activity / chloroplast envelope / mRNA binding / nucleus / plasma membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.6 Å | ||||||
 データ登録者 データ登録者 | Jojoa-Cruz, S. / Lee, W.H. / Ward, A.B. | ||||||
| 資金援助 |  米国, 1件 米国, 1件
| ||||||
 引用 引用 |  ジャーナル: Elife / 年: 2024 ジャーナル: Elife / 年: 2024タイトル: Structure-guided mutagenesis of OSCAs reveals differential activation to mechanical stimuli. 著者: Sebastian Jojoa-Cruz / Adrienne E Dubin / Wen-Hsin Lee / Andrew B Ward /  要旨: The dimeric two-pore OSCA/TMEM63 family has recently been identified as mechanically activated ion channels. Previously, based on the unique features of the structure of OSCA1.2, we postulated the ...The dimeric two-pore OSCA/TMEM63 family has recently been identified as mechanically activated ion channels. Previously, based on the unique features of the structure of OSCA1.2, we postulated the potential involvement of several structural elements in sensing membrane tension (Jojoa-Cruz et al., 2018). Interestingly, while OSCA1, 2, and 3 clades are activated by membrane stretch in cell-attached patches (i.e. they are stretch-activated channels), they differ in their ability to transduce membrane deformation induced by a blunt probe (poking). Here, in an effort to understand the domains contributing to mechanical signal transduction, we used cryo-electron microscopy to solve the structure of (At) OSCA3.1, which, unlike AtOSCA1.2, only produced stretch- but not poke-activated currents in our initial characterization (Murthy et al., 2018). Mutagenesis and electrophysiological assessment of conserved and divergent putative mechanosensitive features of OSCA1.2 reveal a selective disruption of the macroscopic currents elicited by poking without considerable effects on stretch-activated currents (SAC). Our results support the involvement of the amphipathic helix and lipid-interacting residues in the membrane fenestration in the response to poking. Our findings position these two structural elements as potential sources of functional diversity within the family. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8u53.cif.gz 8u53.cif.gz | 246.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8u53.ent.gz pdb8u53.ent.gz | 197 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8u53.json.gz 8u53.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8u53_validation.pdf.gz 8u53_validation.pdf.gz | 1.6 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8u53_full_validation.pdf.gz 8u53_full_validation.pdf.gz | 1.7 MB | 表示 | |
| XML形式データ |  8u53_validation.xml.gz 8u53_validation.xml.gz | 45.6 KB | 表示 | |
| CIF形式データ |  8u53_validation.cif.gz 8u53_validation.cif.gz | 67.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/u5/8u53 https://data.pdbj.org/pub/pdb/validation_reports/u5/8u53 ftp://data.pdbj.org/pub/pdb/validation_reports/u5/8u53 ftp://data.pdbj.org/pub/pdb/validation_reports/u5/8u53 | HTTPS FTP |
-関連構造データ
| 関連構造データ | 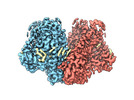 41911MC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 81878.898 Da / 分子数: 2 / 由来タイプ: 組換発現 詳細: The last 10 residues (GTGTLEVLFQ) are leftover of a linker and protease site. 由来: (組換発現)  遺伝子: ERD4 / プラスミド: pcDNA3.1 / 細胞株 (発現宿主): HEK293F / 発現宿主:  Homo sapiens (ヒト) / 参照: UniProt: Q9C8G5 Homo sapiens (ヒト) / 参照: UniProt: Q9C8G5#2: 化合物 | #3: 化合物 | #4: 化合物 | ChemComp-PLM / 研究の焦点であるリガンドがあるか | N | 配列の詳細 | Residues that could not be identified in the map are represented as UNK. The actual sequence of the ...Residues that could not be identified in the map are represented as UNK. The actual sequence of the protein construct is: MEFGSFLVSL | |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: OSCA3.1 dimer in nanodisc / タイプ: COMPLEX / Entity ID: #1 / 由来: RECOMBINANT | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 0.164 MDa / 実験値: YES | ||||||||||||||||||||
| 由来(天然) | 生物種:  | ||||||||||||||||||||
| 由来(組換発現) | 生物種:  Homo sapiens (ヒト) / 細胞: HEK293F / プラスミド: pcDNA3.1 Homo sapiens (ヒト) / 細胞: HEK293F / プラスミド: pcDNA3.1 | ||||||||||||||||||||
| 緩衝液 | pH: 8 | ||||||||||||||||||||
| 緩衝液成分 |
| ||||||||||||||||||||
| 試料 | 濃度: 3.2 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES | ||||||||||||||||||||
| 試料支持 | グリッドの材料: GOLD / グリッドのサイズ: 300 divisions/in. | ||||||||||||||||||||
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 277 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1500 nm / 最小 デフォーカス(公称値): 400 nm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 電子線照射量: 50 e/Å2 フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 撮影したグリッド数: 1 / 実像数: 8375 詳細: Only micrographs with a CTF estimate of 2.6A or better were used for processing |
| 画像スキャン | 動画フレーム数/画像: 38 / 利用したフレーム数/画像: 1-38 |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 1913316 | ||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: C2 (2回回転対称) | ||||||||||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 2.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 197944 / クラス平均像の数: 1 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: RIGID BODY FIT / 空間: REAL | ||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | Accession code: 6MGV / Source name: SwissModel / タイプ: in silico model |
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj