+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8r6y | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the SFTSV L protein stalled in a transcription-specific early elongation state with bound capped RNA [TRANSCRIPTION-EARLY-ELONGATION] | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード |  VIRAL PROTEIN (ウイルスタンパク質) / VIRAL PROTEIN (ウイルスタンパク質) /  SFTSV (重症熱性血小板減少症候群ウイルス) / SFTSV (重症熱性血小板減少症候群ウイルス) /  RNA-DEPENDENT RNA POLYMERASE (RNA依存性RNAポリメラーゼ) / RNA-DEPENDENT RNA POLYMERASE (RNA依存性RNAポリメラーゼ) /  VIRAL RNA (RNAウイルス) VIRAL RNA (RNAウイルス) | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell endoplasmic reticulum /  endoplasmic reticulum-Golgi intermediate compartment / host cell endoplasmic reticulum-Golgi intermediate compartment / host cell Golgi apparatus / endoplasmic reticulum-Golgi intermediate compartment / host cell endoplasmic reticulum-Golgi intermediate compartment / host cell Golgi apparatus /  RNA依存性RNAポリメラーゼ / viral RNA genome replication / RNA依存性RNAポリメラーゼ / viral RNA genome replication /  RNA-dependent RNA polymerase activity / DNA-templated transcription / RNA-dependent RNA polymerase activity / DNA-templated transcription /  metal ion binding metal ion binding類似検索 - 分子機能 | ||||||||||||
| 生物種 |   SFTS virus AH12 (重症熱性血小板減少症候群ウイルス) SFTS virus AH12 (重症熱性血小板減少症候群ウイルス) | ||||||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.4 Å クライオ電子顕微鏡法 / 解像度: 3.4 Å | ||||||||||||
 データ登録者 データ登録者 | Williams, H.M. / Thorkelsson, S.R. / Vogel, D. / Busch, C. / Milewski, M. / Cusack, S. / Grunewald, K. / Quemin, E.R.J. / Rosenthal, M. | ||||||||||||
| 資金援助 |  ドイツ, 3件 ドイツ, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2024 ジャーナル: Nucleic Acids Res / 年: 2024タイトル: Structural snapshots of phenuivirus cap-snatching and transcription. 著者: Harry M Williams / Sigurdur R Thorkelsson / Dominik Vogel / Carola Busch / Morlin Milewski / Stephen Cusack / Kay Grünewald / Emmanuelle R J Quemin / Maria Rosenthal /   要旨: Severe fever with thrombocytopenia syndrome virus (SFTSV) is a human pathogen that is now endemic to several East Asian countries. The viral large (L) protein catalyzes viral transcription by ...Severe fever with thrombocytopenia syndrome virus (SFTSV) is a human pathogen that is now endemic to several East Asian countries. The viral large (L) protein catalyzes viral transcription by stealing host mRNA caps via a process known as cap-snatching. Here, we establish an in vitro cap-snatching assay and present three high-quality electron cryo-microscopy (cryo-EM) structures of the SFTSV L protein in biologically relevant, transcription-specific states. In a priming-state structure, we show capped RNA bound to the L protein cap-binding domain (CBD). The L protein conformation in this priming structure is significantly different from published replication-state structures, in particular the N- and C-terminal domains. The capped-RNA is positioned in a way that it can feed directly into the RNA-dependent RNA polymerase (RdRp) ready for elongation. We also captured the L protein in an early-elongation state following primer-incorporation demonstrating that this priming conformation is retained at least in the very early stages of primer extension. This structural data is complemented by in vitro biochemical and cell-based assays. Together, these insights further our mechanistic understanding of how SFTSV and other bunyaviruses incorporate stolen host mRNA fragments into their viral transcripts thereby allowing the virus to hijack host cell translation machinery. #1:  ジャーナル: BiorXiv / 年: 2023 ジャーナル: BiorXiv / 年: 2023タイトル: Structural snapshots of phenuivirus cap-snatching and transcription 著者: Williams, H.M. / Thorkelsson, S.R. / Vogel, D. / Busch, C. / Milewski, M. / Cusack, S. / Grunewald, K. / Quemin, E.R.J. / Rosenthal, M. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8r6y.cif.gz 8r6y.cif.gz | 432.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8r6y.ent.gz pdb8r6y.ent.gz | 338 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8r6y.json.gz 8r6y.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/r6/8r6y https://data.pdbj.org/pub/pdb/validation_reports/r6/8r6y ftp://data.pdbj.org/pub/pdb/validation_reports/r6/8r6y ftp://data.pdbj.org/pub/pdb/validation_reports/r6/8r6y | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  18969MC  8r6uC  8r6wC C: 同じ文献を引用 ( M: このデータのモデリングに利用したマップデータ |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 1種, 1分子 A
| #1: タンパク質 | 分子量: 235698.500 Da / 分子数: 1 / 変異: D112A / 由来タイプ: 組換発現 由来: (組換発現)   SFTS virus AH12 (重症熱性血小板減少症候群ウイルス) SFTS virus AH12 (重症熱性血小板減少症候群ウイルス)発現宿主:   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: U3GU88 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: U3GU88 |
|---|
-RNA鎖 , 4種, 4分子 PTGC
| #2: RNA鎖 | 分子量: 6451.975 Da / 分子数: 1 / 由来タイプ: 合成 / 詳細: Synthesised by Biomers. 由来: (合成)   SFTS virus AH12 (重症熱性血小板減少症候群ウイルス) SFTS virus AH12 (重症熱性血小板減少症候群ウイルス) |
|---|---|
| #3: RNA鎖 | 分子量: 6480.872 Da / 分子数: 1 / 由来タイプ: 合成 / 詳細: Synthesised by Biomers. 由来: (合成)   SFTS virus AH12 (重症熱性血小板減少症候群ウイルス) SFTS virus AH12 (重症熱性血小板減少症候群ウイルス) |
| #4: RNA鎖 | 分子量: 918.636 Da / 分子数: 1 / 由来タイプ: 合成 / 詳細: Produced by the viral polymerase - not synthesised. 由来: (合成)   SFTS virus AH12 (重症熱性血小板減少症候群ウイルス) SFTS virus AH12 (重症熱性血小板減少症候群ウイルス) |
| #5: RNA鎖 | 分子量: 5863.617 Da / 分子数: 1 / 由来タイプ: 合成 / 詳細: Synthesised by ChemGenes. 由来: (合成)   SFTS virus AH12 (重症熱性血小板減少症候群ウイルス) SFTS virus AH12 (重症熱性血小板減少症候群ウイルス) |
-非ポリマー , 2種, 2分子 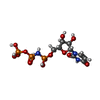


| #6: 化合物 | ChemComp-2KH / |
|---|---|
| #7: 化合物 | ChemComp-MG / |
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: SFTSV L protein in complex with 5' and 3' RNA, as well as RNA acting as a primer. タイプ: COMPLEX 詳細: SFTSV L protein expressed recombinantly in complex with several RNA species. Entity ID: #1-#4 / 由来: RECOMBINANT |
|---|---|
| 分子量 | 値: 0.24 MDa / 実験値: YES |
| 由来(天然) | 生物種:   SFTS virus AH12 (重症熱性血小板減少症候群ウイルス) SFTS virus AH12 (重症熱性血小板減少症候群ウイルス) |
| 由来(組換発現) | 生物種:   Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 緩衝液 | pH: 7 |
| 試料 | 濃度: 0.8 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES / 詳細: This sample was monodisperse. : YES / 詳細: This sample was monodisperse. |
| 試料支持 | Film material: CARBON / グリッドの材料: GOLD / グリッドのタイプ: Quantifoil R2/1 |
急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE-PROPANE / 湿度: 100 % / 凍結前の試料温度: 277 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 130000 X / 最大 デフォーカス(公称値): 2600 nm / 最小 デフォーカス(公称値): 800 nm / Cs Bright-field microscopy / 倍率(公称値): 130000 X / 最大 デフォーカス(公称値): 2600 nm / 最小 デフォーカス(公称値): 800 nm / Cs : 2.7 mm : 2.7 mm |
| 試料ホルダ | 凍結剤: NITROGEN |
| 撮影 | 電子線照射量: 50 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 27095 |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 2998852 | ||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 3.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 264083 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: RIGID BODY FIT / Target criteria: Cross-correlation coefficient | ||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj































