+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8r4c | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
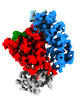
| タイトル | LRR domain of Roco protein from C. tepidum bound to the activating Nanobody NbRoco2 | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード | HYDROLASE / GTPase / Nanobody / Parkinson's Disease / Allostery activator | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |   Chlorobaculum tepidum (バクテリア) Chlorobaculum tepidum (バクテリア) | ||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.55 Å | ||||||||||||
 データ登録者 データ登録者 | Galicia, C. / Versees, W. | ||||||||||||
| 資金援助 |  ベルギー, 3件 ベルギー, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2024 ジャーナル: Elife / 年: 2024タイトル: Structural insights into the GTP-driven monomerization and activation of a bacterial LRRK2 homolog using allosteric nanobodies. 著者: Christian Galicia / Giambattista Guaitoli / Marcus Fislage / Christian Johannes Gloeckner / Wim Versées /   要旨: Roco proteins entered the limelight after mutations in human LRRK2 were identified as a major cause of familial Parkinson's disease. LRRK2 is a large and complex protein combining a GTPase and ...Roco proteins entered the limelight after mutations in human LRRK2 were identified as a major cause of familial Parkinson's disease. LRRK2 is a large and complex protein combining a GTPase and protein kinase activity, and disease mutations increase the kinase activity, while presumably decreasing the GTPase activity. Although a cross-communication between both catalytic activities has been suggested, the underlying mechanisms and the regulatory role of the GTPase domain remain unknown. Several structures of LRRK2 have been reported, but structures of Roco proteins in their activated GTP-bound state are lacking. Here, we use single-particle cryo-electron microscopy to solve the structure of a bacterial Roco protein (CtRoco) in its GTP-bound state, aided by two conformation-specific nanobodies: Nb and Nb. This structure presents CtRoco in an active monomeric state, featuring a very large GTP-induced conformational change using the LRR-Roc linker as a hinge. Furthermore, this structure shows how Nb and Nb collaborate to activate CtRoco in an allosteric way. Altogether, our data provide important new insights into the activation mechanism of Roco proteins, with relevance to LRRK2 regulation, and suggest new routes for the allosteric modulation of their GTPase activity. #1:  ジャーナル: Elife / 年: 2023 ジャーナル: Elife / 年: 2023タイトル: Structural insights in the GTP-driven monomerization and activation of a bacterial LRRK2 homologue using allosteric nanobodies 著者: Galicia, C. / Guaitoli, G. / Fislage, M. / Gloeckner, C.J. / Versees, W. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8r4c.cif.gz 8r4c.cif.gz | 118.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8r4c.ent.gz pdb8r4c.ent.gz | 79.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8r4c.json.gz 8r4c.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8r4c_validation.pdf.gz 8r4c_validation.pdf.gz | 938 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8r4c_full_validation.pdf.gz 8r4c_full_validation.pdf.gz | 945.4 KB | 表示 | |
| XML形式データ |  8r4c_validation.xml.gz 8r4c_validation.xml.gz | 28.1 KB | 表示 | |
| CIF形式データ |  8r4c_validation.cif.gz 8r4c_validation.cif.gz | 40.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/r4/8r4c https://data.pdbj.org/pub/pdb/validation_reports/r4/8r4c ftp://data.pdbj.org/pub/pdb/validation_reports/r4/8r4c ftp://data.pdbj.org/pub/pdb/validation_reports/r4/8r4c | HTTPS FTP |
-関連構造データ
| 関連構造データ |  18885MC  8r4bC  8r4dC C: 同じ文献を引用 ( M: このデータのモデリングに利用したマップデータ |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: 抗体 | 分子量: 14180.744 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   |
|---|---|
| #2: タンパク質 | 分子量: 124313.453 Da / 分子数: 1 / 由来タイプ: 組換発現 / 詳細: LRR domain of CtRoco 由来: (組換発現)  Chlorobaculum tepidum (バクテリア) Chlorobaculum tepidum (バクテリア)遺伝子: CT1526 / 発現宿主:  |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: CtRoco in the active GTP state bound to the two activating Nanobodies NbRoco1 and NbRoco2 タイプ: COMPLEX / Entity ID: all / 由来: MULTIPLE SOURCES |
|---|---|
| 分子量 | 値: 0.15 MDa / 実験値: NO |
| 由来(天然) | 生物種:  Chlorobaculum tepidum (バクテリア) Chlorobaculum tepidum (バクテリア) |
| 由来(組換発現) | 生物種:  |
| 緩衝液 | pH: 7.5 |
| 試料 | 濃度: 0.08 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE / 湿度: 90 % |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 顕微鏡 | モデル: JEOL CRYO ARM 300 |
|---|---|
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 60000 X / 最大 デフォーカス(公称値): 3000 nm / 最小 デフォーカス(公称値): 1000 nm / Cs: 2.55 mm / C2レンズ絞り径: 100 µm |
| 撮影 | 電子線照射量: 63 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) |
- 解析
解析
| EMソフトウェア | 名称: cryoSPARC / カテゴリ: 3次元再構成 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.55 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 160925 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: FLEXIBLE FIT | ||||||||||||||||||||||||
| 原子モデル構築 | 3D fitting-ID: 1
| ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー






 PDBj
PDBj








