+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8pn1 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
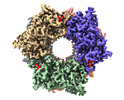
| タイトル | CryoEM structure of Nal1 protein, allele SPIKE, from Oryza sativa japonica group | |||||||||
 要素 要素 | Protein NARROW LEAF 1 | |||||||||
 キーワード キーワード | PLANT PROTEIN / Serine protease | |||||||||
| 機能・相同性 | stem vascular tissue pattern formation / internode patterning / regulation of leaf development / leaf vascular tissue pattern formation / Peptidase S1, PA clan / nucleoplasm / cytoplasm / ADENOSINE-5'-TRIPHOSPHATE / Protein NARROW LEAF 1 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.4 Å | |||||||||
 データ登録者 データ登録者 | Huang, L.Y. / Rety, S. / Xi, X.G. | |||||||||
| 資金援助 |  中国, 中国,  フランス, 2件 フランス, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Plants / 年: 2024 ジャーナル: Nat Plants / 年: 2024タイトル: The catalytic triad of rice NARROW LEAF1 involves H234. 著者: Ling-Yun Huang / Na-Nv Liu / Wei-Fei Chen / Xia Ai / Hai-Hong Li / Ze-Lin Zhang / Xi-Miao Hou / Philippe Fossé / Olivier Mauffret / Dong-Sheng Lei / Stephane Rety / Xu-Guang Xi /   要旨: NARROW LEAF1 (NAL1) exerts a multifaceted influence on leaf morphology and crop yield. Recent crystal study proposed that histidine 233 (H233) is part of the catalytic triad. Here we report that ...NARROW LEAF1 (NAL1) exerts a multifaceted influence on leaf morphology and crop yield. Recent crystal study proposed that histidine 233 (H233) is part of the catalytic triad. Here we report that unlike suggested previously, H234 instead of H233 is a component of the catalytic triad alongside residues D291 and S385 in NAL1. Remarkably, residue 233 unexpectedly plays a pivotal role in regulating NAL1's proteolytic activity. These findings establish a strong foundation for utilizing NAL1 in breeding programs aimed at improving crop yield. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8pn1.cif.gz 8pn1.cif.gz | 579.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8pn1.ent.gz pdb8pn1.ent.gz | 表示 |  PDB形式 PDB形式 | |
| PDBx/mmJSON形式 |  8pn1.json.gz 8pn1.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8pn1_validation.pdf.gz 8pn1_validation.pdf.gz | 1.5 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8pn1_full_validation.pdf.gz 8pn1_full_validation.pdf.gz | 1.5 MB | 表示 | |
| XML形式データ |  8pn1_validation.xml.gz 8pn1_validation.xml.gz | 70.9 KB | 表示 | |
| CIF形式データ |  8pn1_validation.cif.gz 8pn1_validation.cif.gz | 103 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/pn/8pn1 https://data.pdbj.org/pub/pdb/validation_reports/pn/8pn1 ftp://data.pdbj.org/pub/pdb/validation_reports/pn/8pn1 ftp://data.pdbj.org/pub/pdb/validation_reports/pn/8pn1 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  17766MC  8pmeC  8pmgC  8pmiC  8pmlC  8pmmC  8pn2C M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域: Ens-ID: ens_1
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj