+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8ost | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure of human terminal uridylyltransferase 4 (TUT4, ZCCHC11) in complex with pre-let7g miRNA and Lin28A | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | CELL CYCLE / Polymerase / uridylation / RNA maturation and turnover control / RNA | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of glial cell differentiation / positive regulation of cell proliferation involved in kidney development / negative regulation of pre-miRNA processing / polyuridylation-dependent mRNA catabolic process / retrotransposon silencing by mRNA destabilization / protein-RNA adaptor activity / miRNA catabolic process / RNA 3'-end processing / RNA 3' uridylation / RNA uridylyltransferase ...negative regulation of glial cell differentiation / positive regulation of cell proliferation involved in kidney development / negative regulation of pre-miRNA processing / polyuridylation-dependent mRNA catabolic process / retrotransposon silencing by mRNA destabilization / protein-RNA adaptor activity / miRNA catabolic process / RNA 3'-end processing / RNA 3' uridylation / RNA uridylyltransferase / pre-miRNA binding / RNA uridylyltransferase activity / Transcriptional regulation of pluripotent stem cells / miRNA metabolic process / Z-decay: degradation of maternal mRNAs by zygotically expressed factors / Deadenylation of mRNA / pre-miRNA processing / positive regulation of cytoplasmic translation / sequence-specific mRNA binding / miRNA binding / oocyte maturation / stem cell population maintenance / germ cell development / Zygotic genome activation (ZGA) / positive regulation of TOR signaling / rough endoplasmic reticulum / translation initiation factor binding / positive regulation of neuron differentiation / stem cell differentiation / cellular response to glucose stimulus / P-body / cytoplasmic stress granule / cytoplasmic ribonucleoprotein granule / G-quadruplex RNA binding / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / negative regulation of translation / mRNA binding / nucleolus / RNA binding / extracellular space / extracellular exosome / zinc ion binding / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.69 Å | ||||||
 データ登録者 データ登録者 | Gilbert, R.J. / Yi, G. / Ye, M. | ||||||
| 資金援助 |  英国, 1件 英国, 1件
| ||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Structural basis for activity switching in polymerases determining the fate of let-7 pre-miRNAs. 著者: Gangshun Yi / Mingda Ye / Loic Carrique / Afaf El-Sagheer / Tom Brown / Chris J Norbury / Peijun Zhang / Robert J C Gilbert /  要旨: Tumor-suppressor let-7 pre-microRNAs (miRNAs) are regulated by terminal uridylyltransferases TUT7 and TUT4 that either promote let-7 maturation by adding a single uridine nucleotide to the pre-miRNA ...Tumor-suppressor let-7 pre-microRNAs (miRNAs) are regulated by terminal uridylyltransferases TUT7 and TUT4 that either promote let-7 maturation by adding a single uridine nucleotide to the pre-miRNA 3' end or mark them for degradation by the addition of multiple uridines. Oligo-uridylation is increased in cells by enhanced TUT7/4 expression and especially by the RNA-binding pluripotency factor LIN28A. Using cryogenic electron microscopy, we captured high-resolution structures of active forms of TUT7 alone, of TUT7 plus pre-miRNA and of both TUT7 and TUT4 bound with pre-miRNA and LIN28A. Our structures reveal that pre-miRNAs engage the enzymes in fundamentally different ways depending on the presence of LIN28A, which clamps them onto the TUTs to enable processive 3' oligo-uridylation. This study reveals the molecular basis for mono- versus oligo-uridylation by TUT7/4, as determined by the presence of LIN28A, and thus their mechanism of action in the regulation of cell fate and in cancer. | ||||||
| 履歴 |
|
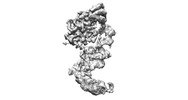
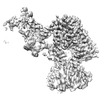
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8ost.cif.gz 8ost.cif.gz | 229.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8ost.ent.gz pdb8ost.ent.gz | 163.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8ost.json.gz 8ost.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/os/8ost https://data.pdbj.org/pub/pdb/validation_reports/os/8ost ftp://data.pdbj.org/pub/pdb/validation_reports/os/8ost ftp://data.pdbj.org/pub/pdb/validation_reports/os/8ost | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  17164MC  8oefC  8oppC  8opsC  8optC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 185433.734 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: TUT4, KIAA0191, ZCCHC11 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: TUT4, KIAA0191, ZCCHC11 / 発現宿主:  | ||
|---|---|---|---|
| #2: タンパク質 | 分子量: 22778.975 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: LIN28A, CSDD1, LIN28, ZCCHC1 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: LIN28A, CSDD1, LIN28, ZCCHC1 / 発現宿主:  | ||
| #3: RNA鎖 | 分子量: 22287.232 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)  Homo sapiens (ヒト) / 参照: GenBank: 15281257 Homo sapiens (ヒト) / 参照: GenBank: 15281257 | ||
| #4: 化合物 | | 研究の焦点であるリガンドがあるか | N | |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 実験値: NO | ||||||||||||||||||||||||
| 由来(天然) |
| ||||||||||||||||||||||||
| 由来(組換発現) |
| ||||||||||||||||||||||||
| 緩衝液 | pH: 7.8 | ||||||||||||||||||||||||
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES | ||||||||||||||||||||||||
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: TFS KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2500 nm / 最小 デフォーカス(公称値): 1500 nm |
| 撮影 | 電子線照射量: 50 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) |
- 解析
解析
| CTF補正 | タイプ: PHASE FLIPPING ONLY | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 3次元再構成 | 解像度: 3.69 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 186227 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー





 PDBj
PDBj



































