+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8oh9 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
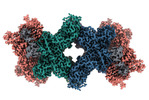
| タイトル | Cryo-EM structure of the electron bifurcating transhydrogenase StnABC complex from Sporomusa Ovata (state 1) | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | ELECTRON TRANSPORT / flavin based Electron-bifurcation / transhydrogenases / anaerobic metabolism | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報hydrogen dehydrogenase / hydrogen dehydrogenase activity / formate dehydrogenase / iron-sulfur cluster binding / 2 iron, 2 sulfur cluster binding / 4 iron, 4 sulfur cluster binding / oxidoreductase activity / DNA binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Sporomusa ovata DSM 2662 (バクテリア) Sporomusa ovata DSM 2662 (バクテリア) | |||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Kumar, A. / Kremp, F. / Mueller, V. / Schuller, J.M. | |||||||||
| 資金援助 | European Union,  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Molecular architecture and electron transfer pathway of the Stn family transhydrogenase. 著者: Anuj Kumar / Florian Kremp / Jennifer Roth / Sven A Freibert / Volker Müller / Jan M Schuller /  要旨: The challenge of endergonic reduction of NADP using NADH is overcome by ferredoxin-dependent transhydrogenases that employ electron bifurcation for electron carrier adjustments in the ancient Wood- ...The challenge of endergonic reduction of NADP using NADH is overcome by ferredoxin-dependent transhydrogenases that employ electron bifurcation for electron carrier adjustments in the ancient Wood-Ljungdahl pathway. Recently, an electron-bifurcating transhydrogenase with subunit compositions distinct from the well-characterized Nfn-type transhydrogenase was described: the Stn complex. Here, we present the single-particle cryo-EM structure of the Stn family transhydrogenase from the acetogenic bacterium Sporomusa ovata and functionally dissect its electron transfer pathway. Stn forms a tetramer consisting of functional heterotrimeric StnABC complexes. Our findings demonstrate that the StnAB subunits assume the structural and functional role of a bifurcating module, homologous to the HydBC core of the electron-bifurcating HydABC complex. Moreover, StnC contains a NuoG-like domain and a GltD-like NADPH binding domain that resembles the NfnB subunit of the NfnAB complex. However, in contrast to NfnB, StnC lost the ability to bifurcate electrons. Structural comparison allows us to describe how the same fold on one hand evolved bifurcation activity on its own while on the other hand combined with an associated bifurcating module, exemplifying modular evolution in anaerobic metabolism to produce activities critical for survival at the thermodynamic limit of life. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8oh9.cif.gz 8oh9.cif.gz | 1.2 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8oh9.ent.gz pdb8oh9.ent.gz | 1 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8oh9.json.gz 8oh9.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8oh9_validation.pdf.gz 8oh9_validation.pdf.gz | 2.3 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8oh9_full_validation.pdf.gz 8oh9_full_validation.pdf.gz | 2.3 MB | 表示 | |
| XML形式データ |  8oh9_validation.xml.gz 8oh9_validation.xml.gz | 189 KB | 表示 | |
| CIF形式データ |  8oh9_validation.cif.gz 8oh9_validation.cif.gz | 293.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/oh/8oh9 https://data.pdbj.org/pub/pdb/validation_reports/oh/8oh9 ftp://data.pdbj.org/pub/pdb/validation_reports/oh/8oh9 ftp://data.pdbj.org/pub/pdb/validation_reports/oh/8oh9 | HTTPS FTP |
-関連構造データ
| 関連構造データ | 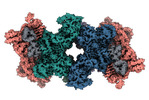 16879MC  8oh5C M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 3種, 12分子 ADGJBEHKCFIL
| #1: タンパク質 | 分子量: 18973.877 Da / 分子数: 4 / 由来タイプ: 天然 / 由来: (天然)  Sporomusa ovata DSM 2662 (バクテリア) / 参照: UniProt: A0A0U1KYW8 Sporomusa ovata DSM 2662 (バクテリア) / 参照: UniProt: A0A0U1KYW8#2: タンパク質 | 分子量: 63569.254 Da / 分子数: 4 / 由来タイプ: 天然 / 由来: (天然)  Sporomusa ovata DSM 2662 (バクテリア) / 参照: UniProt: A0A0U1KYM9 Sporomusa ovata DSM 2662 (バクテリア) / 参照: UniProt: A0A0U1KYM9#3: タンパク質 | 分子量: 126488.734 Da / 分子数: 4 / 由来タイプ: 天然 / 由来: (天然)  Sporomusa ovata DSM 2662 (バクテリア) / 参照: UniProt: A0A0U1KYI6 Sporomusa ovata DSM 2662 (バクテリア) / 参照: UniProt: A0A0U1KYI6 |
|---|
-非ポリマー , 4種, 56分子 






| #4: 化合物 | ChemComp-FES / #5: 化合物 | ChemComp-ZN / #6: 化合物 | ChemComp-SF4 / #7: 化合物 | ChemComp-FAD / |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Cryo-EM structure of the electron bifurcating trans-hydrogenase StnABC complex from Sporomusa Ovata in the oxidised state タイプ: COMPLEX / Entity ID: #1-#3 / 由来: NATURAL |
|---|---|
| 分子量 | 値: 0.85 MDa / 実験値: YES |
| 由来(天然) | 生物種:  Sporomusa ovata DSM 2662 (バクテリア) Sporomusa ovata DSM 2662 (バクテリア) |
| 緩衝液 | pH: 8 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 800 nm |
| 撮影 | 電子線照射量: 50 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) |
- 解析
解析
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 3次元再構成 | 解像度: 3.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 116573 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj
















