+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8off | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
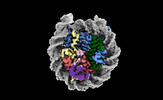
| タイトル | Structure of BARD1 ARD-BRCTs in complex with H2AKc15ub nucleosomes (Map1) | |||||||||||||||
 要素 要素 |
| |||||||||||||||
 キーワード キーワード | DNA BINDING PROTEIN / BRCA1-BARD1 / chromatin recognition / nucleosomes / ubiquitin | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報BRCA1-BARD1 complex / BRCA1-C complex / BRCA1-B complex / BRCA1-A complex / protein K6-linked ubiquitination / regulation of phosphorylation / negative regulation of protein export from nucleus / negative regulation of megakaryocyte differentiation / protein localization to CENP-A containing chromatin / Chromatin modifying enzymes ...BRCA1-BARD1 complex / BRCA1-C complex / BRCA1-B complex / BRCA1-A complex / protein K6-linked ubiquitination / regulation of phosphorylation / negative regulation of protein export from nucleus / negative regulation of megakaryocyte differentiation / protein localization to CENP-A containing chromatin / Chromatin modifying enzymes / Replacement of protamines by nucleosomes in the male pronucleus / CENP-A containing nucleosome / heterochromatin organization / epigenetic regulation of gene expression / Packaging Of Telomere Ends / Recognition and association of DNA glycosylase with site containing an affected purine / Cleavage of the damaged purine / Maturation of protein E / Deposition of new CENPA-containing nucleosomes at the centromere / Maturation of protein E / nucleosomal DNA binding / ER Quality Control Compartment (ERQC) / Myoclonic epilepsy of Lafora / FLT3 signaling by CBL mutants / Prevention of phagosomal-lysosomal fusion / IRAK2 mediated activation of TAK1 complex / Alpha-protein kinase 1 signaling pathway / Glycogen synthesis / Recognition and association of DNA glycosylase with site containing an affected pyrimidine / Cleavage of the damaged pyrimidine / IRAK1 recruits IKK complex / IRAK1 recruits IKK complex upon TLR7/8 or 9 stimulation / Membrane binding and targetting of GAG proteins / Endosomal Sorting Complex Required For Transport (ESCRT) / IRAK2 mediated activation of TAK1 complex upon TLR7/8 or 9 stimulation / PTK6 Regulates RTKs and Their Effectors AKT1 and DOK1 / Negative regulation of FLT3 / Constitutive Signaling by NOTCH1 HD Domain Mutants / Regulation of FZD by ubiquitination / TICAM1,TRAF6-dependent induction of TAK1 complex / NOTCH2 Activation and Transmission of Signal to the Nucleus / TICAM1-dependent activation of IRF3/IRF7 / Inhibition of DNA recombination at telomere / Meiotic synapsis / APC/C:Cdc20 mediated degradation of Cyclin B / p75NTR recruits signalling complexes / Downregulation of ERBB4 signaling / telomere organization / APC-Cdc20 mediated degradation of Nek2A / PINK1-PRKN Mediated Mitophagy / TRAF6-mediated induction of TAK1 complex within TLR4 complex / TRAF6 mediated IRF7 activation in TLR7/8 or 9 signaling / Pexophagy / Regulation of innate immune responses to cytosolic DNA / InlA-mediated entry of Listeria monocytogenes into host cells / VLDLR internalisation and degradation / RNA Polymerase I Promoter Opening / Interleukin-7 signaling / Downregulation of ERBB2:ERBB3 signaling / NF-kB is activated and signals survival / NRIF signals cell death from the nucleus / Regulation of PTEN localization / Assembly of the ORC complex at the origin of replication / Activated NOTCH1 Transmits Signal to the Nucleus / SUMOylation of chromatin organization proteins / Regulation of BACH1 activity / Translesion synthesis by REV1 / Synthesis of active ubiquitin: roles of E1 and E2 enzymes / Translesion synthesis by POLK / MAP3K8 (TPL2)-dependent MAPK1/3 activation / TICAM1, RIP1-mediated IKK complex recruitment / Downregulation of TGF-beta receptor signaling / DNA methylation / Activation of IRF3, IRF7 mediated by TBK1, IKKε (IKBKE) / Translesion synthesis by POLI / Gap-filling DNA repair synthesis and ligation in GG-NER / Josephin domain DUBs / Regulation of activated PAK-2p34 by proteasome mediated degradation / Condensation of Prophase Chromosomes / InlB-mediated entry of Listeria monocytogenes into host cell / IKK complex recruitment mediated by RIP1 / JNK (c-Jun kinases) phosphorylation and activation mediated by activated human TAK1 / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / SIRT1 negatively regulates rRNA expression / Chromatin modifications during the maternal to zygotic transition (MZT) / HCMV Late Events / TGF-beta receptor signaling in EMT (epithelial to mesenchymal transition) / N-glycan trimming in the ER and Calnexin/Calreticulin cycle / PRC2 methylates histones and DNA / innate immune response in mucosa / Autodegradation of Cdh1 by Cdh1:APC/C / TNFR1-induced NF-kappa-B signaling pathway / APC/C:Cdc20 mediated degradation of Securin / Asymmetric localization of PCP proteins / Defective pyroptosis / TCF dependent signaling in response to WNT / SCF-beta-TrCP mediated degradation of Emi1 / AUF1 (hnRNP D0) binds and destabilizes mRNA / NIK-->noncanonical NF-kB signaling / Ubiquitin-dependent degradation of Cyclin D 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||||||||
 データ登録者 データ登録者 | Foglizzo, M. / Burdett, H. / Wilson, M.D. / Zeqiraj, E. | |||||||||||||||
| 資金援助 |  英国, 4件 英国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2023 ジャーナル: Nucleic Acids Res / 年: 2023タイトル: BRCA1-BARD1 combines multiple chromatin recognition modules to bridge nascent nucleosomes. 著者: Hayden Burdett / Martina Foglizzo / Laura J Musgrove / Dhananjay Kumar / Gillian Clifford / Lisa J Campbell / George R Heath / Elton Zeqiraj / Marcus D Wilson /  要旨: Chromatin association of the BRCA1-BARD1 heterodimer is critical to promote homologous recombination repair of DNA double-strand breaks (DSBs) in S/G2. How the BRCA1-BARD1 complex interacts with ...Chromatin association of the BRCA1-BARD1 heterodimer is critical to promote homologous recombination repair of DNA double-strand breaks (DSBs) in S/G2. How the BRCA1-BARD1 complex interacts with chromatin that contains both damage induced histone H2A ubiquitin and inhibitory H4K20 methylation is not fully understood. We characterised BRCA1-BARD1 binding and enzymatic activity to an array of mono- and di-nucleosome substrates using biochemical, structural and single molecule imaging approaches. We found that the BRCA1-BARD1 complex preferentially interacts and modifies di-nucleosomes over mono-nucleosomes, allowing integration of H2A Lys-15 ubiquitylation signals with other chromatin modifications and features. Using high speed- atomic force microscopy (HS-AFM) to monitor how the BRCA1-BARD1 complex recognises chromatin in real time, we saw a highly dynamic complex that bridges two nucleosomes and associates with the DNA linker region. Bridging is aided by multivalent cross-nucleosome interactions that enhance BRCA1-BARD1 E3 ubiquitin ligase catalytic activity. Multivalent interactions across nucleosomes explain how BRCA1-BARD1 can recognise chromatin that retains partial di-methylation at H4 Lys-20 (H4K20me2), a parental histone mark that blocks BRCA1-BARD1 interaction with nucleosomes, to promote its enzymatic and DNA repair activities. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8off.cif.gz 8off.cif.gz | 365 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8off.ent.gz pdb8off.ent.gz | 表示 |  PDB形式 PDB形式 | |
| PDBx/mmJSON形式 |  8off.json.gz 8off.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8off_validation.pdf.gz 8off_validation.pdf.gz | 1.3 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8off_full_validation.pdf.gz 8off_full_validation.pdf.gz | 1.3 MB | 表示 | |
| XML形式データ |  8off_validation.xml.gz 8off_validation.xml.gz | 44 KB | 表示 | |
| CIF形式データ |  8off_validation.cif.gz 8off_validation.cif.gz | 68.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/of/8off https://data.pdbj.org/pub/pdb/validation_reports/of/8off ftp://data.pdbj.org/pub/pdb/validation_reports/of/8off ftp://data.pdbj.org/pub/pdb/validation_reports/of/8off | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-DNA鎖 , 1種, 2分子 JI
| #1: DNA鎖 | 分子量: 108097.586 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 発現宿主: Homo sapiens (ヒト) / 発現宿主:  |
|---|
-タンパク質 , 6種, 11分子 CaCbBaBbAaAbDaDbEaEbFa
| #2: タンパク質 | 分子量: 15366.088 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) Homo sapiens (ヒト)遺伝子: H3C1, H3FA, HIST1H3A, H3C2, H3FL, HIST1H3B, H3C3, H3FC HIST1H3C, H3C4, H3FB, HIST1H3D, H3C6, H3FD, HIST1H3E, H3C7, H3FI, HIST1H3F, H3C8, H3FH, HIST1H3G, H3C10, H3FK, HIST1H3H, H3C11, H3FF, ...遺伝子: H3C1, H3FA, HIST1H3A, H3C2, H3FL, HIST1H3B, H3C3, H3FC HIST1H3C, H3C4, H3FB, HIST1H3D, H3C6, H3FD, HIST1H3E, H3C7, H3FI, HIST1H3F, H3C8, H3FH, HIST1H3G, H3C10, H3FK, HIST1H3H, H3C11, H3FF, HIST1H3I, H3C12, H3FJ, HIST1H3J 発現宿主:  #3: タンパク質 | 分子量: 13937.213 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) Homo sapiens (ヒト)遺伝子: HIST1H2BC, H2BFL, HIST1H2BE, H2BFH, HIST1H2BF, H2BFG, HIST1H2BG, H2BFA, HIST1H2BI, H2BFK 発現宿主:  #4: タンパク質 | 分子量: 14121.537 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) Homo sapiens (ヒト)遺伝子: H2AC11, H2AFP, HIST1H2AG, H2AC13, H2AFC, HIST1H2AI, H2AC15, H2AFD, HIST1H2AK, H2AC16, H2AFI, HIST1H2AL, H2AC17, H2AFN, HIST1H2AM 発現宿主:  #5: タンパク質 | 分子量: 11394.426 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) Homo sapiens (ヒト)遺伝子: HIST1H4A, H4/A, H4FA, HIST1H4B, H4/I, H4FI, HIST1H4C, H4/G, H4FG, HIST1H4D, H4/B, H4FB, HIST1H4E, H4/J, H4FJ, HIST1H4F, H4/C, H4FC, HIST1H4H, H4/H, H4FH, HIST1H4I, H4/M, H4FM, HIST1H4J, ...遺伝子: HIST1H4A, H4/A, H4FA, HIST1H4B, H4/I, H4FI, HIST1H4C, H4/G, H4FG, HIST1H4D, H4/B, H4FB, HIST1H4E, H4/J, H4FJ, HIST1H4F, H4/C, H4FC, HIST1H4H, H4/H, H4FH, HIST1H4I, H4/M, H4FM, HIST1H4J, H4/E, H4FE, HIST1H4K, H4/D, H4FD, HIST1H4L, H4/K, H4FK, HIST2H4A, H4/N, H4F2, H4FN, HIST2H4, HIST2H4B, H4/O, H4FO, HIST4H4 発現宿主:  #6: タンパク質 | 分子量: 85897.531 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: M3WD26 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: M3WD26#7: タンパク質 | | 分子量: 8576.831 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: UBC / 発現宿主: Homo sapiens (ヒト) / 遺伝子: UBC / 発現宿主:  |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | N |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 0.246 MDa / 実験値: NO | ||||||||||||||||||||||||
| 由来(天然) |
| ||||||||||||||||||||||||
| 由来(組換発現) |
| ||||||||||||||||||||||||
| 緩衝液 | pH: 7.5 / 詳細: 20 mM HEPES pH 7.5, 50 mM NaCl and 1 mM DTT | ||||||||||||||||||||||||
| 緩衝液成分 |
| ||||||||||||||||||||||||
| 試料 | 濃度: 1 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES 詳細: Purified cat BRCA1dExon11-FL BARD1 (at 3 uM) was incubated with H2AKc15ub mono-nucleosomes (at 1.5 uM) for 1 h on ice before cryo-EM grids preparation | ||||||||||||||||||||||||
| 試料支持 | 詳細: Quantifoil R3.5/1 200-mesh grids (Quantifoil Micro Tools GmbH) were glow-discharged for 30 s at 40 mA using a GloQube (Quorum) glow discharge unit グリッドの材料: COPPER / グリッドのサイズ: 200 divisions/in. / グリッドのタイプ: Quantifoil R3.5/1 | ||||||||||||||||||||||||
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 277 K / 詳細: blot force = 0 N blot time = 8 s |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 96000 X / 最大 デフォーカス(公称値): 3100 nm / 最小 デフォーカス(公称値): 1700 nm / Cs: 2.7 mm / C2レンズ絞り径: 70 µm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 5 sec. / 電子線照射量: 36.37 e/Å2 フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 撮影したグリッド数: 1 / 実像数: 16015 |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 7204662 | ||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: C1 (非対称) | ||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 359954 / クラス平均像の数: 3 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | 空間: REAL | ||||||||||||||||||||||||||||||||||||
| 精密化 | 交差検証法: NONE 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2 | ||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 72.1 Å2 | ||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー






 PDBj
PDBj





















































