+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8gf5 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
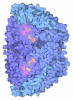
| タイトル | McrD binds asymmetrically to methyl-coenzyme M reductase improving active site accessibility during assembly | ||||||||||||||||||||||||
 要素 要素 |
| ||||||||||||||||||||||||
 キーワード キーワード | TRANSFERASE / methanogenesis / MCR complex | ||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報coenzyme-B sulfoethylthiotransferase / coenzyme-B sulfoethylthiotransferase activity / methanogenesis / metal ion binding / cytoplasm 類似検索 - 分子機能 | ||||||||||||||||||||||||
| 生物種 |  Methanosarcina acetivorans C2A (古細菌) Methanosarcina acetivorans C2A (古細菌) | ||||||||||||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Joiner, A.M.N. / Chadwick, G.L. / Nayak, D.D. | ||||||||||||||||||||||||
| 資金援助 |  米国, 7件 米国, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2023 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023タイトル: McrD binds asymmetrically to methyl-coenzyme M reductase improving active-site accessibility during assembly. 著者: Grayson L Chadwick / Aaron M N Joiner / Sangeetha Ramesh / Douglas A Mitchell / Dipti D Nayak /  要旨: Methyl-coenzyme M reductase (MCR) catalyzes the formation of methane, and its activity accounts for nearly all biologically produced methane released into the atmosphere. The assembly of MCR is an ...Methyl-coenzyme M reductase (MCR) catalyzes the formation of methane, and its activity accounts for nearly all biologically produced methane released into the atmosphere. The assembly of MCR is an intricate process involving the installation of a complex set of posttranslational modifications and the unique Ni-containing tetrapyrrole called coenzyme F. Despite decades of research, details of MCR assembly remain largely unresolved. Here, we report the structural characterization of MCR in two intermediate states of assembly. These intermediate states lack one or both F cofactors and form complexes with the previously uncharacterized McrD protein. McrD is found to bind asymmetrically to MCR, displacing large regions of the alpha subunit and increasing active-site accessibility for the installation of F-shedding light on the assembly of MCR and the role of McrD therein. This work offers crucial information for the expression of MCR in a heterologous host and provides targets for the design of MCR inhibitors. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8gf5.cif.gz 8gf5.cif.gz | 419.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8gf5.ent.gz pdb8gf5.ent.gz | 346.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8gf5.json.gz 8gf5.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8gf5_validation.pdf.gz 8gf5_validation.pdf.gz | 1.8 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8gf5_full_validation.pdf.gz 8gf5_full_validation.pdf.gz | 1.8 MB | 表示 | |
| XML形式データ |  8gf5_validation.xml.gz 8gf5_validation.xml.gz | 87.4 KB | 表示 | |
| CIF形式データ |  8gf5_validation.cif.gz 8gf5_validation.cif.gz | 128 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/gf/8gf5 https://data.pdbj.org/pub/pdb/validation_reports/gf/8gf5 ftp://data.pdbj.org/pub/pdb/validation_reports/gf/8gf5 ftp://data.pdbj.org/pub/pdb/validation_reports/gf/8gf5 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  29978MC  8gf6C M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-Methyl-coenzyme M reductase subunit ... , 3種, 6分子 ABCDEF
| #1: タンパク質 | 分子量: 62180.078 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)  Methanosarcina acetivorans C2A (古細菌) Methanosarcina acetivorans C2A (古細菌)参照: UniProt: Q8THH1, coenzyme-B sulfoethylthiotransferase #2: タンパク質 | 分子量: 45174.070 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)  Methanosarcina acetivorans C2A (古細菌) / 参照: UniProt: Q8THG7 Methanosarcina acetivorans C2A (古細菌) / 参照: UniProt: Q8THG7#3: タンパク質 | 分子量: 27630.184 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)  Methanosarcina acetivorans C2A (古細菌) / 参照: UniProt: Q8THH0 Methanosarcina acetivorans C2A (古細菌) / 参照: UniProt: Q8THH0 |
|---|
-タンパク質 , 1種, 1分子 X
| #4: タンパク質 | 分子量: 21842.742 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Methanosarcina acetivorans C2A (古細菌) Methanosarcina acetivorans C2A (古細菌)遺伝子: mcrD / 発現宿主:  Methanosarcina acetivorans C2A (古細菌) / 参照: UniProt: Q8THG8 Methanosarcina acetivorans C2A (古細菌) / 参照: UniProt: Q8THG8 |
|---|
-非ポリマー , 3種, 3分子 



| #5: 化合物 | ChemComp-COM / |
|---|---|
| #6: 化合物 | ChemComp-F43 / |
| #7: 化合物 | ChemComp-TP7 / |
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Assembly intermediate of the MCR complex bound to mcrD タイプ: COMPLEX / Entity ID: #1-#4 / 由来: NATURAL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 由来(天然) | 生物種:  Methanosarcina acetivorans C2A (古細菌) Methanosarcina acetivorans C2A (古細菌) | |||||||||||||||
| 緩衝液 | pH: 8 | |||||||||||||||
| 緩衝液成分 |
| |||||||||||||||
| 試料 | 濃度: 0.55 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES | |||||||||||||||
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 277 K / 詳細: Vitrification occurred under aerobic conditions |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TALOS ARCTICA |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 36000 X / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 800 nm |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 電子線照射量: 50 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) |
- 解析
解析
| EMソフトウェア | 名称: SerialEM / カテゴリ: 画像取得 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 65000 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー




 PDBj
PDBj