+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8cf1 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Tetracycline bound to the 30S head | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード | RIBOSOME / Antibiotic | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報transcription antitermination factor activity, RNA binding / transcription elongation factor complex / regulation of DNA-templated transcription elongation / transcription antitermination / maintenance of translational fidelity / ribosome biogenesis / ribosomal small subunit assembly / small ribosomal subunit / cytosolic small ribosomal subunit / cytoplasmic translation ...transcription antitermination factor activity, RNA binding / transcription elongation factor complex / regulation of DNA-templated transcription elongation / transcription antitermination / maintenance of translational fidelity / ribosome biogenesis / ribosomal small subunit assembly / small ribosomal subunit / cytosolic small ribosomal subunit / cytoplasmic translation / tRNA binding / negative regulation of translation / rRNA binding / structural constituent of ribosome / ribosome / translation / response to antibiotic / mRNA binding / RNA binding / zinc ion binding / membrane / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 1.82 Å | ||||||||||||
 データ登録者 データ登録者 | Paternoga, H. / Crowe-McAuliffe, C. / Beckert, B. / Wilson, D.N. | ||||||||||||
| 資金援助 | European Union, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: Structural conservation of antibiotic interaction with ribosomes. 著者: Helge Paternoga / Caillan Crowe-McAuliffe / Lars V Bock / Timm O Koller / Martino Morici / Bertrand Beckert / Alexander G Myasnikov / Helmut Grubmüller / Jiří Nováček / Daniel N Wilson /    要旨: The ribosome is a major target for clinically used antibiotics, but multidrug resistant pathogenic bacteria are making our current arsenal of antimicrobials obsolete. Here we present cryo-electron- ...The ribosome is a major target for clinically used antibiotics, but multidrug resistant pathogenic bacteria are making our current arsenal of antimicrobials obsolete. Here we present cryo-electron-microscopy structures of 17 distinct compounds from six different antibiotic classes bound to the bacterial ribosome at resolutions ranging from 1.6 to 2.2 Å. The improved resolution enables a precise description of antibiotic-ribosome interactions, encompassing solvent networks that mediate multiple additional interactions between the drugs and their target. Our results reveal a high structural conservation in the binding mode between antibiotics with the same scaffold, including ordered water molecules. Water molecules are visualized within the antibiotic binding sites that are preordered, become ordered in the presence of the drug and that are physically displaced on drug binding. Insight into RNA-ligand interactions will facilitate development of new antimicrobial agents, as well as other RNA-targeting therapies. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8cf1.cif.gz 8cf1.cif.gz | 533.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8cf1.ent.gz pdb8cf1.ent.gz | 390.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8cf1.json.gz 8cf1.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8cf1_validation.pdf.gz 8cf1_validation.pdf.gz | 1.3 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8cf1_full_validation.pdf.gz 8cf1_full_validation.pdf.gz | 1.3 MB | 表示 | |
| XML形式データ |  8cf1_validation.xml.gz 8cf1_validation.xml.gz | 57.7 KB | 表示 | |
| CIF形式データ |  8cf1_validation.cif.gz 8cf1_validation.cif.gz | 90.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/cf/8cf1 https://data.pdbj.org/pub/pdb/validation_reports/cf/8cf1 ftp://data.pdbj.org/pub/pdb/validation_reports/cf/8cf1 ftp://data.pdbj.org/pub/pdb/validation_reports/cf/8cf1 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  16615MC  8ca7C  8caiC  8camC  8cazC  8cepC  8ceuC  8cf8C  8cgdC  8cgiC  8cgjC  8cgkC  8cgrC  8cguC  8cgvC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-RNA鎖 , 1種, 1分子 A
| #1: RNA鎖 | 分子量: 499197.938 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
|---|
-30S ribosomal protein ... , 2種, 2分子 BG
| #2: タンパク質 | 分子量: 26781.670 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
|---|---|
| #5: タンパク質 | 分子量: 20055.156 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
-Small ribosomal subunit protein ... , 7種, 7分子 CEIJMNS
| #3: タンパク質 | 分子量: 26031.316 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
|---|---|
| #4: タンパク質 | 分子量: 17629.398 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #6: タンパク質 | 分子量: 14886.270 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #7: タンパク質 | 分子量: 11755.597 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #8: タンパク質 | 分子量: 13128.467 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #9: タンパク質 | 分子量: 11606.560 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #10: タンパク質 | 分子量: 10455.355 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
-非ポリマー , 4種, 721分子 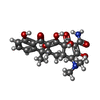






| #11: 化合物 | ChemComp-TAC / | ||||
|---|---|---|---|---|---|
| #12: 化合物 | ChemComp-MG / #13: 化合物 | ChemComp-K / #14: 水 | ChemComp-HOH / | |
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: 70S ribosomes with antibiotic cocktail / タイプ: RIBOSOME 詳細: 70S + Tetracycline, Lincomycin, Avilamycin, Streptomycin Entity ID: #1-#10 / 由来: NATURAL | |||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 実験値: NO | |||||||||||||||||||||||||||||||||||
| 由来(天然) | 生物種:  | |||||||||||||||||||||||||||||||||||
| 緩衝液 | pH: 7.5 | |||||||||||||||||||||||||||||||||||
| 緩衝液成分 |
| |||||||||||||||||||||||||||||||||||
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES | |||||||||||||||||||||||||||||||||||
| 試料支持 | グリッドの材料: GOLD / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Quantifoil R1.2/1.3 | |||||||||||||||||||||||||||||||||||
| 急速凍結 | 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 277.15 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1000 nm / 最小 デフォーカス(公称値): 400 nm |
| 撮影 | 平均露光時間: 4.5 sec. / 電子線照射量: 60 e/Å2 / 検出モード: COUNTING フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) |
- 解析
解析
| EMソフトウェア | 名称: REFMAC / バージョン: 5.8.0415 / カテゴリ: モデル精密化 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 1.82 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 419159 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | 解像度: 1.82→143.01 Å / Cor.coef. Fo:Fc: 0.733 / SU B: 3.157 / SU ML: 0.084 / ESU R: 0.097 立体化学のターゲット値: MAXIMUM LIKELIHOOD WITH PHASES 詳細: HYDROGENS HAVE BEEN USED IF PRESENT IN THE INPUT
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: PARAMETERS FOR MASK CACLULATION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 46.945 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 合計: 18957 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー

















 PDBj
PDBj































