+ データを開く
データを開く
- 基本情報
基本情報
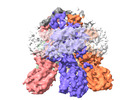
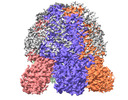
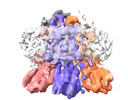
| 登録情報 | データベース: PDB / ID: 8bri | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | VaPomAB MSP1D1 nanodisc | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード | MOTOR PROTEIN / Flagellar sodium-driven stator unit | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報bacterial-type flagellum-dependent swarming motility / proton transmembrane transport / chemotaxis / plasma membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Vibrio alginolyticus (バクテリア) Vibrio alginolyticus (バクテリア) | ||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | ||||||||||||
 データ登録者 データ登録者 | Hu, H. / Taylor, N.M.I. | ||||||||||||
| 資金援助 |  デンマーク, 3件 デンマーク, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Ion selectivity and rotor coupling of the Vibrio flagellar sodium-driven stator unit. 著者: Haidai Hu / Philipp F Popp / Mònica Santiveri / Aritz Roa-Eguiara / Yumeng Yan / Freddie J O Martin / Zheyi Liu / Navish Wadhwa / Yong Wang / Marc Erhardt / Nicholas M I Taylor /     要旨: Bacteria swim using a flagellar motor that is powered by stator units. Vibrio spp. are highly motile bacteria responsible for various human diseases, the polar flagella of which are exclusively ...Bacteria swim using a flagellar motor that is powered by stator units. Vibrio spp. are highly motile bacteria responsible for various human diseases, the polar flagella of which are exclusively driven by sodium-dependent stator units (PomAB). However, how ion selectivity is attained, how ion transport triggers the directional rotation of the stator unit, and how the stator unit is incorporated into the flagellar rotor remained largely unclear. Here, we have determined by cryo-electron microscopy the structure of Vibrio PomAB. The electrostatic potential map uncovers sodium binding sites, which together with functional experiments and molecular dynamics simulations, reveal a mechanism for ion translocation and selectivity. Bulky hydrophobic residues from PomA prime PomA for clockwise rotation. We propose that a dynamic helical motif in PomA regulates the distance between PomA subunit cytoplasmic domains, stator unit activation, and torque transmission. Together, our study provides mechanistic insights for understanding ion selectivity and rotor incorporation of the stator unit of the bacterial flagellum. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8bri.cif.gz 8bri.cif.gz | 228.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8bri.ent.gz pdb8bri.ent.gz | 183.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8bri.json.gz 8bri.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8bri_validation.pdf.gz 8bri_validation.pdf.gz | 1.3 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8bri_full_validation.pdf.gz 8bri_full_validation.pdf.gz | 1.3 MB | 表示 | |
| XML形式データ |  8bri_validation.xml.gz 8bri_validation.xml.gz | 58.6 KB | 表示 | |
| CIF形式データ |  8bri_validation.cif.gz 8bri_validation.cif.gz | 85 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/br/8bri https://data.pdbj.org/pub/pdb/validation_reports/br/8bri ftp://data.pdbj.org/pub/pdb/validation_reports/br/8bri ftp://data.pdbj.org/pub/pdb/validation_reports/br/8bri | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 27283.031 Da / 分子数: 5 / 由来タイプ: 組換発現 由来: (組換発現)  Vibrio alginolyticus (バクテリア) Vibrio alginolyticus (バクテリア)遺伝子: pomA / 発現宿主:  #2: タンパク質 | 分子量: 35587.254 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)  Vibrio alginolyticus (バクテリア) Vibrio alginolyticus (バクテリア)遺伝子: N646_2843 / 発現宿主:  |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: flagellar sodium-driven stator unit / タイプ: COMPLEX / Entity ID: all / 由来: RECOMBINANT |
|---|---|
| 由来(天然) | 生物種:  Vibrio alginolyticus (バクテリア) Vibrio alginolyticus (バクテリア) |
| 由来(組換発現) | 生物種:  |
| 緩衝液 | pH: 7.5 / 詳細: 300mM NaCl, 20mM HEPES 7.5, 0.002%LMNG |
| 試料 | 濃度: 16 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: SPOT SCAN FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: SPOT SCAN |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2500 nm / 最小 デフォーカス(公称値): 500 nm |
| 撮影 | 電子線照射量: 38 e/Å2 フィルム・検出器のモデル: FEI FALCON II (4k x 4k) |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.20.1_4487: / 分類: 精密化 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: NONE | ||||||||||||
| 3次元再構成 | 解像度: 3.9 Å / 解像度の算出法: DIFFRACTION PATTERN/LAYERLINES / 粒子像の数: 66296 / 対称性のタイプ: POINT | ||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー




 PDBj
PDBj