+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8b9f | ||||||
|---|---|---|---|---|---|---|---|
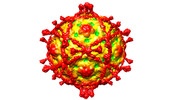
| タイトル | Structure of Echovirus 11 complexed with DAF (CD55) calculated from symmetry expansion | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | VIRUS / Echovirus / receptor / CD55 / DAF | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of lipopolysaccharide-mediated signaling pathway / negative regulation of complement activation / negative regulation of complement activation, classical pathway / regulation of complement-dependent cytotoxicity / RNA-protein covalent cross-linking / regulation of complement activation / T cell mediated immunity / respiratory burst / : / positive regulation of CD4-positive, alpha-beta T cell activation ...regulation of lipopolysaccharide-mediated signaling pathway / negative regulation of complement activation / negative regulation of complement activation, classical pathway / regulation of complement-dependent cytotoxicity / RNA-protein covalent cross-linking / regulation of complement activation / T cell mediated immunity / respiratory burst / : / positive regulation of CD4-positive, alpha-beta T cell activation / positive regulation of CD4-positive, alpha-beta T cell proliferation / : / Class B/2 (Secretin family receptors) / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / ficolin-1-rich granule membrane / side of membrane / COPI-mediated anterograde transport / transport vesicle / picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / ribonucleoside triphosphate phosphatase activity / endoplasmic reticulum-Golgi intermediate compartment membrane / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / secretory granule membrane / T=pseudo3 icosahedral viral capsid / complement activation, classical pathway / Regulation of Complement cascade / host cell cytoplasmic vesicle membrane / cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / positive regulation of T cell cytokine production / nucleoside-triphosphate phosphatase / protein complex oligomerization / monoatomic ion channel activity / virus receptor activity / symbiont-mediated suppression of host gene expression / positive regulation of cytosolic calcium ion concentration / DNA replication / RNA helicase activity / induction by virus of host autophagy / membrane raft / RNA-directed RNA polymerase / Golgi membrane / viral RNA genome replication / cysteine-type endopeptidase activity / RNA-dependent RNA polymerase activity / innate immune response / DNA-templated transcription / lipid binding / host cell nucleus / Neutrophil degranulation / virion attachment to host cell / structural molecule activity / cell surface / proteolysis / RNA binding / extracellular exosome / extracellular region / ATP binding / metal ion binding / plasma membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト)  Echovirus E11 (ウイルス) Echovirus E11 (ウイルス) | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.93 Å | ||||||
 データ登録者 データ登録者 | Stuart, D.I. / Ren, J. / Qin, L. / Zhou, D. | ||||||
| 資金援助 |  英国, 1件 英国, 1件
| ||||||
 引用 引用 |  ジャーナル: Viruses / 年: 2022 ジャーナル: Viruses / 年: 2022タイトル: Switching of Receptor Binding Poses between Closely Related Enteroviruses. 著者: Daming Zhou / Ling Qin / Helen M E Duyvesteyn / Yuguang Zhao / Tzou-Yien Lin / Elizabeth E Fry / Jingshan Ren / Kuan-Ying A Huang / David I Stuart /   要旨: Echoviruses, for which there are currently no approved vaccines or drugs, are responsible for a range of human diseases, for example echovirus 11 (E11) is a major cause of serious neonatal morbidity ...Echoviruses, for which there are currently no approved vaccines or drugs, are responsible for a range of human diseases, for example echovirus 11 (E11) is a major cause of serious neonatal morbidity and mortality. Decay-accelerating factor (DAF, also known as CD55) is an attachment receptor for E11. Here, we report the structure of the complex of E11 and the full-length ectodomain of DAF (short consensus repeats, SCRs, 1-4) at 3.1 Å determined by cryo-electron microscopy (cryo-EM). SCRs 3 and 4 of DAF interact with E11 at the southern rim of the canyon via the VP2 EF and VP3 BC loops. We also observe an unexpected interaction between the N-linked glycan (residue 95 of DAF) and the VP2 BC loop of E11. DAF is a receptor for at least 20 enteroviruses and we classify its binding patterns from reported DAF/virus complexes into two distinct positions and orientations, named as E6 and E11 poses. Whilst 60 DAF molecules can attach to the virion in the E6 pose, no more than 30 can attach to E11 due to steric restrictions. Analysis of the distinct modes of interaction and structure and sequence-based phylogenies suggests that the two modes evolved independently, with the E6 mode likely found earlier. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8b9f.cif.gz 8b9f.cif.gz | 156.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8b9f.ent.gz pdb8b9f.ent.gz | 119.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8b9f.json.gz 8b9f.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8b9f_validation.pdf.gz 8b9f_validation.pdf.gz | 1.3 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8b9f_full_validation.pdf.gz 8b9f_full_validation.pdf.gz | 1.3 MB | 表示 | |
| XML形式データ |  8b9f_validation.xml.gz 8b9f_validation.xml.gz | 35.9 KB | 表示 | |
| CIF形式データ |  8b9f_validation.cif.gz 8b9f_validation.cif.gz | 50.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/b9/8b9f https://data.pdbj.org/pub/pdb/validation_reports/b9/8b9f ftp://data.pdbj.org/pub/pdb/validation_reports/b9/8b9f ftp://data.pdbj.org/pub/pdb/validation_reports/b9/8b9f | HTTPS FTP |
-関連構造データ
| 関連構造データ |  15930MC  8b8rC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 29771.059 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: CD55, CR, DAF / 細胞株 (発現宿主): HEK293T / 発現宿主: Homo sapiens (ヒト) / 遺伝子: CD55, CR, DAF / 細胞株 (発現宿主): HEK293T / 発現宿主:  Homo sapiens (ヒト) / 参照: UniProt: P08174 Homo sapiens (ヒト) / 参照: UniProt: P08174 |
|---|---|
| #2: タンパク質 | 分子量: 28886.428 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Echovirus E11 (ウイルス) / 細胞株 (発現宿主): Rhabdomyosarcoma (RD) cells / 発現宿主: Echovirus E11 (ウイルス) / 細胞株 (発現宿主): Rhabdomyosarcoma (RD) cells / 発現宿主:  Homo sapiens (ヒト) Homo sapiens (ヒト)参照: UniProt: A0A6M5CIM5, picornain 2A, nucleoside-triphosphate phosphatase, picornain 3C, RNA-directed RNA polymerase |
| #3: タンパク質 | 分子量: 26029.674 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Echovirus E11 (ウイルス) / 細胞株 (発現宿主): Rhabdomyosarcoma (RD) cells / 発現宿主: Echovirus E11 (ウイルス) / 細胞株 (発現宿主): Rhabdomyosarcoma (RD) cells / 発現宿主:  Homo sapiens (ヒト) Homo sapiens (ヒト)参照: UniProt: A0A7T7IN41, picornain 2A, nucleoside-triphosphate phosphatase, picornain 3C, RNA-directed RNA polymerase |
| #4: タンパク質 | 分子量: 32284.262 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Echovirus E11 (ウイルス) / 細胞株 (発現宿主): Rhabdomyosarcoma (RD) cells / 発現宿主: Echovirus E11 (ウイルス) / 細胞株 (発現宿主): Rhabdomyosarcoma (RD) cells / 発現宿主:  Homo sapiens (ヒト) Homo sapiens (ヒト)参照: UniProt: A0A6M5CIM5, picornain 2A, nucleoside-triphosphate phosphatase, picornain 3C, RNA-directed RNA polymerase |
| #5: 化合物 | ChemComp-SPH / |
| 研究の焦点であるリガンドがあるか | N |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Complex of Echovirus 11 with DAF / タイプ: COMPLEX / Entity ID: #1-#4 / 由来: RECOMBINANT |
|---|---|
| 由来(天然) | 生物種:   Echovirus E11 (ウイルス) Echovirus E11 (ウイルス) |
| 由来(組換発現) | 生物種:  Homo sapiens (ヒト) / 細胞: HEK293T Homo sapiens (ヒト) / 細胞: HEK293T |
| 緩衝液 | pH: 7.4 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: SPOT SCAN FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: SPOT SCAN |
| 電子レンズ | モード: DARK FIELD / 最大 デフォーカス(公称値): 3000 nm / 最小 デフォーカス(公称値): 1000 nm |
| 撮影 | 電子線照射量: 41 e/Å2 フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) |
- 解析
解析
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION |
|---|---|
| 3次元再構成 | 解像度: 3.93 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 201358 / 対称性のタイプ: POINT |
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj








