+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7pky | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Half-vault structure | |||||||||
 要素 要素 | Major vault protein | |||||||||
 キーワード キーワード | STRUCTURAL PROTEIN / Transport | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein activation cascade / negative regulation of protein autophosphorylation / ERBB signaling pathway / Neutrophil degranulation / negative regulation of epidermal growth factor receptor signaling pathway / protein phosphatase binding / cell population proliferation / cytoskeleton / ribonucleoprotein complex / protein kinase binding ...protein activation cascade / negative regulation of protein autophosphorylation / ERBB signaling pathway / Neutrophil degranulation / negative regulation of epidermal growth factor receptor signaling pathway / protein phosphatase binding / cell population proliferation / cytoskeleton / ribonucleoprotein complex / protein kinase binding / perinuclear region of cytoplasm / identical protein binding / nucleus / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.9 Å | |||||||||
 データ登録者 データ登録者 | Guerra, P. / Gonzalez-Alamos, M. / Llauro, A. / Casanas, A. / Querol-Audi, J. / de Pablo, P. / Verdaguer, N. | |||||||||
| 資金援助 |  スペイン, 2件 スペイン, 2件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: Symmetry disruption commits vault particles to disassembly. 著者: Pablo Guerra / María González-Alamos / Aida Llauró / Arnau Casañas / Jordi Querol-Audí / Pedro J de Pablo / Núria Verdaguer /  要旨: Vaults are ubiquitous ribonucleoprotein particles involved in a diversity of cellular processes, with promising applications as nanodevices for delivery of multiple cargos. The vault shell is ...Vaults are ubiquitous ribonucleoprotein particles involved in a diversity of cellular processes, with promising applications as nanodevices for delivery of multiple cargos. The vault shell is assembled by the symmetrical association of multiple copies of the major vault protein that, initially, generates half vaults. The pairwise, anti-parallel association of two half vaults produces whole vaults. Here, using a combination of vault recombinant reconstitution and structural techniques, we characterized the molecular determinants for the vault opening process. This process commences with a relaxation of the vault waist, causing the expansion of the inner cavity. Then, local disengagement of amino-terminal domains at the vault midsection seeds a conformational change that leads to the aperture, facilitating access to the inner cavity where cargo is hosted. These results inform a hitherto uncharacterized step of the vault cycle and will aid current engineering efforts leveraging vault for tailored cargo delivery. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7pky.cif.gz 7pky.cif.gz | 5.1 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7pky.ent.gz pdb7pky.ent.gz | 表示 |  PDB形式 PDB形式 | |
| PDBx/mmJSON形式 |  7pky.json.gz 7pky.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7pky_validation.pdf.gz 7pky_validation.pdf.gz | 1.4 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7pky_full_validation.pdf.gz 7pky_full_validation.pdf.gz | 1.5 MB | 表示 | |
| XML形式データ |  7pky_validation.xml.gz 7pky_validation.xml.gz | 647.2 KB | 表示 | |
| CIF形式データ |  7pky_validation.cif.gz 7pky_validation.cif.gz | 1020.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/pk/7pky https://data.pdbj.org/pub/pdb/validation_reports/pk/7pky ftp://data.pdbj.org/pub/pdb/validation_reports/pk/7pky ftp://data.pdbj.org/pub/pdb/validation_reports/pk/7pky | HTTPS FTP |
-関連構造データ
| 関連構造データ |  13482MC  7pkrC  7pkzC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 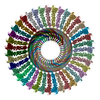
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
|
 ムービー
ムービー コントローラー
コントローラー





 PDBj
PDBj





 microscopy
microscopy