+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9277 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single particle cryoEM structure of a DARPin-aldolase platform in complex with GFP | |||||||||
 マップデータ マップデータ | DARPin-aldolase platform in complex with GFP, Relion Post processed map with B factor -25 angstrom squared | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | synthetic construct / platform / single particle cryoEM / small protein display / LYASE-FLUORESCENT PROTEIN complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of Arp2/3 complex-mediated actin nucleation / fructose-bisphosphate aldolase / fructose-bisphosphate aldolase activity / M band / I band / fructose 1,6-bisphosphate metabolic process / bioluminescence / generation of precursor metabolites and energy / glycolytic process / protein homotetramerization ...negative regulation of Arp2/3 complex-mediated actin nucleation / fructose-bisphosphate aldolase / fructose-bisphosphate aldolase activity / M band / I band / fructose 1,6-bisphosphate metabolic process / bioluminescence / generation of precursor metabolites and energy / glycolytic process / protein homotetramerization / positive regulation of cell migration / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 | synthetic construct (人工物) /   | |||||||||
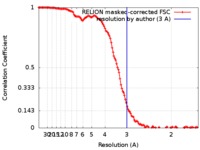
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Weaver SJ / Yao Q | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2019 ジャーナル: Structure / 年: 2019タイトル: Fusion of DARPin to Aldolase Enables Visualization of Small Protein by Cryo-EM. 著者: Qing Yao / Sara J Weaver / Jee-Young Mock / Grant J Jensen /  要旨: Solving protein structures by single-particle cryoelectron microscopy (cryo-EM) has become a crucial tool in structural biology. While exciting progress is being made toward the visualization of ...Solving protein structures by single-particle cryoelectron microscopy (cryo-EM) has become a crucial tool in structural biology. While exciting progress is being made toward the visualization of small macromolecules, the median protein size in both eukaryotes and bacteria is still beyond the reach of cryo-EM. To overcome this problem, we implemented a platform strategy in which a small protein target was rigidly attached to a large, symmetric base via a selectable adapter. Of our seven designs, the best construct used a designed ankyrin repeat protein (DARPin) rigidly fused to tetrameric rabbit muscle aldolase through a helical linker. The DARPin retained its ability to bind its target: GFP. We solved the structure of this complex to 3.0 Å resolution overall, with 5-8 Å resolution in the GFP region. As flexibility in the DARPin position limited the overall resolution of the target, we describe strategies to rigidify this element. #1:  ジャーナル: Biorxiv / 年: 2018 ジャーナル: Biorxiv / 年: 2018タイトル: Fusion of DARPin to aldolase enables visualization of small protein by cryoEM 著者: Yao Q / Weaver SJ / Mock JY / Jensen GJ | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9277.map.gz emd_9277.map.gz | 14.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9277-v30.xml emd-9277-v30.xml emd-9277.xml emd-9277.xml | 24.2 KB 24.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9277_fsc.xml emd_9277_fsc.xml | 12.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9277.png emd_9277.png | 67.9 KB | ||
| Filedesc metadata |  emd-9277.cif.gz emd-9277.cif.gz | 7.5 KB | ||
| その他 |  emd_9277_half_map_1.map.gz emd_9277_half_map_1.map.gz emd_9277_half_map_2.map.gz emd_9277_half_map_2.map.gz | 140.8 MB 140.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9277 http://ftp.pdbj.org/pub/emdb/structures/EMD-9277 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9277 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9277 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9277_validation.pdf.gz emd_9277_validation.pdf.gz | 768.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9277_full_validation.pdf.gz emd_9277_full_validation.pdf.gz | 768.3 KB | 表示 | |
| XML形式データ |  emd_9277_validation.xml.gz emd_9277_validation.xml.gz | 19.6 KB | 表示 | |
| CIF形式データ |  emd_9277_validation.cif.gz emd_9277_validation.cif.gz | 25.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9277 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9277 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9277 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9277 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9277.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9277.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DARPin-aldolase platform in complex with GFP, Relion Post processed map with B factor -25 angstrom squared | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8315 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Half map 1 of a DARPin-aldolase platform in complex with GFP
| ファイル | emd_9277_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 of a DARPin-aldolase platform in complex with GFP | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2 of a DARPin-aldolase platform in complex with GFP
| ファイル | emd_9277_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 of a DARPin-aldolase platform in complex with GFP | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : DARPin-aldolase platform in complex with GFP
| 全体 | 名称: DARPin-aldolase platform in complex with GFP |
|---|---|
| 要素 |
|
-超分子 #1: DARPin-aldolase platform in complex with GFP
| 超分子 | 名称: DARPin-aldolase platform in complex with GFP / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 314 KDa |
-超分子 #2: DARPin, Muscle-type aldolase chimeric fusion
| 超分子 | 名称: DARPin, Muscle-type aldolase chimeric fusion / タイプ: complex / ID: 2 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: Green fluorescent protein
| 超分子 | 名称: Green fluorescent protein / タイプ: complex / ID: 3 / 親要素: 1 |
|---|
-分子 #1: DARPin, Muscle-type aldolase chimeric fusion
| 分子 | 名称: DARPin, Muscle-type aldolase chimeric fusion / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号: fructose-bisphosphate aldolase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 53.113434 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGSDLGKKLL EAARAGQDDE VRILMANGAD VNALDRFGLT PLHLAAQRGH LEIVEVLLKC GADVNAADLW GQTPLHLAAT AGHLEIVEV LLKYGADVNA LDLIGKTPLH LTAIDGHLEI VEVLLKHGAD VNAQDKFGKT AFDISIDNGN EDLAEILQKL N LSDIAHRI ...文字列: SGSDLGKKLL EAARAGQDDE VRILMANGAD VNALDRFGLT PLHLAAQRGH LEIVEVLLKC GADVNAADLW GQTPLHLAAT AGHLEIVEV LLKYGADVNA LDLIGKTPLH LTAIDGHLEI VEVLLKHGAD VNAQDKFGKT AFDISIDNGN EDLAEILQKL N LSDIAHRI VAPGKGILAA DESTGSIAKR LQSIGTENTE ENRRFYRQLL LTADDRVNPC IGGVILFHET LYQKADDGRP FP QVIKSKG GVVGIKVDKG VVPLAGTNGE TTTQGLDGLS ERCAQYKKDG ADFAKWRCVL KIGEHTPSAL AIMENANVLA RYA SICQQN GIVPIVEPEI LPDGDHDLKR CQYVTEKVLA AVYKALSDHH IYLEGTLLKP NMVTPGHACT QKYSHEEIAM ATVT ALRRT VPPAVTGVTF LSGGQSEEEA SINLNAINKC PLLKPWALTF SYGRALQASA LKAWGGKKEN LKAAQEEYVK RALAN SLAC QGKYTPSGQ UniProtKB: Fructose-bisphosphate aldolase A |
-分子 #2: Green fluorescent protein
| 分子 | 名称: Green fluorescent protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 25.473697 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SKGEELFTGV VPILVELDGD VNGHKFSVSG EGEGDATYGK LTLKFICTTG KLPVPWPTLV TTLVQCFSRY PDHMKQHDFF KSAMPEGYV QERTIFFKDD GNYKTRAEVK FEGDTLVNRI ELKGIDFKED GNILGHKLEY NYNSHNVYIM ADKQKNGIKV N FKIRHNIE ...文字列: SKGEELFTGV VPILVELDGD VNGHKFSVSG EGEGDATYGK LTLKFICTTG KLPVPWPTLV TTLVQCFSRY PDHMKQHDFF KSAMPEGYV QERTIFFKDD GNYKTRAEVK FEGDTLVNRI ELKGIDFKED GNILGHKLEY NYNSHNVYIM ADKQKNGIKV N FKIRHNIE DGSVQLADHY QQNTPIGDGP VLLPDNHYLS TQSALSKDPN EKRDHMVLLE FVTAAGIT UniProtKB: Green fluorescent protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| グリッド | モデル: UltrAuFoil / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: PLASMA CLEANING | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277.15 K / 装置: HOMEMADE PLUNGER 詳細: Grids were frozen on a manual plunger at the Scripps Research Institute Core Microscopy Facility in a 4 degrees C cold room humidified to >95%.. | |||||||||
| 詳細 | The protein complex was then purified with Ni-NTA affinity chromatography (Qiagen), and Superdex 200 chromatography (GE healthcare). The purified GFP-DARPin-aldolase complex was concentrated to 2.5mg/ml in a buffer containing 25 mM Tris-HCl pH 8.0 and 150 mM NaCl. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) #0 - 検出モード: SUPER-RESOLUTION / #0 - 撮影したグリッド数: 1 / #0 - 実像数: 1133 / #0 - 平均露光時間: 4.0 sec. / #0 - 平均電子線量: 2.3 e/Å2 #0 - 詳細: All three microscope sessions used grids frozen in the same session. In the first microscopy session the stage was untilted (0 degrees). #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) #1 - 検出モード: SUPER-RESOLUTION / #1 - 撮影したグリッド数: 1 / #1 - 実像数: 548 / #1 - 平均露光時間: 4.0 sec. / #1 - 平均電子線量: 2.3 e/Å2 #1 - 詳細: All three microscope sessions used grids frozen in the same session. In the second microscopy session the stage was untilted (0 degrees). #2 - Image recording ID: 3 #2 - フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) #2 - 検出モード: SUPER-RESOLUTION / #2 - デジタル化 - 画像ごとのフレーム数: 1-40 / #2 - 撮影したグリッド数: 1 / #2 - 実像数: 1180 / #2 - 平均露光時間: 4.0 sec. / #2 - 平均電子線量: 1.1 e/Å2 #2 - 詳細: All three microscope sessions used grids frozen in the same session. In the third microscopy session the stage was tilted to 26 degrees. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 30.0 µm / 最小 デフォーカス(公称値): 10.0 µm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 詳細 | PDB models 5vy5 and 5ma6 were used as starting points. These models were mutated to match the sequence of the DARPin-aldolase platform in complex with GFP that were used. The PDB models were docked into the cryoEM density using Chimera fit into map function. No model refinement was used. | ||||||||
| 精密化 | プロトコル: RIGID BODY FIT | ||||||||
| 得られたモデル |  PDB-6mwq: |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)