+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6907 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Atomic structure of the herpes simplex virus type 2 B-capsid | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | herpesvirus / capsid / cryo-em / atomic structure / VIRUS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報T=16 icosahedral viral capsid / viral capsid assembly / viral process / virion component / viral capsid / host cell nucleus / structural molecule activity / DNA binding 類似検索 - 分子機能 | |||||||||
| 生物種 |   Human herpesvirus 2 (ヘルペスウイルス) Human herpesvirus 2 (ヘルペスウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Yuan S / Wang JL / Zhu DJ / Wang N / Gao Q / Chen WY / Tang H / Wang JZ / Zhang XZ / Liu HR ...Yuan S / Wang JL / Zhu DJ / Wang N / Gao Q / Chen WY / Tang H / Wang JZ / Zhang XZ / Liu HR / Rao ZH / Wang XX | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2018 ジャーナル: Nat Commun / 年: 2018タイトル: Pushing the resolution limit by correcting the Ewald sphere effect in single-particle Cryo-EM reconstructions. 著者: Dongjie Zhu / Xiangxi Wang / Qianglin Fang / James L Van Etten / Michael G Rossmann / Zihe Rao / Xinzheng Zhang /   要旨: The Ewald sphere effect is generally neglected when using the Central Projection Theorem for cryo electron microscopy single-particle reconstructions. This can reduce the resolution of a ...The Ewald sphere effect is generally neglected when using the Central Projection Theorem for cryo electron microscopy single-particle reconstructions. This can reduce the resolution of a reconstruction. Here we estimate the attainable resolution and report a "block-based" reconstruction method for extending the resolution limit. We find the Ewald sphere effect limits the resolution of large objects, especially large viruses. After processing two real datasets of large viruses, we show that our procedure can extend the resolution for both datasets and can accommodate the flexibility associated with large protein complexes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6907.map.gz emd_6907.map.gz | 1.2 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6907-v30.xml emd-6907-v30.xml emd-6907.xml emd-6907.xml | 15 KB 15 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_6907.png emd_6907.png | 297.7 KB | ||
| Filedesc metadata |  emd-6907.cif.gz emd-6907.cif.gz | 6.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6907 http://ftp.pdbj.org/pub/emdb/structures/EMD-6907 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6907 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6907 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6907_validation.pdf.gz emd_6907_validation.pdf.gz | 550.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6907_full_validation.pdf.gz emd_6907_full_validation.pdf.gz | 550.1 KB | 表示 | |
| XML形式データ |  emd_6907_validation.xml.gz emd_6907_validation.xml.gz | 12.4 KB | 表示 | |
| CIF形式データ |  emd_6907_validation.cif.gz emd_6907_validation.cif.gz | 14.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6907 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6907 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6907 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6907 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  5zapMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6907.map.gz / 形式: CCP4 / 大きさ: 6.4 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6907.map.gz / 形式: CCP4 / 大きさ: 6.4 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.38 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
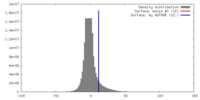
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Human herpesvirus 2
| 全体 | 名称:   Human herpesvirus 2 (ヘルペスウイルス) Human herpesvirus 2 (ヘルペスウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Human herpesvirus 2
| 超分子 | 名称: Human herpesvirus 2 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1, #4, #2-#3 / NCBI-ID: 10310 / 生物種: Human herpesvirus 2 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:   Human herpesvirus 2 (ヘルペスウイルス) Human herpesvirus 2 (ヘルペスウイルス) |
-分子 #1: Major capsid protein
| 分子 | 名称: Major capsid protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 16 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human herpesvirus 2 (ヘルペスウイルス) Human herpesvirus 2 (ヘルペスウイルス) |
| 分子量 | 理論値: 149.370359 KDa |
| 配列 | 文字列: MAAPARDPPG YRYAAAMVPT GSILSTIEVA SHRRLFDFFA RVRSDENSLY DVEFDALLGS YCNTLSLVRF LELGLSVACV CTKFPELAY MNEGRVQFEV HQPLIARDGP HPVEQPVHNY MTKVIDRRAL NAAFSLATEA IALLTGEALD GTGISLHRQL R AIQQLARN ...文字列: MAAPARDPPG YRYAAAMVPT GSILSTIEVA SHRRLFDFFA RVRSDENSLY DVEFDALLGS YCNTLSLVRF LELGLSVACV CTKFPELAY MNEGRVQFEV HQPLIARDGP HPVEQPVHNY MTKVIDRRAL NAAFSLATEA IALLTGEALD GTGISLHRQL R AIQQLARN VQAVLGAFER GTADQMLHVL LEKAPPLALL LPMQRYLDNG RLATRVARAT LVAELKRSFC DTSFFLGKAG HR REAIEAW LVDLTTATQP SVAVPRLTHA DTRGRPVDGV LVTTAAIKQR LLQSFLKVED TEADVPVTYG EMVLNGANLV TAL VMGKAV RSLDDVGRHL LDMQEEQLEA NRETLDELES APQTTRVRAD LVAIGDRLVF LEALEKRIYA ATNVPYPLVG AMDL TFVLP LGLFNPAMER FAAHAGDLVP APGHPEPRAF PPRQLFFWGK DHQVLRLSME NAVGTVCHPS LMNIDAAVGG VNHDP VEAA NPYGAYVAAP AGPGADMQQR FLNAWRQRLA HGRVRWVAEC QMTAEQFMQP DNANLALELH PAFDFFAGVA DVELPG GEV PPAGPGAIQA TWRVVNGNLP LALCPVAFRD ARGLELGVGR HAMAPATIAA VRGAFEDRSY PAVFYLLQAA IHGSEHV FC ALARLVTQCI TSYWNNTRCA AFVNDYSLVS YIVTYLGGDL PEECMAVYRD LVAHVEALAQ LVDDFTLPGP ELGGQAQA E LNHLMRDPAL LPPLVWDCDG LMRHAALDRH RDCRIDAGGH EPVYAAACNV ATADFNRNDG RLLHNTQARA ADAADDRPH RPADWTVHHK IYYYVLVPAF SRGRCCTAGV RFDRVYATLQ NMVVPEIAPG EECPSDPVTD PAHPLHPANL VANTVNAMFH NGRVVVDGP AMLTLQVLAH NMAERTTALL CSAAPDAGAN TASTANMRIF DGALHAGVLL MAPQHLDHTI QNGEYFYVLP V HALFAGAD HVANAPNFPP ALRDLARHVP LVPPALGANY FSSIRQPVVQ HARESAAGEN ALTYALMAGY FKMSPVALYH QL KTGLHPG FGFTVVRQDR FVTENVLFSE RASEAYFLGQ LQVARHETGG GVNFTLTQPR GNVDLGVGYT AVAATATVRN PVT DMGNLP QNFYLGRGAP PLLDNAAAVY LRNAVVAGNR LGPAQPLPVF GCAQVPRRAG MDHGQDAVCE FIATPVATDI NYFR RPCNP RGRAAGGVYA GDKEGDVIAL MYDHGQSDPA RPFAATANPW ASQRFSYGDL LYNGAYHLNG ASPVLSPCFK FFTAA DITA KHRCLERLIV ETGSAVSTAT AASDVQFKRP PGCRELVEDP CGLFQEAYPI TCASDPALLR SARDGEAHAR ETHFTQ YLI YDASPLKGLS L UniProtKB: Major capsid protein |
-分子 #2: Triplex capsid protein 2
| 分子 | 名称: Triplex capsid protein 2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 10 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human herpesvirus 2 (ヘルペスウイルス) Human herpesvirus 2 (ヘルペスウイルス) |
| 分子量 | 理論値: 34.373785 KDa |
| 配列 | 文字列: MITDCFEADI AIPSGISRPD AAALQRCEGR VVFLPTIRRQ LALADVAHES FVSGGVSPDT LGLLLAYRRR FPAVITRVLP TRIVACPVD LGLTHAGTVN LRNTSPVDLC NGDPVSLVPP VFEGQATDVR LESLDLTLRF PVPLPTPLAR EIVARLVARG I RDLNPDPR ...文字列: MITDCFEADI AIPSGISRPD AAALQRCEGR VVFLPTIRRQ LALADVAHES FVSGGVSPDT LGLLLAYRRR FPAVITRVLP TRIVACPVD LGLTHAGTVN LRNTSPVDLC NGDPVSLVPP VFEGQATDVR LESLDLTLRF PVPLPTPLAR EIVARLVARG I RDLNPDPR TPGELPDLNV LYYNGARLSL VADVQQLASV NTELRSLVLN MVYSITEGTT LILTLIPRLL ALSAQDGYVN AL LQMQSVT REAAQLIHPE APMLMQDGER RLPLYEALVA WLAHAGQLGD ILALAPAVRV CTFDGAAVVQ SGDMAPVIRY P UniProtKB: Capsid triplex subunit 2 |
-分子 #3: Triplex capsid protein 1
| 分子 | 名称: Triplex capsid protein 1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human herpesvirus 2 (ヘルペスウイルス) Human herpesvirus 2 (ヘルペスウイルス) |
| 分子量 | 理論値: 50.542617 KDa |
| 配列 | 文字列: MKTKPLPTAP MAWAESAVET TTSPRELAGH APLRRVLRPP IARRDGPVLL GDRAPRRTAS TMWLLGIDPA ESSPGTRATR DDTEQAVDK ILRGARRAGG LTVPGAPRYH LTRQVTLTDL CQPNAERAGA LLLALRHPTD LPHLARHRAP PGRQTERLAE A WGQLLEAS ...文字列: MKTKPLPTAP MAWAESAVET TTSPRELAGH APLRRVLRPP IARRDGPVLL GDRAPRRTAS TMWLLGIDPA ESSPGTRATR DDTEQAVDK ILRGARRAGG LTVPGAPRYH LTRQVTLTDL CQPNAERAGA LLLALRHPTD LPHLARHRAP PGRQTERLAE A WGQLLEAS ALGSGRAESG CARAGLVSFN FLVAACTAAY DARDAAEAVR AHITTNYGGT RAGARLDRFS ECLRAMVHTH VF PHEVMRF FGGLVSWVTQ DELASVTAVC SGPQEATHTG HPGRPCSAVT IPACAFVDLD AELCLGGPGA AFLYLVFTYR QCR DQELCC VYVVKSQLPP RGLEAALERL FGRLRITNTI HGAEDMTPPP PNRNVDFPLA VLAASSQSPR CSASQVTNPQ FVDR LYRWQ PDLRGRPTAR TCTYAAFAEL GVMPDDSPRC LHRTERFGAV GVPVVILEGV VWRPGGWRAC A UniProtKB: Capsid triplex subunit 1 |
-分子 #4: Small capsomere-interacting protein
| 分子 | 名称: Small capsomere-interacting protein / タイプ: protein_or_peptide / ID: 4 / コピー数: 15 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human herpesvirus 2 (ヘルペスウイルス) Human herpesvirus 2 (ヘルペスウイルス) |
| 分子量 | 理論値: 12.147707 KDa |
| 配列 | 文字列: MAAPQFHRPS TITADNVRAL GMRGLVLATN NAQFIMDNSY PHPHGTQGAV REFLRGQAAA LTDLGVTHAN NTFAPQPMFA GDAAAEWLR PSFGLKRTYS PFVVRDPKTP STP UniProtKB: Small capsomere-interacting protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 平均電子線量: 1.1 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: EMDB MAP |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 45000 |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: COMMON LINE |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)