[日本語] English
 万見
万見- EMDB-50339: Cryo-EM structure of Trypanosoma cruzi glycosomal malate dehydrogenase -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Trypanosoma cruzi glycosomal malate dehydrogenase | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Oxidoreductase (酸化還元酵素) / Oxidoreductase (酸化還元酵素) /  Tetramer (四量体) / Metabolic enzyme Tetramer (四量体) / Metabolic enzyme | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 リンゴ酸デヒドロゲナーゼ / L-malate dehydrogenase (NAD+) activity / carboxylic acid metabolic process / リンゴ酸デヒドロゲナーゼ / L-malate dehydrogenase (NAD+) activity / carboxylic acid metabolic process /  クエン酸回路 クエン酸回路類似検索 - 分子機能 | |||||||||
| 生物種 |   Trypanosoma cruzi strain CL Brener (クルーズトリパノソーマ) Trypanosoma cruzi strain CL Brener (クルーズトリパノソーマ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.03 Å クライオ電子顕微鏡法 / 解像度: 3.03 Å | |||||||||
 データ登録者 データ登録者 | Lipinski O / Sonani RR / Blat A / Jemiola-Rzeminska M / Patel SN / Sood T / Dubin G | |||||||||
| 資金援助 |  ポーランド, 2件 ポーランド, 2件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-EM structure of Trypanosoma cruzi glycosomal malate dehydrogenase 著者: Lipinski O / Sonani RR / Blat A / Jemiola-Rzeminska M / Patel SN / Sood T / Dubin G | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_50339.map.gz emd_50339.map.gz | 38.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-50339-v30.xml emd-50339-v30.xml emd-50339.xml emd-50339.xml | 13.4 KB 13.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_50339.png emd_50339.png | 172.7 KB | ||
| Filedesc metadata |  emd-50339.cif.gz emd-50339.cif.gz | 5.3 KB | ||
| その他 |  emd_50339_half_map_1.map.gz emd_50339_half_map_1.map.gz emd_50339_half_map_2.map.gz emd_50339_half_map_2.map.gz | 37.8 MB 37.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-50339 http://ftp.pdbj.org/pub/emdb/structures/EMD-50339 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50339 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50339 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9feeMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_50339.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_50339.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_50339_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
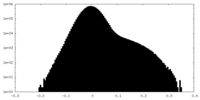
| 投影像・断面図 |
| ||||||||||||
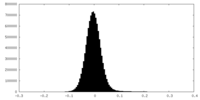
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_50339_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
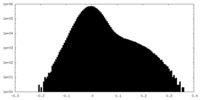
| 投影像・断面図 |
| ||||||||||||
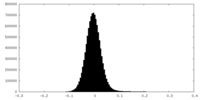
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Tetrameric complex of glycosomal malate dehydrogenase
| 全体 | 名称: Tetrameric complex of glycosomal malate dehydrogenase |
|---|---|
| 要素 |
|
-超分子 #1: Tetrameric complex of glycosomal malate dehydrogenase
| 超分子 | 名称: Tetrameric complex of glycosomal malate dehydrogenase タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Trypanosoma cruzi strain CL Brener (クルーズトリパノソーマ) Trypanosoma cruzi strain CL Brener (クルーズトリパノソーマ) |
| 分子量 | 理論値: 140 KDa |
-分子 #1: malate dehydrogenase
| 分子 | 名称: malate dehydrogenase / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号:  リンゴ酸デヒドロゲナーゼ リンゴ酸デヒドロゲナーゼ |
|---|---|
| 由来(天然) | 生物種:   Trypanosoma cruzi strain CL Brener (クルーズトリパノソーマ) Trypanosoma cruzi strain CL Brener (クルーズトリパノソーマ) |
| 分子量 | 理論値: 35.13798 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MAHHHHHHMV NVAVIGAAGG IGQSLSLLLL RELPFGSTLS LYDVVGAPGV AADLSHIDRA GITVKHAAGK LPPVPRDPAL TELAEGVDV FVIVAGVPRK PGMTRDDLFN VNAGIVMDLV LTCASVSPNA CFCIVTNPVN STTPIAAQTL RKIGVYNKNK L LGVSLLDG ...文字列: MAHHHHHHMV NVAVIGAAGG IGQSLSLLLL RELPFGSTLS LYDVVGAPGV AADLSHIDRA GITVKHAAGK LPPVPRDPAL TELAEGVDV FVIVAGVPRK PGMTRDDLFN VNAGIVMDLV LTCASVSPNA CFCIVTNPVN STTPIAAQTL RKIGVYNKNK L LGVSLLDG LRATRFINNA RHPLVVPYVP VVGGHSDVTI VPLYSQIPGP LPDESTLKEI RKRVQVAGTE VVKAKAGRGS AT LSMAEAG ARFTMHVVKA LMGLDTPMVY AYVDTDGEHE CPFLAMPVVL GKNGIERRLP IGPITTVEKE MLEEAVGVVK KNI AKGETF ARSKL UniProtKB:  リンゴ酸デヒドロゲナーゼ リンゴ酸デヒドロゲナーゼ |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.9 µm Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.9 µm |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 42.25 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.03 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 312103 |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X