+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
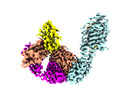
| タイトル | Cryo-EM structure of CCR6 bound by SQA1 and OXM1 | |||||||||
 マップデータ マップデータ | Cryo-EM reconstruction of CCR6 bound by SQA1 and OXM1 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | GPCR / Antagonist / CCR6 / BRIL / MEMBRANE PROTEIN | |||||||||
| 生物種 |  Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | |||||||||
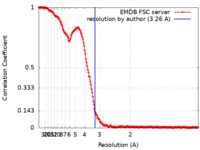
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.26 Å | |||||||||
 データ登録者 データ登録者 | Wasilko DJ / Wu H | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Structural basis for CCR6 modulation by allosteric antagonists. 著者: David Jonathan Wasilko / Brian S Gerstenberger / Kathleen A Farley / Wei Li / Jennifer Alley / Mark E Schnute / Ray J Unwalla / Jorge Victorino / Kimberly K Crouse / Ru Ding / Parag V ...著者: David Jonathan Wasilko / Brian S Gerstenberger / Kathleen A Farley / Wei Li / Jennifer Alley / Mark E Schnute / Ray J Unwalla / Jorge Victorino / Kimberly K Crouse / Ru Ding / Parag V Sahasrabudhe / Fabien Vincent / Richard K Frisbie / Alpay Dermenci / Andrew Flick / Chulho Choi / Gary Chinigo / James J Mousseau / John I Trujillo / Philippe Nuhant / Prolay Mondal / Vincent Lombardo / Daniel Lamb / Barbara J Hogan / Gurdeep Singh Minhas / Elena Segala / Christine Oswald / Ian W Windsor / Seungil Han / Mathieu Rappas / Robert M Cooke / Matthew F Calabrese / Gabriel Berstein / Atli Thorarensen / Huixian Wu /   要旨: The CC chemokine receptor 6 (CCR6) is a potential target for chronic inflammatory diseases. Previously, we reported an active CCR6 structure in complex with its cognate chemokine CCL20, revealing the ...The CC chemokine receptor 6 (CCR6) is a potential target for chronic inflammatory diseases. Previously, we reported an active CCR6 structure in complex with its cognate chemokine CCL20, revealing the molecular basis of CCR6 activation. Here, we present two inactive CCR6 structures in ternary complexes with different allosteric antagonists, CCR6/SQA1/OXM1 and CCR6/SQA1/OXM2. The oxomorpholine analogues, OXM1 and OXM2 are highly selective CCR6 antagonists which bind to an extracellular pocket and disrupt the receptor activation network. An energetically favoured U-shaped conformation in solution that resembles the bound form is observed for the active analogues. SQA1 is a squaramide derivative with close-in analogues reported as antagonists of chemokine receptors including CCR6. SQA1 binds to an intracellular pocket which overlaps with the G protein site, stabilizing a closed pocket that is a hallmark of inactive GPCRs. Minimal communication between the two allosteric pockets is observed, in contrast to the prevalent allosteric cooperativity model of GPCRs. This work highlights the versatility of GPCR antagonism by small molecules, complementing previous knowledge of CCR6 activation, and sheds light on drug discovery targeting CCR6. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_46534.map.gz emd_46534.map.gz | 506.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-46534-v30.xml emd-46534-v30.xml emd-46534.xml emd-46534.xml | 22.8 KB 22.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_46534_fsc.xml emd_46534_fsc.xml | 23.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_46534.png emd_46534.png | 70.2 KB | ||
| Filedesc metadata |  emd-46534.cif.gz emd-46534.cif.gz | 7.1 KB | ||
| その他 |  emd_46534_half_map_1.map.gz emd_46534_half_map_1.map.gz emd_46534_half_map_2.map.gz emd_46534_half_map_2.map.gz | 497.2 MB 497.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-46534 http://ftp.pdbj.org/pub/emdb/structures/EMD-46534 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-46534 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-46534 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_46534_validation.pdf.gz emd_46534_validation.pdf.gz | 902.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_46534_full_validation.pdf.gz emd_46534_full_validation.pdf.gz | 902.4 KB | 表示 | |
| XML形式データ |  emd_46534_validation.xml.gz emd_46534_validation.xml.gz | 25.1 KB | 表示 | |
| CIF形式データ |  emd_46534_validation.cif.gz emd_46534_validation.cif.gz | 33.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-46534 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-46534 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-46534 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-46534 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_46534.map.gz / 形式: CCP4 / 大きさ: 536.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_46534.map.gz / 形式: CCP4 / 大きさ: 536.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM reconstruction of CCR6 bound by SQA1 and OXM1 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.575 Å | ||||||||||||||||||||||||||||||||||||
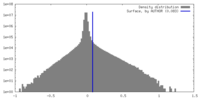
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: EM half map 1
| ファイル | emd_46534_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
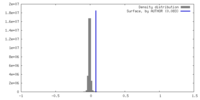
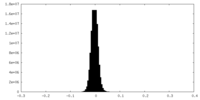
| 密度ヒストグラム |
-ハーフマップ: EM half map 2
| ファイル | emd_46534_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
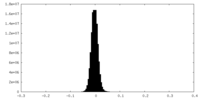
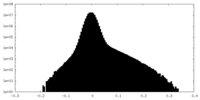
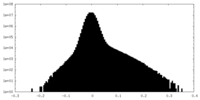
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CCR6-BRIL/Fab/Nb in complex with SQA1 and OXM1
| 全体 | 名称: CCR6-BRIL/Fab/Nb in complex with SQA1 and OXM1 |
|---|---|
| 要素 |
|
-超分子 #1: CCR6-BRIL/Fab/Nb in complex with SQA1 and OXM1
| 超分子 | 名称: CCR6-BRIL/Fab/Nb in complex with SQA1 and OXM1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 詳細: CCR6-BRIL/Fab/Nb complex co-purified with SQA1 and OXM1. |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 120 kDa/nm |
-分子 #1: CCR6, Soluble cytochrome b562
| 分子 | 名称: CCR6, Soluble cytochrome b562 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 54.62302 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKTIIALSYI FCLVFADYKD DDDAKLQTMH HHHHHHHHHE NLYFQGGTSV DSEMLLCSLQ EVRQFSRAFV PIAYSLICVF GLLGNILVV ITFAFYKKAR SMTDVYLANM AIADILFALT LPFWAVSHAT GAWVFSNATC KLLKGIYAIN FNCGMWLLTC I AMDRYIAI ...文字列: MKTIIALSYI FCLVFADYKD DDDAKLQTMH HHHHHHHHHE NLYFQGGTSV DSEMLLCSLQ EVRQFSRAFV PIAYSLICVF GLLGNILVV ITFAFYKKAR SMTDVYLANM AIADILFALT LPFWAVSHAT GAWVFSNATC KLLKGIYAIN FNCGMWLLTC I AMDRYIAI VQATKSFRLR SATLPRSKII CLVVWGLSVI ISSSTFVFNQ KYNTQGSDVC EPKYQTVSEP IRWKLLMLGL EL LFGFFIP LMFMIFCYTA IVKTLRRQLA DLEDNWETLN DNLKVIEKAD NAAQVKDALT KMRAAALDAQ KATPPKLEDK SPD SPEMKD FRHGFDILVG QIDDALKLAN EGKVKEAQAA AEQLKTTRNA YIQKYLERAR STLQKEVKAI RVIIAVVLVF LACQ IPHNM VLLVTAANLG KMNRSCQSEK LIAYTKTVTE VLAFLHCCLN PVLYAFIGQK FRNYFLKILK DLWCVRRKYK SSGFS |
-分子 #2: anti-BRIL Fab Heavy chain
| 分子 | 名称: anti-BRIL Fab Heavy chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 28.832098 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGWSCIILFL VATATGVHSE ISEVQLVESG GGLVQPGGSL RLSCAASGFN VVDFSLHWVR QAPGKGLEWV AYISSSSGST SYADSVKGR FTISADTSKN TAYLQMNSLR AEDTAVYYCA RWGYWPGEPW WKAFDYWGQG TLVTVSSAST KGPSVFPLAP S SKSTSGGT ...文字列: MGWSCIILFL VATATGVHSE ISEVQLVESG GGLVQPGGSL RLSCAASGFN VVDFSLHWVR QAPGKGLEWV AYISSSSGST SYADSVKGR FTISADTSKN TAYLQMNSLR AEDTAVYYCA RWGYWPGEPW WKAFDYWGQG TLVTVSSAST KGPSVFPLAP S SKSTSGGT AALGCLVKDY FPEPVTVSWN SGALTSGVHT FPAVLQSSGL YSLSSVVTVP SSSLGTQTYI CNVNHKPSNT KV DKKVEPK SGGSENLYFQ GSHHHHHHHH HH |
-分子 #3: anti-BRIL Fab Nanobody
| 分子 | 名称: anti-BRIL Fab Nanobody / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 15.755214 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGHHHHHHHH HHENLYFQGS QVQLQESGGG LVQPGGSLRL SCAASGRTIS RYAMSWFRQA PGKEREFVAV ARRSGDGAFY ADSVQGRFT VSRDDAKNTV YLQMNSLKPE DTAVYYCAID SDTFYSGSYD YWGQGTQVTV SS |
-分子 #4: anti-BRIL Fab Light chain
| 分子 | 名称: anti-BRIL Fab Light chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.343348 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGWSCIILFL VATATGVHSS DIQMTQSPSS LSASVGDRVT ITCRASQSVS SAVAWYQQKP GKAPKLLIYS ASSLYSGVPS RFSGSRSGT DFTLTISSLQ PEDFATYYCQ QYLYYSLVTF GQGTKVEIKR TVAAPSVFIF PPSDSQLKSG TASVVCLLNN F YPREAKVQ ...文字列: MGWSCIILFL VATATGVHSS DIQMTQSPSS LSASVGDRVT ITCRASQSVS SAVAWYQQKP GKAPKLLIYS ASSLYSGVPS RFSGSRSGT DFTLTISSLQ PEDFATYYCQ QYLYYSLVTF GQGTKVEIKR TVAAPSVFIF PPSDSQLKSG TASVVCLLNN F YPREAKVQ WKVDNALQSG NSQESVTEQD SKDSTYSLSS TLTLSKADYE KHKVYACEVT HQGLSSPVTK SFNRG |
-分子 #5: CHOLESTEROL
| 分子 | 名称: CHOLESTEROL / タイプ: ligand / ID: 5 / コピー数: 1 / 式: CLR |
|---|---|
| 分子量 | 理論値: 386.654 Da |
| Chemical component information |  ChemComp-CLR: |
-分子 #6: 4-[[3,4-bis(oxidanylidene)-2-[[(1~{R})-1-(4-propan-2-ylfuran-2-yl...
| 分子 | 名称: 4-[[3,4-bis(oxidanylidene)-2-[[(1~{R})-1-(4-propan-2-ylfuran-2-yl)propyl]amino]cyclobuten-1-yl]amino]-~{N},~{N}-dimethyl-3-oxidanyl-pyridine-2-carboxamide タイプ: ligand / ID: 6 / コピー数: 1 / 式: EBX |
|---|---|
| 分子量 | 理論値: 426.466 Da |
| Chemical component information | 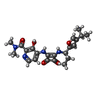 ChemComp-EBX: |
-分子 #7: 1-(4-chlorophenyl)-N-{[(2R)-4-(2,3-dihydro-1H-inden-2-yl)-5-oxomo...
| 分子 | 名称: 1-(4-chlorophenyl)-N-{[(2R)-4-(2,3-dihydro-1H-inden-2-yl)-5-oxomorpholin-2-yl]methyl}cyclopropane-1-carboxamide タイプ: ligand / ID: 7 / コピー数: 1 / 式: A1A2A |
|---|---|
| 分子量 | 理論値: 424.92 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 50 mM HEPES pH 7.5, 150 mM NaCl, 0.003% LMNG, 0.0003% CHS, 50 uM OXM1 and 50 uM of SQA1 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | CCR6-BRIL/Fab/Nb complex co-purified with SQA1 and OXM1. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 215000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-9d3g: |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)