+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4626 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | ClpB (DWB and K476C mutant) bound to casein in presence of ATPgammaS - state KC-2A | ||||||||||||
 マップデータ マップデータ | ClpB (DWB and K476C mutant) bound to a casein substrate in presence of ATPgammaS - State KC2A | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | disaggregase / proteostasis / AAA / CHAPERONE | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cellular response to heat / response to heat / protein refolding / ATP hydrolysis activity / ATP binding / identical protein binding / membrane / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||||||||
| 生物種 |   | ||||||||||||
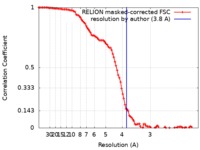
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | ||||||||||||
 データ登録者 データ登録者 | Deville C / Saibil HR | ||||||||||||
| 資金援助 |  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2019 ジャーナル: Cell Rep / 年: 2019タイトル: Two-Step Activation Mechanism of the ClpB Disaggregase for Sequential Substrate Threading by the Main ATPase Motor. 著者: Célia Deville / Kamila Franke / Axel Mogk / Bernd Bukau / Helen R Saibil /   要旨: AAA+ proteins form asymmetric hexameric rings that hydrolyze ATP and thread substrate proteins through a central channel via mobile substrate-binding pore loops. Understanding how ATPase and ...AAA+ proteins form asymmetric hexameric rings that hydrolyze ATP and thread substrate proteins through a central channel via mobile substrate-binding pore loops. Understanding how ATPase and threading activities are regulated and intertwined is key to understanding the AAA+ protein mechanism. We studied the disaggregase ClpB, which contains tandem ATPase domains (AAA1, AAA2) and shifts between low and high ATPase and threading activities. Coiled-coil M-domains repress ClpB activity by encircling the AAA1 ring. Here, we determine the mechanism of ClpB activation by comparing ATPase mechanisms and cryo-EM structures of ClpB wild-type and a constitutively active ClpB M-domain mutant. We show that ClpB activation reduces ATPase cooperativity and induces a sequential mode of ATP hydrolysis in the AAA2 ring, the main ATPase motor. AAA1 and AAA2 rings do not work synchronously but in alternating cycles. This ensures high grip, enabling substrate threading via a processive, rope-climbing mechanism. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4626.map.gz emd_4626.map.gz | 85.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4626-v30.xml emd-4626-v30.xml emd-4626.xml emd-4626.xml | 25.6 KB 25.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4626_fsc.xml emd_4626_fsc.xml | 10.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4626.png emd_4626.png | 163.7 KB | ||
| Filedesc metadata |  emd-4626.cif.gz emd-4626.cif.gz | 7.4 KB | ||
| その他 |  emd_4626_additional.map.gz emd_4626_additional.map.gz emd_4626_half_map_1.map.gz emd_4626_half_map_1.map.gz emd_4626_half_map_2.map.gz emd_4626_half_map_2.map.gz | 57.7 MB 71.3 MB 29.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4626 http://ftp.pdbj.org/pub/emdb/structures/EMD-4626 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4626 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4626 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4626_validation.pdf.gz emd_4626_validation.pdf.gz | 468.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4626_full_validation.pdf.gz emd_4626_full_validation.pdf.gz | 467.5 KB | 表示 | |
| XML形式データ |  emd_4626_validation.xml.gz emd_4626_validation.xml.gz | 15.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4626 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4626 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4626 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4626 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6qs7MC  4621C  4622C  4623C  4624C  4625C  4627C  4940C  4941C  4942C  6qs4C  6qs6C  6qs8C  6rn2C  6rn3C  6rn4C C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4626.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4626.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ClpB (DWB and K476C mutant) bound to a casein substrate in presence of ATPgammaS - State KC2A | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
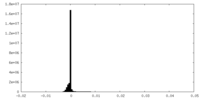
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: map filtered according to local resolution
| ファイル | emd_4626_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | map filtered according to local resolution | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
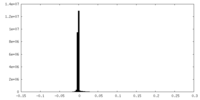
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_4626_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
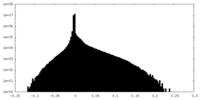
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_4626_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
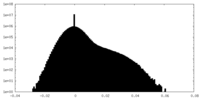
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : ClpB-DWB-K476C bound to casein in presence of ATPgammaS
| 全体 | 名称: ClpB-DWB-K476C bound to casein in presence of ATPgammaS |
|---|---|
| 要素 |
|
-超分子 #1: ClpB-DWB-K476C bound to casein in presence of ATPgammaS
| 超分子 | 名称: ClpB-DWB-K476C bound to casein in presence of ATPgammaS タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 分子量 | 理論値: 570 KDa |
-超分子 #2: ClpB-DWB-K476C
| 超分子 | 名称: ClpB-DWB-K476C / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: casein
| 超分子 | 名称: casein / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Chaperone protein ClpB
| 分子 | 名称: Chaperone protein ClpB / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 95.708133 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRLDRLTNKF QLALADAQSL ALGHDNQFIE PLHLMSALLN QEGGSVSPLL TSAGINAGQL RTDINQALNR LPQVEGTGGD VQPSQDLVR VLNLCDKLAQ KRGDNFISSE LFVLAALESR GTLADILKAA GATTANITQA IEQMRGGESV NDQGAEDQRQ A LKKYTIDL ...文字列: MRLDRLTNKF QLALADAQSL ALGHDNQFIE PLHLMSALLN QEGGSVSPLL TSAGINAGQL RTDINQALNR LPQVEGTGGD VQPSQDLVR VLNLCDKLAQ KRGDNFISSE LFVLAALESR GTLADILKAA GATTANITQA IEQMRGGESV NDQGAEDQRQ A LKKYTIDL TERAEQGKLD PVIGRDEEIR RTIQVLQRRT KNNPVLIGEP GVGKTAIVEG LAQRIINGEV PEGLKGRRVL AL DMGALVA GAKYRGEFEE RLKGVLNDLA KQEGNVILFI DALHTMVGAG KADGAMDAGN MLKPALARGE LHCVGATTLD EYR QYIEKD AALERRFQKV FVAEPSVEDT IAILRGLKER YELHHHVQIT DPAIVAAATL SHRYIADRQL PDKAIDLIDE AASS IRMQI DSKPEELDRL DRRIIQLKLE QQALMKESDE ASKKRLDMLN EELSDKERQY SELEEEWKAE KASLSGTQTI CAELE QAKI AIEQARRVGD LARMSELQYG KIPELEKQLE AATQLEGKTM RLLRNKVTDA EIAEVLARWT GIPVSRMMES EREKLL RME QELHHRVIGQ NEAVDAVSNA IRRSRAGLAD PNRPIGSFLF LGPTGVGKTE LCKALANFMF DSDEAMVRID MSEFMEK HS VSRLVGAPPG YVGYEEGGYL TEAVRRRPYS VILLDAVEKA HPDVFNILLQ VLDDGRLTDG QGRTVDFRNT VVIMTSNL G SDLIQERFGE LDYAHMKELV LGVVSHNFRP EFINRIDEVV VFHPLGEQHI ASIAQIQLKR LYKRLEERGY EIHISDEAL KLLSENGYDP VYGARPLKRA IQQQIENPLA QQILSGELVP GKVIRLEVNE DRIVAVQH UniProtKB: Chaperone protein ClpB |
-分子 #2: casein
| 分子 | 名称: casein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 2.060531 KDa |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) |
-分子 #3: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 3 / コピー数: 10 / 式: AGS |
|---|---|
| 分子量 | 理論値: 523.247 Da |
| Chemical component information |  ChemComp-AGS: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 8 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #5: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 1 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.6 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
| グリッド | モデル: Quantifoil, UltrAuFoil / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 6060 / 平均電子線量: 1.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)