+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human claudin-4 complex with Clostridium perfringens enterotoxin, sFab COP-1, and Nanobody | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | claudin / Fab / enterotoxin / nanobody / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報paracellular transport / positive regulation of metallopeptidase activity / calcium-independent cell-cell adhesion via plasma membrane cell-adhesion molecules / Tight junction interactions / bicellular tight junction assembly / apicolateral plasma membrane / regulation of cell morphogenesis / tight junction / chloride channel activity / positive regulation of wound healing ...paracellular transport / positive regulation of metallopeptidase activity / calcium-independent cell-cell adhesion via plasma membrane cell-adhesion molecules / Tight junction interactions / bicellular tight junction assembly / apicolateral plasma membrane / regulation of cell morphogenesis / tight junction / chloride channel activity / positive regulation of wound healing / renal absorption / chloride channel complex / bicellular tight junction / lateral plasma membrane / establishment of skin barrier / basal plasma membrane / response to progesterone / female pregnancy / circadian rhythm / transmembrane signaling receptor activity / cell-cell junction / toxin activity / cell adhesion / positive regulation of cell migration / apical plasma membrane / structural molecule activity / extracellular region / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 | synthetic construct (人工物) /  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
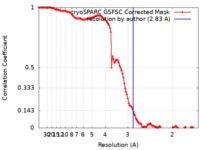
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.83 Å | |||||||||
 データ登録者 データ登録者 | Vecchio AJ | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-EM Structures of Clostridium perfringens Enterotoxin Bound to its Human Receptor, Claudin-4 著者: Vecchio AJ / Kossiakoff AA / Erramilli SK / Rathnayake SS | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_45749.map.gz emd_45749.map.gz | 88.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-45749-v30.xml emd-45749-v30.xml emd-45749.xml emd-45749.xml | 23.4 KB 23.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_45749_fsc.xml emd_45749_fsc.xml | 11.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_45749.png emd_45749.png | 81.6 KB | ||
| Filedesc metadata |  emd-45749.cif.gz emd-45749.cif.gz | 7.3 KB | ||
| その他 |  emd_45749_half_map_1.map.gz emd_45749_half_map_1.map.gz emd_45749_half_map_2.map.gz emd_45749_half_map_2.map.gz | 165 MB 165 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-45749 http://ftp.pdbj.org/pub/emdb/structures/EMD-45749 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45749 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45749 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_45749_validation.pdf.gz emd_45749_validation.pdf.gz | 767.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_45749_full_validation.pdf.gz emd_45749_full_validation.pdf.gz | 767 KB | 表示 | |
| XML形式データ |  emd_45749_validation.xml.gz emd_45749_validation.xml.gz | 20.6 KB | 表示 | |
| CIF形式データ |  emd_45749_validation.cif.gz emd_45749_validation.cif.gz | 26.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45749 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45749 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45749 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45749 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9cmiMC  9cmhC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_45749.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_45749.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.827 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_45749_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_45749_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human claudin-4 complex with Clostridium perfringens enterotoxin,...
| 全体 | 名称: Human claudin-4 complex with Clostridium perfringens enterotoxin, sFab COP-1, and Nanobody |
|---|---|
| 要素 |
|
-超分子 #1: Human claudin-4 complex with Clostridium perfringens enterotoxin,...
| 超分子 | 名称: Human claudin-4 complex with Clostridium perfringens enterotoxin, sFab COP-1, and Nanobody タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 / 詳細: 5-protein complex |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 120 KDa |
-分子 #1: Claudin-4
| 分子 | 名称: Claudin-4 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 22.234332 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: GSMASMGLQV MGIALAVLGW LAVMLCCALP MWRVTAFIGS NIVTSQTIWE GLWMNCVVQS TGQMQCKVYD SLLALPQDLQ AARALVIIS IIVAALGVLL SVVGGKCTNC LEDESAKAKT MIVAGVVFLL AGLMVIVPVS WTAHNIIQDF YNPLVASGQK R EMGASLYV ...文字列: GSMASMGLQV MGIALAVLGW LAVMLCCALP MWRVTAFIGS NIVTSQTIWE GLWMNCVVQS TGQMQCKVYD SLLALPQDLQ AARALVIIS IIVAALGVLL SVVGGKCTNC LEDESAKAKT MIVAGVVFLL AGLMVIVPVS WTAHNIIQDF YNPLVASGQK R EMGASLYV GWAASGLLLL GGGLLCCNCP PRTDKPYSAK YSAARSAAAS NYV UniProtKB: Claudin-4 |
-分子 #2: Heat-labile enterotoxin B chain
| 分子 | 名称: Heat-labile enterotoxin B chain / タイプ: protein_or_peptide / ID: 2 / 詳細: Trypsin treated CpE / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 33.000582 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: TPINITNSNS NLSDGLYVID KGDGWILGEP SVVSSQILNP NETGTFSQSL TKSKEVSINV NFSVGFTSEF IQASVEYGFG ITIGEQNTI ERSVSTTAGP NEYVYYKVYA TYRKYQAIRI SHGNISDDGS IYKLTGIWLS KTSADSLGNI DQGSLIETGE R CVLTVPST ...文字列: TPINITNSNS NLSDGLYVID KGDGWILGEP SVVSSQILNP NETGTFSQSL TKSKEVSINV NFSVGFTSEF IQASVEYGFG ITIGEQNTI ERSVSTTAGP NEYVYYKVYA TYRKYQAIRI SHGNISDDGS IYKLTGIWLS KTSADSLGNI DQGSLIETGE R CVLTVPST DIEKEILDLA AATERLNLTD ALNSNPAGNL YDWRSSNSYP WTQKLNLHLT ITATGQKYRI LASKIVDFNI YS NNFNNLV KLEQSLGDGV KDHYVDISLD AGQYVLVMKA NSSYSGNYPY SILFQKFGLV PR UniProtKB: Heat-labile enterotoxin B chain |
-分子 #3: COP-1 sFab Heavy Chain
| 分子 | 名称: COP-1 sFab Heavy Chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 28.064498 KDa |
| 配列 | 文字列: MKKNIAFLLA SMFVFSIATN AYAEISEVQL VESGGGLVQP GGSLRLSCAA SGFNFSSSYI HWVRQAPGKG LEWVASISSS SGSTSYADS VKGRFTISAD TSKNTAYLQM NSLRAEDTAV YYCARWFHPW WWWEYLFRGA IDYWGQGTLV TVSSASTKGP S VFPLAPSS ...文字列: MKKNIAFLLA SMFVFSIATN AYAEISEVQL VESGGGLVQP GGSLRLSCAA SGFNFSSSYI HWVRQAPGKG LEWVASISSS SGSTSYADS VKGRFTISAD TSKNTAYLQM NSLRAEDTAV YYCARWFHPW WWWEYLFRGA IDYWGQGTLV TVSSASTKGP S VFPLAPSS KSTSGGTAAL GCLVKDYFPE PVTVSWNSGA LTSGVHTFPA VLQSSGLYSL SSVVTVPSSS LGTQTYICNV NH KPSNTKV DKKVEPKSCD KTHT |
-分子 #4: Anti-Fab Nanobody
| 分子 | 名称: Anti-Fab Nanobody / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 13.175438 KDa |
| 配列 | 文字列: GSVQLQESGG GLVQPGGSLR LSCAASGRTI SRYAMSWFRQ APGKEREFVA VARRSGDGAF YADSVQGRFT VSRDDAKNTV YLQMNSLKP EDTAVYYCAI DSDTFYSGSY DYWGQGTQVT VS |
-分子 #5: COP-1 sFab Light Chain
| 分子 | 名称: COP-1 sFab Light Chain / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.794859 KDa |
| 配列 | 文字列: MKKNIAFLLA SMFVFSIATN AYASDIQMTQ SPSSLSASVG DRVTITCRAS QSVSSAVAWY QQKPGKAPKL LIYSASSLYS GVPSRFSGS RSGTDFTLTI SSLQPEDFAT YYCQQSSSSL ITFGQGTKVE IKRTVAAPSV FIFPPSDSQL KSGTASVVCL L NNFYPREA ...文字列: MKKNIAFLLA SMFVFSIATN AYASDIQMTQ SPSSLSASVG DRVTITCRAS QSVSSAVAWY QQKPGKAPKL LIYSASSLYS GVPSRFSGS RSGTDFTLTI SSLQPEDFAT YYCQQSSSSL ITFGQGTKVE IKRTVAAPSV FIFPPSDSQL KSGTASVVCL L NNFYPREA KVQWKVDNAL QSGNSQESVT EQDSKDSTYS LSSTLTLSKA DYEKHKVYAC EVTHQGLSSP VTKSFNRGEC |
-分子 #6: Lauryl Maltose Neopentyl Glycol
| 分子 | 名称: Lauryl Maltose Neopentyl Glycol / タイプ: ligand / ID: 6 / コピー数: 1 / 式: AV0 |
|---|---|
| 分子量 | 理論値: 1.005188 KDa |
| Chemical component information |  ChemComp-AV0: |
-分子 #7: water
| 分子 | 名称: water / タイプ: ligand / ID: 7 / コピー数: 2 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5.6 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: 20 mM Hepes pH 7.4, 100 mM NaCl, and 0.003% LMNG | ||||||||||||
| グリッド | モデル: UltrAuFoil / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. 詳細: glow-discharged for 30 seconds at 20 W using a Solarus 950 (Gatan) plasma cleaner | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum |
| ソフトウェア | 名称: EPU |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 6774 / 平均電子線量: 70.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.1 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| ソフトウェア | 名称:  UCSF Chimera UCSF Chimera | ||||||
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT | ||||||
| 得られたモデル |  PDB-9cmi: |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)